
Descrevemos aqui uma classe de métodos de aprendizagem que foi desenvolvida separadamente em diferentes campos: estatística e inteligência artificial, com base em modelos essencialmente idênticos. A idéia central é extrair combinações lineares das entradas como recursos derivados e modelar o alvo como uma função não linear desses recursos. O resultado é um poderoso método de aprendizado, com aplicações difundidas em muitos campos. Primeiro discutimos o modelo de prospeção de projeção, que evoluiu no domínio de estatísticas semiparamétricas e suavização. Depois nos dedicamos aos modelos de redes neurais.
Como em nosso problema genérico de aprendizado supervisionado, suponha que temos um vetor de entrada \(X\) com \(p\) componentes e um \(Y\) de destino. Sejam \(\omega_m\), \(m = 1, 2,\cdots,M\) vetores de dimensão \(p\) unitários de parâmetros desconhecidos. O modelo de regressão por prospeção de projeção (PPR) tem a forma \begin{equation*} f(X) \, = \, \sum_{m=1}^M g_m(\omega_m^\top X)\cdot \end{equation*}
Este é um modelo aditivo, mas nos recursos derivados \(V_m = \omega_ω^\top X\) em vez das próprias entradas. As funções \(g_m\) não são especificadas e são estimadas junto com as direções \(\omega_m\) usando algum método de suavização flexível, veja abaixo.
 |
 |
 |  |
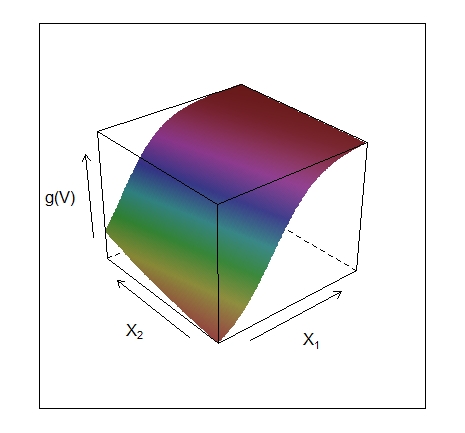
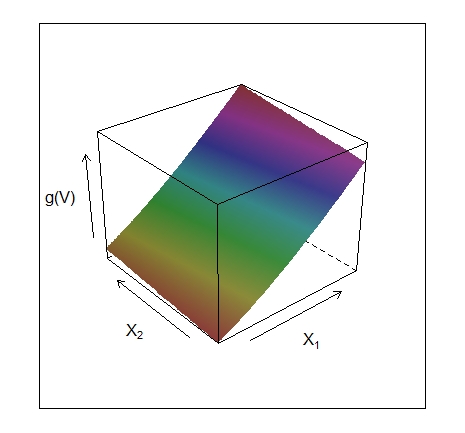
A função \(g_m(\omega_m^\top X)\) é chamada de função de crista em \(\mathbb{R}^p\). Ela varia apenas na direção definida pelo vetor \(\omega_m\). A variável escalar \(V_m = \omega_ω^\top X\) é a projeção de \(X\) no vetor unitário \(\omega_m\), e buscamos \(\omega_m\) para que o modelo se ajuste bem, daí o nome busca de projeção. A Figura V.1 mostra alguns exemplos de funções de crista. No exemplo à esquerda \(\omega = (1/\sqrt{2}) (1,1)^\top\), de forma que a função só varia na direção \(X_1+X_2\). No exemplo à direita, \(\omega = (1,0)\).
O modelo PPR é muito geral, uma vez que a operação de formação de funções não lineares de combinações lineares gera uma classe surpreendentemente grande de modelos. Por exemplo, o produto \(X_1\times X_2\) pode ser escrito como \begin{equation*} \dfrac{1}{4}\big( (X_1+X_2)^2-(X_1-X_2)^2\big) \end{equation*} e produtos de ordem superior podem ser representados de forma semelhante.
Na verdade, se \(M\) for considerado arbitrariamente grande, para a escolha apropriada de \(g_m\), o modelo PPR pode se aproximar arbitrariamente de qualquer função contínua em \(\mathbb{R}^p\). Essa classe de modelos é chamada de aproximador universal. No entanto, essa generalidade tem um preço. A interpretação do modelo ajustado geralmente é difícil, porque cada entrada entra no modelo de uma forma complexa e multifacetada. Como resultado, o modelo PPR &ecute; mais útil para previsão e não muito útil para produzir um modelo compreensível para os dados. O modelo \(M = 1\), conhecido como modelo de índice único em econometria, é uma exceção. É um pouco mais geral do que o modelo de regressão linear e oferece uma interpretação semelhante.
Como ajustamos um modelo PPR, dados os dados de treinamento \((x_i, y_i)\), \(i = 1, 2,\cdots, N\)? Procuramos os minimizadores aproximados da função de erro \begin{equation} \sum_{i=1}^N \left( y_i-\sum_{m=1}^M g_m\big(\omega_m^\top x_i\big)\right)^2 \end{equation} sobre as funções \(g_m\) e vetores de direção \(\omega_m\), \(m = 1,2,\cdots, M\).
Como em outros problemas de suavização, precisamos explicitamente ou implicitamente impor restrições de complexidade ao \(g_m\), para evitar soluções de ajuste excessivo.
Considere apenas um termo, \(M = 1\) e elimine o subscrito. Dado o vetor de direção \(\omega\), formamos as variáveis derivadas \(v_i = \omega^\top x_i\). Então, temos um problema de suavização unidimensional e podemos aplicar qualquer gráfico de dispersão mais suave, como um spline de suavização, para obter uma estimativa de \(g\).
Por outro lado, dado \(g\), queremos minimizar a função de erro em \(\omega\). Uma busca de Gauss-Newton é conveniente para essa tarefa. Este é um método quase Newton, no qual a parte do Hessiano envolvendo a segunda derivada de \(g\) é descartada. Ele pode ser simplesmente derivado da seguinte forma. Seja \(\omega_{old}\) a estimativa atual para \(\omega\). Nós escrevemos \begin{equation*} g(\omega^\top x_i) \approx g(\omega_{old}^\top x_i)+g'(\omega_{old}^\top x_i)(\omega-\omega_{old})^\top x_i \end{equation*} para obtermos \begin{equation*} \sum_{i=1}^N \big(y_i-g(\omega^\top x_i)\big)^2 \approx \sum_{i=1}^N g'(\omega_{old}^\top x_i)^2 \left( \omega_{old}^\top x_i +\dfrac{y_i-g(\omega_{old}^\top x_i)}{g'(\omega_{old}^\top x_i)} - \omega^\top x_i\right)^2\cdot \end{equation*}
Para minimizar o lado direito, realizamos uma regressão de mínimos quadrados com alvo \begin{equation*} \omega_{old}^\top x_i +\big(y_i-g(\omega_{old}^\top x_i)\big)/{g'(\omega_{old}^\top x_i)} \end{equation*} na entrada \(x_i\), com pesos \(g'(\omega_{old}^\top x_i)^2\) e sem termo de intercepto ou viés. Isso produz o vetor de coeficientes atualizado \(\omega_{new}\).
Essas duas etapas, estimativas de \(g\) e \(\omega\), são iteradas até a convergência. Com mais de um termo no modelo PPR, o modelo é construído de uma maneira avançada de estágio, adicionando um par \((\omega_m, g_m)\) em cada estágio.
Existem vários detalhes de implementação.
Existem muitas outras aplicações, como estimação de densidades (Friedman et al., 1984; Friedman, 1987), onde a ideia de busca de projeção pode ser usada. No entanto, o modelo de regressão de busca por projeção não tem sido amplamente utilizado no campo da estatística, talvez porque, na época de sua introdução (1981), suas demandas computacionais excediam as capacidades da maioria dos computadores disponíveis. Mas representa um importante avanço intelectual, que floresceu em sua reencarnação no campo das redes neurais, o tópico do restante deste capítulo.
Rede neural ou rede neural artificial tem a capacidade de aprender por meio de exemplos, é um modelo de processamento de informações inspirado no sistema biológico de neurônios. É composto por um grande número de elementos de processamento altamente interconectados, conhecidos como neurônios, para resolver problemas. Ele segue o caminho não linear e processa as informações em paralelo em todos os nós. Uma rede neural é um sistema adaptativo complexo. Adaptável significa que ele tem a capacidade de alterar sua estrutura interna ajustando os pesos das entradas.
O termo rede neural evoluiu para abranger uma grande classe de modelos e métodos de aprendizagem. Aqui descrevemos a rede neural mais usada, às vezes chamada de rede de propagação reversa de camada oculta. Como deixaremos claro são apenas modelos estatísticos não-lineares, muito parecido com o modelo de prospeção de projeção discutido antes.
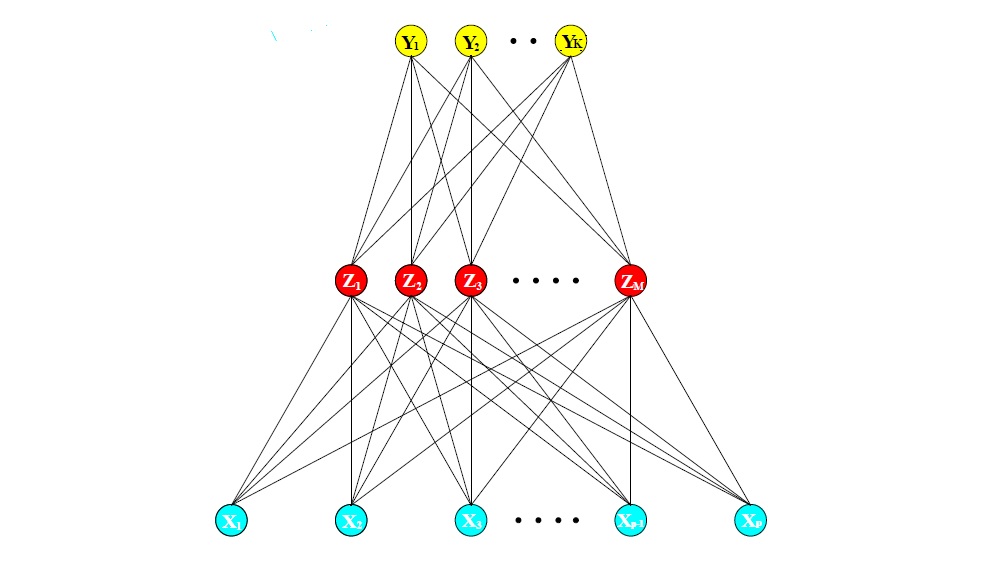
Uma rede neural é um modelo de regressão ou classificação de dois estágios, tipicamente representado por um diagrama de rede, como na Figura V.2. Essa rede se aplica tanto à regressão quanto à classificação. Para regressão, normalmente \(K = 1\) e há apenas uma unidade de saída \(Y_1\) no topo. No entanto, essas redes podem lidar com várias respostas quantitativas de maneira perfeita, por isso lidaremos com o caso geral.
A rede neural foi projetada para resolver problemas fáceis para os humanos e difíceis para as máquinas, como a identificação de fotos de cães e gatos, a identificação de fotos numeradas. Esses problemas são freqüentemente chamados de reconhecimento de padrões. Sua aplicação abrange desde o reconhecimento óptico de caracteres até a deteção de objetos.
Para a classificação \(K\)-classes, existem \(K\) unidades no topo, com a \(k\)-ésima unidade modelando a probabilidade da classe \(k\). Existem \(K\) medições de alvo \(Y_k\), \(k = 1,\cdots, K\), sendo cada uma codificada como uma variável 0 - 1 para a \(k\)-classe.
Os recursos derivados \(Z_m\) são criados a partir de combinações lineares das entradas e, em seguida, o alvo \(Y_k\) é modelado como uma função das combinações lineares do \(Z_m\), \begin{equation} \begin{array}{rclc} Z_m & = & \sigma(\alpha_{0m}+\alpha^\top_m X), & m=1,\cdots,M, \\ T_k & = & \beta_{0k} \, + \, \beta_k^\top Z, & k=1,\cdots,K, \\ f_k(X) & = & g_k(T), & k=1,\cdots,K, \end{array} \end{equation} onde \(Z=(Z_1,Z_2,\cdots,Z_M)\) e \(T=(T_1,T_2,\cdots,T_K)\).
 |
|---|
| Figura V.2. Esquema de uma única camada oculta, rede neural feed-forward. |
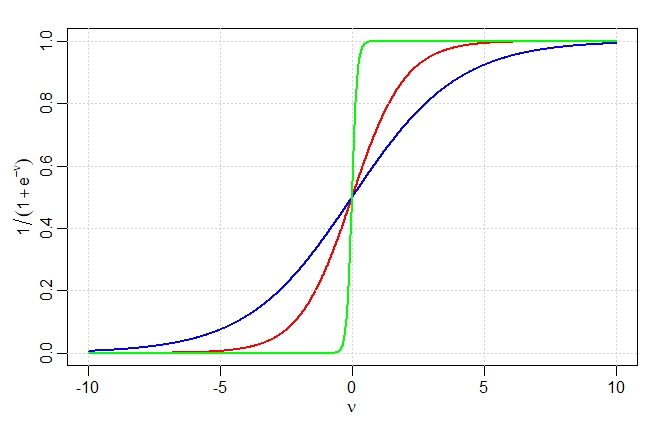
A função de ativação \(\sigma(\nu)\) é geralmente escolhida como sendo o sigmóide \(\sigma(\nu) = 1 / (1 + e^{-\nu})\); veja na Figura V.3 abaixo para uma plotagem desta curva. Às vezes, as funções de base radial gaussiana são usadas para o \(\sigma(\nu)\), produzindo o que é conhecido como uma rede de função de base radial.
Diagramas de rede neural, como a Figura V.2, às vezes são desenhados com uma unidade adicional de polarização alimentando cada unidade nas camadas oculta e de saída. Pensando na constante 1 como um recurso de entrada adicional, esta unidade de polarização captura os interceptos \(\alpha_{0m}\) e \(\beta_{0k}\) no modelo acima.
A função de saída \(g_k(T)\) permite uma transformação final do vetor de saídas \(T\). Para a regressão, normalmente escolhemos a função de identidade \(g_k(T) = T_k\). Os primeiros trabalhos na classificação da classe \(K\) também usaram a função identidade, mas isso foi posteriormente abandonado em favor da função softmax \begin{equation*} g_k(T) \, = \, \dfrac{e^{T_k}}{\sum_{\ell=1}^K e^{T_\ell}}\cdot \end{equation*}
É claro que essa é exatamente a transformação usada no modelo multilogit e produz estimativas positivas que somam um. Outras funções de ativação linear podem ser utilizadas.
As unidades no meio da rede, calculando os recursos derivados \(Z_m\), são chamadas de unidades ocultas porque os valores \(Z_m\) não são observados diretamente. Em geral, pode haver mais de uma camada oculta. Podemos pensar nos \(Z_m\) como uma expansão básica das entradas originais \(X\); a rede neural é então um modelo linear padrão, ou modelo multilogit linear, usando essas transformações como entradas. Há, entretanto, um importante aprimoramento sobre as técnicas de expansão da base, os parâmetros das funções básicas são aprendidos a partir dos dados.
 |
|---|
| Figura V.3. Gráfico da função sigmóide \(\sigma(\nu) = 1/(1 + \exp(−\nu))\) (curva vermelha), comumente usada na camada oculta de uma rede neural. Estão incluídos \(\sigma(s\nu)\) para \(s = 1/2\) (curva azul) e \(s = 10\) (curva verde). O parâmetro de escala \(s\) controla a taxa de ativação, e podemos ver que \(s\) grande equivale a uma ativação forte em \(\nu = 0\). Observe que \(\sigma \big(s (\nu - \nu_0)\big)\) muda o limite de ativação de 0 para \(\nu_0\). |
Observe que se \(\sigma\) for a fun&cceil;ão identidade, o modelo inteiro se reduz a um modelo linear nas entradas. Portanto, uma rede neural pode ser considerada uma generalização não linear do modelo linear, tanto para regressão quanto para classificação. Ao introduzir a transformação não linear \(\sigma\), ela amplia muito a classe de modelos lineares. Na Figura V.3 vemos que a taxa de ativação do sigmóide depende da norma de \(\alpha_m\), e se \(||\alpha_m||\) for muito pequena, a unidade estará de fato operando na parte linear de sua função de ativação.
Observe também que o modelo de rede neural com uma camada oculta tem exatamente a mesma forma que o modelo de busca de projeção descrito acima. A diferença é que o modelo PPR usa funções não paramétricas \(g_m(\nu)\), enquanto a rede neural usa uma função muito mais simples baseada em \(\sigma(\nu)\), com três parâmetros livres em seu argumento. Em detalhes, vendo o modelo de rede neural como um modelo PPR, identificamos \begin{equation*} \begin{array}{rcl} g_m(\omega_m^\top X) & = & \beta_m \sigma\big( \alpha_{0m}+\alpha_m^\top X\big)\\[0.4em] & = & \beta_m \sigma\big( \alpha_{0m}+||\alpha_m||(\omega_m^\top X)\big), \end{array} \end{equation*} onde \(\omega_m = \alpha_m/||\alpha_m||\) é o \(m\)-ésimo vetor unitário.
Uma vez que \begin{equation*} \sigma_{\beta,\alpha_0,s}(\nu) = \beta \sigma(\alpha_0 + s\nu) \end{equation*} tem menor complexidade do que um \(g\) não paramétrico mais geral \(g(\nu)\), não é surpreendente que uma rede neural possa usar 20 ou 100 dessas funções, enquanto o modelo PPR normalmente usa menos termos, \(M = 5\) ou 10, por exemplo.
Finalmente, notamos que o nome redes neurais deriva do fato de que foram inicialmente desenvolvidas como modelos para o cérebro humano. Cada unidade representa um neurónio e as conexões, links na Figura V.2, representam sinapses. Nos primeiros modelos, os neurónios disparavam quando o sinal total passado para aquela unidade excedia um certo limite. No modelo acima, isso corresponde ao uso de uma função degrau para \(\sigma(Z)\) e \(g_m(T)\). Mais tarde, a rede neural foi reconhecida como uma ferramenta útil para modelagem estatística não linear e, para esse propósito, a função degrau não é suave o suficiente para otimização. Conseqüentemente, a função degrau foi substituída por uma função de limite mais suave, a sigmóide na Figura V.3.
O modelo de rede neural tem parâmetros desconhecidos, geralmente chamados de pesos e buscamos valores para eles que fazem o modelo se encaixar bem nos dados de treinamento. Denotamos o conjunto completo de pesos por \(\theta\), que consiste em \begin{array}{rcl} \{\alpha_{0m}, \alpha_m \, ; \, m=1,2,\cdots,M\} & M(p+1) & \mbox{pesos} \\ \{\beta_{0k},\beta_k \, : \, k=1,2,\cdots,M\} & K(M+1) & \mbox{pesos} \end{array}
Para a regressão, usamos a soma dos quadrados dos erros como nossa medida de ajuste, considerada a função de erro: \begin{equation} R(\theta) \, = \, \sum_{k=1}^K\sum_{i=1}^N \big(y_{ik}-f_k(x_i) \big)^2\cdot \end{equation}
Para classificação; usamos o erro quadrático, entropia cruzada ou desvio: \begin{equation} R(\theta) \, = \, -\sum_{k=1}^K\sum_{i=1}^N y_{ik}\log\big(f_k(x_i) \big), \end{equation} e o classificador correspondente é \(G(x) = \mbox{argmax}_k f_k(x)\). Com a função de ativação softmax e a função de erro de entropia cruzada, o modelo de rede neural é exatamente um modelo de regressão logística linear nas unidades ocultas e todos os parâmetros são estimados por máxima verossimilhança.
Geralmente, não queremos o minimizador global de \(R(\theta)\), pois é provável que seja uma solução de ajuste excessivo. Em vez disso, é necessária alguma regularização: isso é obtido diretamente por meio de uma pena ou indiretamente por meio de interrupção antecipada. Os detalhes são fornecidos na próxima seção.
A abordagem genérica para minimizar \(R(\theta)\) é por gradiente descendente, chamado de retropropagação neste cenário. Por causa da forma composicional do modelo, o gradiente pode ser facilmente derivado usando a regra da cadeia para diferenciação. Isso pode ser calculado por uma varredura para frente e para trás na rede, mantendo o controle apenas das quantidades locais de cada unidade.
Aqui está a retropropagação em detalhes para perda de erro quadrático. Seja \(z_{mi}=\sigma(\alpha_{0m}+\alpha_m^\top x_i)\), como definido antes e \(z_i=(z_{1i},z_{2i},\cdots,z_{Mi})\). Temos então \begin{equation} R(\theta) \, = \, \sum_{i=1}^N R_i \, = \, \sum_{i=1}^N \sum_{k=1}^K \big(y_{ik}-f_k(x_i) \big)^2, \end{equation} com derivadas \begin{array}{rcl} \dfrac{\partial R_i}{\partial\beta_{km}} & = & -2\big(y_{ik}-f_k(x_i) \big)g'_k\big(\beta_k^\top z_i\big)z_{mi},\\[0.6em] \dfrac{\partial R_i}{\partial\alpha_{m\ell}} & = & \displaystyle -\sum_{k=1}^K 2\big(y_{ik}-f_k(x_i) \big)g'_k\big(\beta_k^\top z_i\big)\beta_{km}\sigma'\big(\alpha_m^\top x_i\big)x_{i\ell}\cdot \end{array}
Dadas essas derivadas, uma atualização de gradiente descendente na iteração \((r+1)\)-ésima tem a forma \begin{array}{rcl} \beta_{km}^{(r+1)} & = & \displaystyle \beta_{km}^{(r)}-\gamma_r\sum_{i=1}^N \dfrac{\partial R_i}{\partial\beta_{km}^{(r)}},\\[0.6em] \alpha_{m\ell}^{(r+1)} & = & \displaystyle \alpha_{m\ell}^{(r)}-\gamma_r\sum_{i=1}^N \dfrac{\partial R_i}{\partial\alpha_{m\ell}^{(r)}}, \end{array} onde \(\gamma_r\) é a taxa de aprendizado, discutida abaixo.
Agora escrevamos as derivadas como \begin{array}{rcl} \dfrac{\partial R_i}{\partial\beta_{km}} & = & \delta_{ki} z_{mi},\\ \dfrac{\partial R_i}{\partial\alpha_{m\ell}} & = & s_{mi}x_{i\ell}\cdot \end{array}
As quantidades \(\delta_{ki}\) e \(s_{mi}\) são erros do modelo atual nas unidades de saída e da camada oculta, respectivamente. A partir de suas definições, esses erros satisfazem \begin{equation} s_{mi} \, = \, \sigma'\big(\alpha_m^\top x_i\big)\sum_{k=1}^K \beta_{km}\delta_{ki}, \end{equation} conhecidas como equações de propagação reversa.
Usando isso, as atualizações podem ser implementadas com um algoritmo de duas passagens. No passo para frente, os pesos atuais são fixos e os valores previstos \(\widehat{f}_k(x_i)\) são calculados a partir das funções das combinações lineares apresentadas no começo da Seção V.2. Na passagem para trás, os erros \(\delta_{ki}\) são calculados e, em seguida, propagados de volta por meio de \(\sigma'\big(\alpha_m^\top x_i\big)\sum_{k=1}^K \beta_{km}\delta_{ki}\) para fornecer os erros \(s_{mi}\). Ambos os conjuntos de erros são então usados para calcular os gradientes para as atualizações de \(\beta_{km}^{(r+1)}\) e \(\alpha_{m\ell}^{(r+1)}\), via \(\partial R_i/\partial\beta_{km}\) e \(\partial R_i/\partial\alpha_{m\ell}\).
Esse procedimento de duas passagens é conhecido como propagação reversa. Também foi chamada de regra delta (Widrow and Hoff, 1960). Os componentes computacionais para entropia cruzada têm a mesma forma que aqueles para a função de erro da soma dos quadrados e são derivados num exercício.
As vantagens da propagação reversa são sua natureza simples e local. No algoritmo de propagação de retorno, cada unidade oculta passa e recebe informações apenas de e para unidades que compartilham uma conexão. Portanto, ele pode ser implementado de forma eficiente em um computador de arquitetura paralela.
As atualizações em \(\beta_{km}^{(r+1)}\) e \(\alpha_{m\ell}^{(r+1)}\) são um tipo de aprendizagem em lote, com as atualizações de parâmetros sendo uma soma de todos os casos de treinamento. O aprendizado também pode ser realizado online - processando cada observação, uma de cada vez, atualizando o gradiente após cada caso de treinamento e percorrendo os casos de treinamento várias vezes. Nesse caso, as somas nas equações que definem as atualizações \(\beta_{km}^{(r+1)}\) e \(\alpha_{m\ell}^{(r+1)}\) são substituídas por uma única soma. Uma época de treinamento se refere a uma varredura em todo o conjunto de treinamento. O treinamento online permite que a rede lide com conjuntos de treinamento muito grandes e também atualize os pesos conforme novas observações surgem.
A taxa de aprendizado \(\gamma_r\) para aprendizado em lote é geralmente considerada uma constante e também pode ser otimizada por uma pesquisa de linha que minimiza a função de erro a cada atualização. Com o aprendizado online, \(\gamma_r\) deve diminuir para zero conforme a iteração \(r \to\infty\). Esse aprendizado é uma forma de aproximação estocástica (Robbins and Munro, 1951); os resultados neste campo garantem a convergência se \(\gamma_r\to 0\), \(\sum_r \gamma_r = \infty\) e \(\sum_r \gamma_r^2 <\infty\), satisfeito, por exemplo, por \(\gamma_r = 1/r\).
A propagação reversa pode ser muito lenta e, por esse motivo, geralmente não é o método escolhido. Técnicas de segunda ordem, como o método de Newton, não são atraentes aqui, porque a matriz derivada de \(R(\theta)\), o Hessiano, pode ser muito grande. Melhores abordagens para o ajuste incluem gradientes conjugados e métodos métricos variáveis. Isso evita o cálculo explícito da segunda matriz de derivadas, embora ainda forneça uma convergência mais rápida.
Ajustamos redes neurais e registramos o erro de teste médio \begin{equation} \mbox{E}_{\mbox{Teste}}\big(Y-\widehat{f}(X)\big)^2\cdot \end{equation} Escolhemos àquela com o menor erro de teste possível.
O treinamento de redes neurais é uma arte e tanto. O modelo geralmente é superparametrizado e o problema de otimização não é convexo e instável, a menos que certas diretrizes sejam seguidas. Nesta seção, resumimos algumas das questões importantes.
Observe que, se os pesos estão próximos de zero, a parte operativa do sigmóide é aproximadamente linear e, portanto, a rede neural colapsa em um modelo aproximadamente linear. Normalmente, os valores iniciais dos pesos são escolhidos para serem valores aleatórios próximos de zero. Portanto, o modelo começa quase linear e se torna não linear à medida que os pesos aumentam. Unidades individuais localizam em direções e introduzem não linearidades onde necessário. O uso de pesos zero exatos leva a derivadas zero e simetria perfeita e o algoritmo nunca se move. Em vez disso, começar com grandes pesos geralmente leva a soluções ruins
Freqüentemente, as redes neurais têm muitos pesos e superdimensionam os dados no mínimo global de \(R(\theta)\). Nos primeiros desenvolvimentos de redes neurais, seja por projeto ou por acidente, uma regra de parada antecipada foi usada para evitar o superajuste. Aqui, treinamos o modelo apenas por um tempo e paramos bem antes de nos aproximarmos do mínimo global. Como os pesos começam em uma soluçõo altamente regularizada linear, isso tem o efeito de reduzir o modelo final em direção a um modelo linear. Um conjunto de dados de validação é útil para determinar quando parar, uma vez que esperamos que o erro de validação comece a aumentar.
Um método mais explícito para regularização é o declínio do peso, que é análogo à regressão de crista usada para modelos lineares. Adicionamos uma penalidade à função de erro \(R(\theta)+\lambda J(\theta)\), onde \begin{equation} J(\theta) \, = \, \sum_{km}\beta^2_{km}+\sum_{m\ell} \alpha^2_{m\ell}, \end{equation} e \(\lambda\geq 0\) é um parâmetro de ajuste.
Valores maiores de \(\lambda\) tenderão a reduzir os pesos até zero: normalmente a validação cruzada é usada para estimar \(\lambda\). O efeito da penalidade é simplesmente adicionar os termos \(2\beta_{km}\) e \(2\alpha_{m\ell}\) às respectivas expressões de gradiente \(\beta_{km}^{(r+1)}\) e \(\alpha_{m\ell}^{(r+1)}\). Outras formas de penalidade foram propostas, por exemplo, \begin{equation} J(\theta) \, = \, \sum_{km} \dfrac{\beta_{km}^2}{1+\beta_{km}^2}+\sum_{m\ell} \dfrac{\alpha_{m\ell}^2}{1+\alpha_{m\ell}^2}, \end{equation} conhecido como penalidade de eliminação de peso. Isso tem o efeito de reduzir pesos menores mais do que \(J(\theta)\) anterior.
Como a escala das entradas determina a escala efetiva dos pesos na camada inferior, ela pode ter um grande efeito na qualidade da solução final. No início, é melhor padronizar todas as entradas para ter média zero e desvio padrão um. Isso garante que todas as entradas sejam tratadas igualmente no processo de regularização e permite escolher um intervalo significativo para os pesos iniciais aleatórios. Com entradas padronizadas, é típico obter pesos uniformes aleatórios no intervalo \((-0.7, +0.7)\).
De modo geral, é melhor ter muitas unidades ocultas do que poucas. Com muito poucas unidades ocultas, o modelo pode não ter flexibilidade suficiente para capturar as não linearidades nos dados; com muitas unidades ocultas, os pesos extras podem ser reduzidos a zero se a regularização apropriada for usada. Normalmente, o número de unidades ocultas está em algum lugar na faixa de 5 a 100, com o número aumentando com o número de entradas e o número de casos de treinamento. É mais comum derrubar um número razoavelmente grande de unidades e treiná-las com regularização.
Alguns pesquisadores usam a validação cruzada para estimar o número ideal, mas isso parece desnecessário se a validação cruzada for usada para estimar o parâmetro de regularização. A escolha do número de camadas ocultas é guiado pelo conhecimento prévio e pela experimentação. Cada camada extrai recursos da entrada para regressão ou classificação. O uso de várias camadas ocultas permite a construção de recursos hierárquicos em diferentes níveis de resolução.
A função de erro \(R(\theta)\) é não convexa, possuindo muitos mínimos locais. Como resultado, a solução final obtida é bastante dependente da escolha dos pesos iniciais. Deve-se tentar pelo menos uma série de configurações iniciais aleatórias e escolher a solução que dá o erro mais baixo penalizado. Provavelmente, a melhor abordagem é usar as previsões médias sobre a coleção de redes como a previsão final (Ripley, 1996). Isso é preferível à média dos pesos, uma vez que a não linearidade do modelo implica que essa solução meacute;dia pode ser muito pobre. Outra abordagem é por meio de bagging, que calcula a média das previsões de treinamento de redes a partir de versões perturbadas aleatoriamente dos dados de treinamento.
Usaremos o conjunto de dados Boston no pacote MASS. Este conjunto de dados é uma coleção de dados sobre valores habitacionais nos subúrbios de Boston. Nosso objetivo é prever o valor mediano das casas ocupadas pelo proprietário (medv) usando todas as outras variáveis contínuas disponíveis.
O conjunto de dados de Boston possui 506 linhas e 14 colunas, as colunas contém as seguintes informações:
Primeiro, precisamos verificar se nenhum ponto de dados está faltando, caso contrário, precisamos consertar o conjunto de dados.
Não há dados faltando, ótimo. Prosseguimos dividindo aleatoriamente os dados em um grupo de treinamento e um conjunto de teste, então ajustamos um modelo de regressão linear e o testamos no conjunto de teste. Observe que estou usando a função glm() em vez de lm(). Isso se tornará útil mais tarde, na validação cruzada do modelo linear.
A função sample(x, size) simplesmente produz um vetor do tamanho especificado de amostras selecionadas aleatoriamente do vetor x. Por padrão, a amostragem é sem reposição: o índice é essencialmente um vetor aleatório de indeces. Como estamos lidando com um problema de regressão, vamos usar o erro quadrático médio (MSE) como uma medida de quanto nossas previsões estão longe dos dados reais.
Antes de ajustar uma rede neural, alguns preparativos precisam ser feitos. As redes neurais não são tão fáceis de treinar e ajustar. Como uma primeira etapa, vamos abordar o pré-processamento de dados.
É uma boa prática normalizar seus dados antes de treinar uma rede neural. Não consigo enfatizar o suficiente a importância dessa etapa: dependendo do seu conjunto de dados, evitar a normalização pode levar a resultados inúteis ou a um processo de treinamento muito difícil, na maioria das vezes o algoritmo não convergirá antes do número máximo de iterações permitidas. Você pode escolher diferentes métodos para dimensionar os dados: normalização \(z\), escala mín-máx, etc. . Escolhi usar o método min-max e dimensionar os dados no intervalo \([0,1]\). Geralmente escalar nos intervalos \([0,1]\) ou \([-1,1]\) tende a dar melhores resultados. Portanto, dimensionamos e dividimos os dados antes de prosseguir:
Observe que scale retorna uma matriz que precisa ser alocada numa data.frame.
Pelo que sabemos, não existe uma regra fixa sobre quantas camadas e neurônios usar, embora existam várias regras básicas mais ou menos aceitas. Normalmente, se necessário, uma camada oculta é suficiente para um grande número de aplicações. No que diz respeito ao número de neurônios, ele deve estar entre o tamanho da camada de entrada e o tamanho da camada de saída, geralmente 2/3 do tamanho de entrada. Pelo menos em minha breve experiência, testar repetidas vezes é a melhor solução, pois não há garantia de que qualquer uma dessas regras se encaixará melhor em seu modelo.
Como este é um exemplo simples, usaremos 2 camadas ocultas com esta configuração: 13: 5: 3: 1. A camada de entrada tem 13 entradas, as duas camadas ocultas têm 5 e 3 neurônios e a camada de saída tem, é claro, uma única saída, já que estamos fazendo a regressão. Vamos ajustar a rede:
Algumas observações:
Esta é a representação gráfica do modelo com os pesos em cada conexão:
 |
|---|
As linhas pretas mostram as conexões entre cada camada e os pesos em cada conexão, enquanto as linhas azuis mostram o termo de polarização adicionado em cada etapa. O viés pode ser pensado como o intercepto de um modelo linear. A rede é essencialmente uma caixa preta, então não podemos dizer muito sobre o encaixe, os pesos e o modelo. Basta dizer que o algoritmo de treinamento convergiu e, portanto, o modelo está pronto para ser usado.
Beck, (2013) descreve o gráfico acima como “deixando muito a desejar”. Essas parcelas são difíceis de trabalhar. Visualmente, eles sofrem de um sério problema de desordem, tornando os pesos de interação praticamente ilegíveis. Além disso, a função plot.nn tem alguns comportamentos indesejáveis. No ambiente RStudio, os gráficos aparecem como pop-outs em vez de na janela do visualizador integrado. A função também não parece ser compatível com o knitr, então as imagens acima foram exportadas manualmente e incluídas como arquivos .png estáticos.
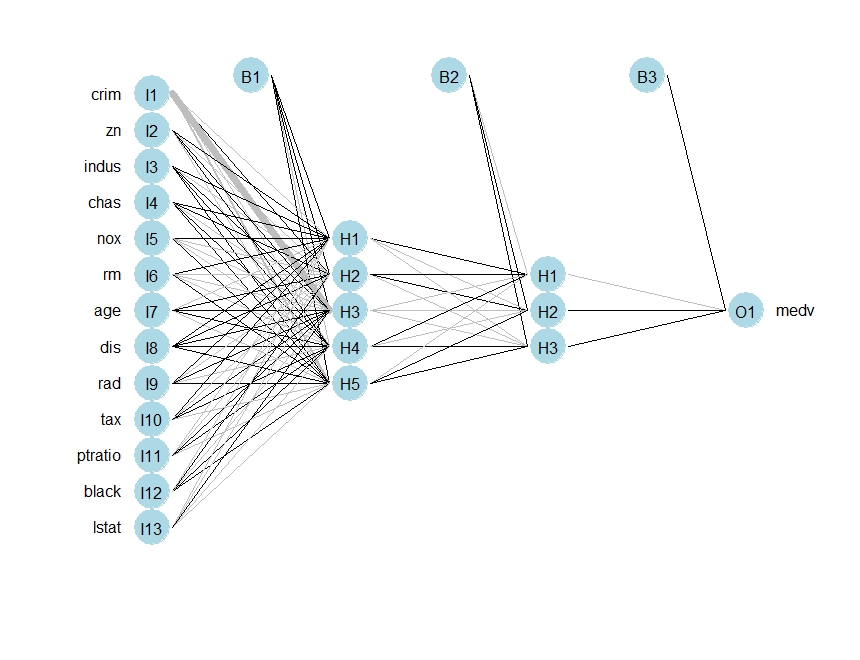
Esta questão foi abordada pelo Diagrama de Interpretação Neural (NID) descrito em Olden & Jackson, (2002) e implementado na biblioteca NeuralNetTools, Beck, (2015) como a função plotnet(). Isso fornece os seguintes gráficos padrão para os mesmos modelos.
 |
|---|
Este gráfico elegante resolve o problema de desordem visual usando a espessura da linha para representar a magnitude do peso e a cor da linha para representar o sinal do peso (preto = positivo, cinza = negativo). Outros recursos úteis incluem uma opção de cor para a camada de entrada para que, por exemplo, a importância variável possa ser representada. Isso será discutido no final.
A função de ativação padrão é a logística. Utilizamos agora uma outra função de ativação:
Agora podemos tentar prever os valores para o conjunto de teste e calcular o MSE. Lembre-se de que a rede produzirá uma previsão normalizada, portanto, precisamos redimensioná-la para fazer uma comparação significativa ou apenas uma previsão simples.
Aparentemente, a rede está fazendo um trabalho melhor do que o modelo linear na previsão do medv. Mais uma vez, tome cuidado porque esse resultado depende da divisão treino-teste realizada acima. A seguir, após o gráfico visual, faremos uma validação cruzada rápida para termos mais confiança nos resultados.
Uma primeira abordagem visual para o desempenho da rede e do modelo linear no conjunto de teste é apresentada abaixo:
 |
|---|
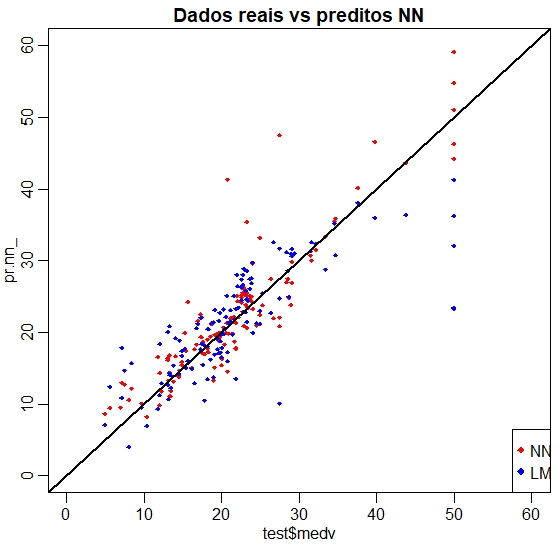
Ao inspecionar visualmente o gráfico, podemos ver que as previsões feitas pela rede neural são, em geral, mais concentradas em torno da linha; um alinhamento perfeito com a linha indicaria um MSE de 0 e, portanto, uma previsão perfeita ideal; do que aquelas feitas por o modelo linear.
Uma comparação visual talvez mais útil é mostrada abaixo:
 |
|---|
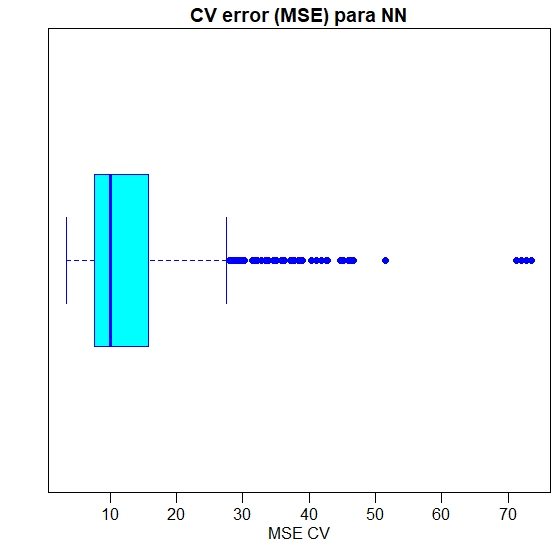
A validação cruzada é outra etapa muito importante na construção de modelos preditivos. Embora existam diferentes tipos de métodos de validação cruzada, a ideia básica é repetir o seguinte processo várias vezes:
 |
|---|
Como podemos ver, o MSE médio da rede neural (13.14) é inferior ao do modelo linear embora pareça haver um certo grau de variação nos MSEs da validação cruzada. Isso pode depender da divisão dos dados ou da inicialização aleatória dos pesos na rede. Ao executar a simulação em tempos diferentes com sementes diferentes, podemos obter uma estimativa pontual mais precisa para o MSE médio.
As redes neurais se parecem muito com caixas pretas: explicar seu resultado é muito mais difícil do que explicar o resultado de um modelo mais simples, como um modelo linear. Portanto, dependendo do tipo de aplicação de que se precise, convém levar em consideração esse fator também. Além disso, como vemos acima, é necessário cuidado extra para ajustar uma rede neural e pequenas mudanças podem levar a resultados diferentes.
Os dados referem-se a campanhas de marketing direto (telefonemas) de uma instituição bancária portuguesa. O objetivo da classificação é prever se o cliente fará um depósito a prazo, variável resposta.
Os dados referem-se a campanhas de marketing direto de uma instituição bancária portuguesa. As campanhas de marketing foram baseadas em ligações telefônicas. Muitas vezes, era necessário mais de um contato para o mesmo cliente, para acessar se o produto, depósito bancário a prazo, seria "sim" ou "não".
Existem quatro conjuntos de dados:
O objetivo da classificação é prever se o cliente irá subscrever ou não (sim/não) um depósito a prazo (variável y).
Variáveis de entrada no banco de dados do cliente:
Este conjunto de dados está disponível ao público para pesquisa. Os detalhes estão descritos no artigo
S.Moro, P.Cortez e P. Rita. A Data-Driven Approach to Predict the Success of Bank Telemarketing. Decision Support Systems, Elsevier, 62:22-31, June 2014.
Artigo relevante relacionado:
S. Moro, R. Laureano and P. Cortez. Using Data Mining for Bank Direct Marketing: An Application of the CRISP-DM Methodology. In P. Novais et al. (Eds.), Proceedings of the European Simulation and Modelling Conference - ESM'2011, pp. 117-121, Guimaraes, Portugal, October, 2011. EUROSIS.
Este e outros bancoa de dados estão disponíveis em
Machine Learning Repository, Center for Machine Learning and Intelligent System.
Observemos nossa base de dados. Fizemos um tabulamento da quantidade de observações em cada categoria dos fatores.
Como as redes neurais usam funções de ativação entre -1 e +1 é importante reduzir suas variáveis. Caso contrário, a rede neural terá que gastar iterações de treinamento fazendo esse dimensionamento para você.
Para representar variáveis de fatores, precisamos convertê-las em variáveis dummy. Uma variável dummy transforma os N valores distintos e os converte em N-1 variáveis. Usamos N-1 porque o valor final é representado por todos os valores fictícios definidos como zero.
Para qualquer linha, uma ou nenhuma das variáveis dummy estará ativa com um (1) ou inativa com zero (0).
Em nosso conjunto de dados bancários, a variável educação tem quatro valores distintos com "primário" (primary) sendo o caso base, ou seja, o primeiro nível. Para tornar esta variável de fator útil para o pacote neuralnet, precisamos usar a função model.matrix ().
A função model.matrix divide a variável education em todos os valores possíveis, exceto o caso base. Ele adiciona uma variável de Intercept que, eventualmente, abandonaremos. Se quiser incluir esse valor espec7iacute;fico, você precisa relevel() seus dados.
Depois de decidir sobre todas as nossas variáveis numéricas e de fator, podemos chamar a função model.matrix uma última vez.
Agora temos uma matriz com 28 colunas, 27 excluindo o Intercept. Usando essa nova variável, podemos começar a construir nossa rede neural.
Antes de chegarmos à construção do modelo, precisamos ter certeza de que todos os nomes das colunas são entradas de modelo aceitáveis. Parece que duas colunas têm caracteres especiais e precisamos consertar isso antes de inseri-las em um modelo.
Agora que todos os nomes das colunas foram limpos, precisamos colocá-los em uma fórmula. Temos que combinar os nomes das colunas (separados por um símbolo de adição) e, em seguida, voltar à variável de resposta.
Finalmente, estamos prontos para usar esta fórmula em nossos modelos.
Com a complexidade das redes neurais, há muitas opções a serem exploradas no pacote neuralnet. Vamos começar com os parâmetros padrão e, em seguida, explorar algumas opções diferentes.
 |
|---|
Uma rede neural com um único nó oculto não é nada melhor do que uma combinação linear, na verdade. Para alterar o número de nós ocultos, simplesmente usamos o parâmetro hidden.
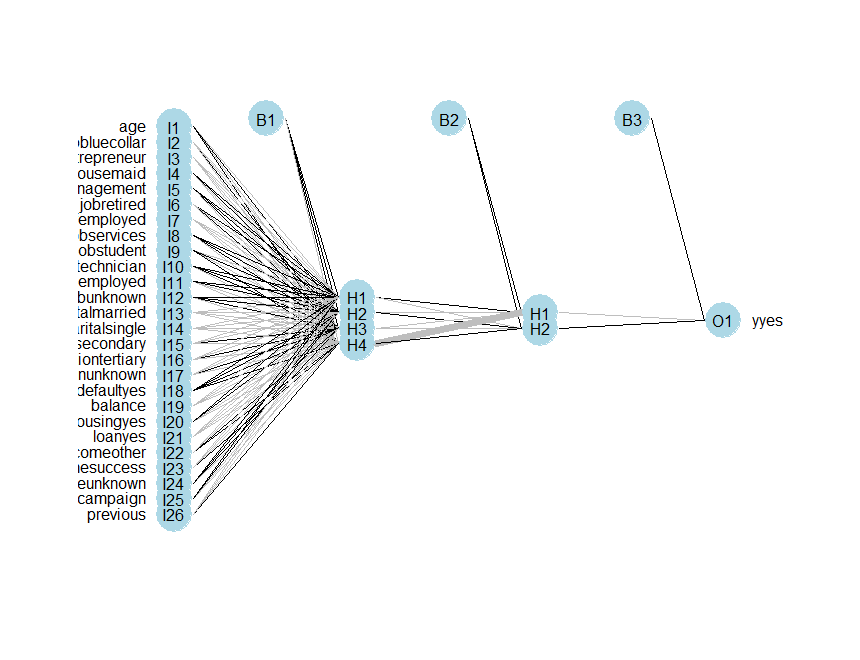 |
|---|
A melhor solução que encontrei considera apenas manter a taxa de aprendizagem relativamente pequena.
Uma outra alternativa é mostrada a seguir; desta maneir conseguimos diminuir em 41% o erro de estimação.
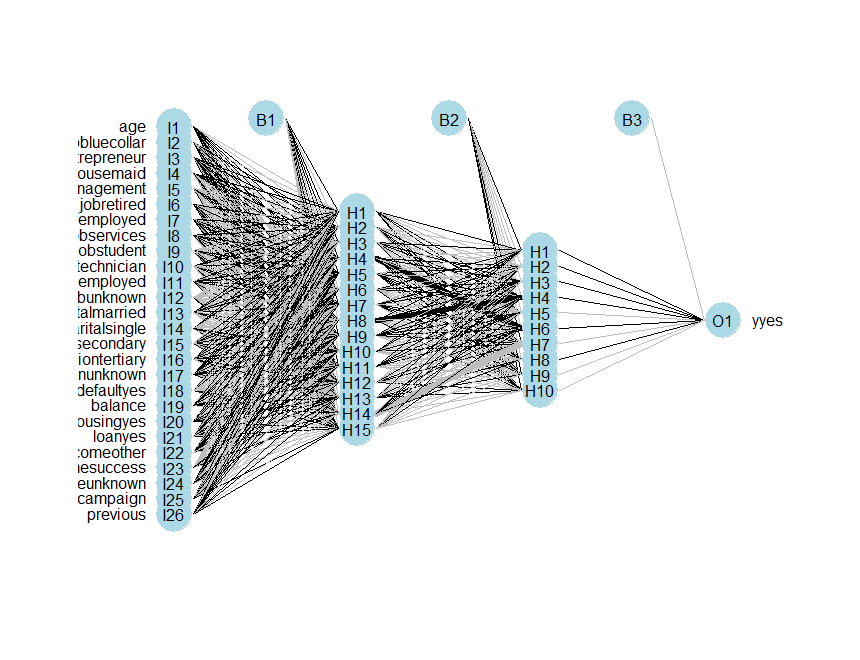 |
|---|
Por padrão, o algoritmo neuralnet usando o algoritmo Resilient Backpropogation (RPROP+). Isso requer um learningrate.limit, ou seja, um vetor ou uma lista contendo o limite inferior e superior para a taxa de aprendizagem e um learningrate.factor, um vetor ou uma lista contendo os fatores de multiplicação para a taxa de aprendizagem superior e inferior.
Agora você sabe quase tudo sobre como usar o pacote neuralnet em R. Existem muitos parâmetros diferentes para mexer e você pode gerar alguns layouts de rede neural complicados com alguns comandos simples.