# Pacotes. ----------------------------------
library(tidyverse)
library(readxl)
source("https://raw.githubusercontent.com/walmes/wzRfun/master/R/pairwise.R")
insert_absorb <- multcomp:::insert_absorbExperimento 3
# Importação. -------------------------------
tb <- read_excel("Pomar Curitiba.xlsx", sheet = 1, skip = 1)
# str(tb)
# names(tb) |>
# setNames(tolower(names(tb))) |>
# dput()
lookup <- c(
product = "Product",
spray = "Spray",
species = "Species",
block = "Block",
audpc = "AUDPC",
sevmax = "Sev Max (%)",
defol = "Defoliation (%)"
)
tb <- tb |>
rename(all_of(lookup))
str(tb)tibble [56 × 7] (S3: tbl_df/tbl/data.frame)
$ product: chr [1:56] "B. alcalophilus" "B. alcalophilus" "B. alcalophilus" "B. alcalophilus" ...
$ spray : chr [1:56] "Conventional" "Conventional" "Conventional" "Conventional" ...
$ species: chr [1:56] "C. fructicola" "C. fructicola" "C. fructicola" "C. fructicola" ...
$ block : num [1:56] 1 2 3 4 1 2 3 4 1 2 ...
$ audpc : num [1:56] 106.8 NA 14.4 14.1 49.6 ...
$ sevmax : num [1:56] 4.48 NA 1.73 1.32 2.9 1.8 4.87 2.57 2.31 1.7 ...
$ defol : num [1:56] 0 NA 10 10 0 10 0 10 10 0 ...# Deixa testemunha como último nível.
tb <- tb |>
mutate(product = fct_relevel(product, "none", after = Inf),
spray = fct_relevel(spray, "none", after = Inf)) |>
mutate(product = fct_recode(product, "None" = "none"),
spray = fct_recode(spray, "None" = "none"),
block = factor(block))
xtabs(~product + spray, data = tb) spray
product Conventional Electrostatic None
B. alcalophilus 8 8 0
Manzate® 8 8 0
Serenade® 8 8 0
None 0 0 8DataExplorer::profile_missing(tb)# A tibble: 7 × 3
feature num_missing pct_missing
<fct> <int> <dbl>
1 product 0 0
2 spray 0 0
3 species 0 0
4 block 0 0
5 audpc 2 0.0357
6 sevmax 2 0.0357
7 defol 2 0.0357# Remove valores ausentes.
tb <- tb |>
drop_na(audpc, sevmax, defol)# Análise exploratória. ---------------------
tb |>
pivot_longer(cols = audpc:defol,
names_to = "variable",
values_to = "values") |>
ggplot(data = _,
mapping = aes(x = product, y = values, color = spray)) +
facet_wrap(facets = ~variable, scale = "free_y", ncol = 1) +
# geom_point()
geom_jitter(width = 0.05)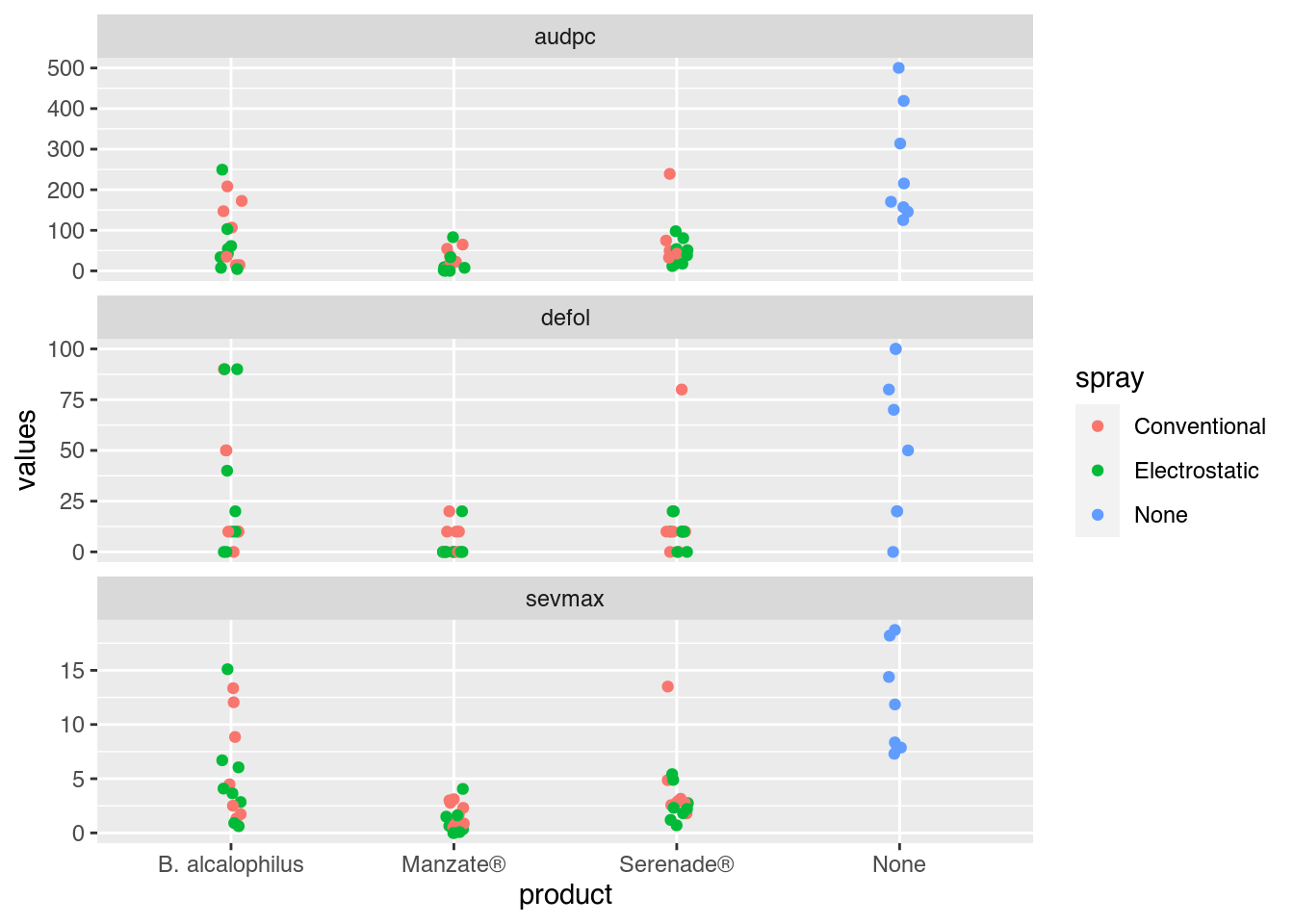
1 Área abaixo da curva de progresso da doença (AUDPC)
ggplot(data = tb,
mapping = aes(x = product,
y = audpc)) +
facet_grid(facets = . ~ spray,
scale = "free",
space = "free") +
geom_jitter(alpha = 0.5,
width = 0.1,
height = 0) +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun = "mean") +
labs(x = "Product",
y = "Area under disease progress curve (AUDPC)")`geom_line()`: Each group consists of only one observation.
ℹ Do you need to adjust the group aesthetic?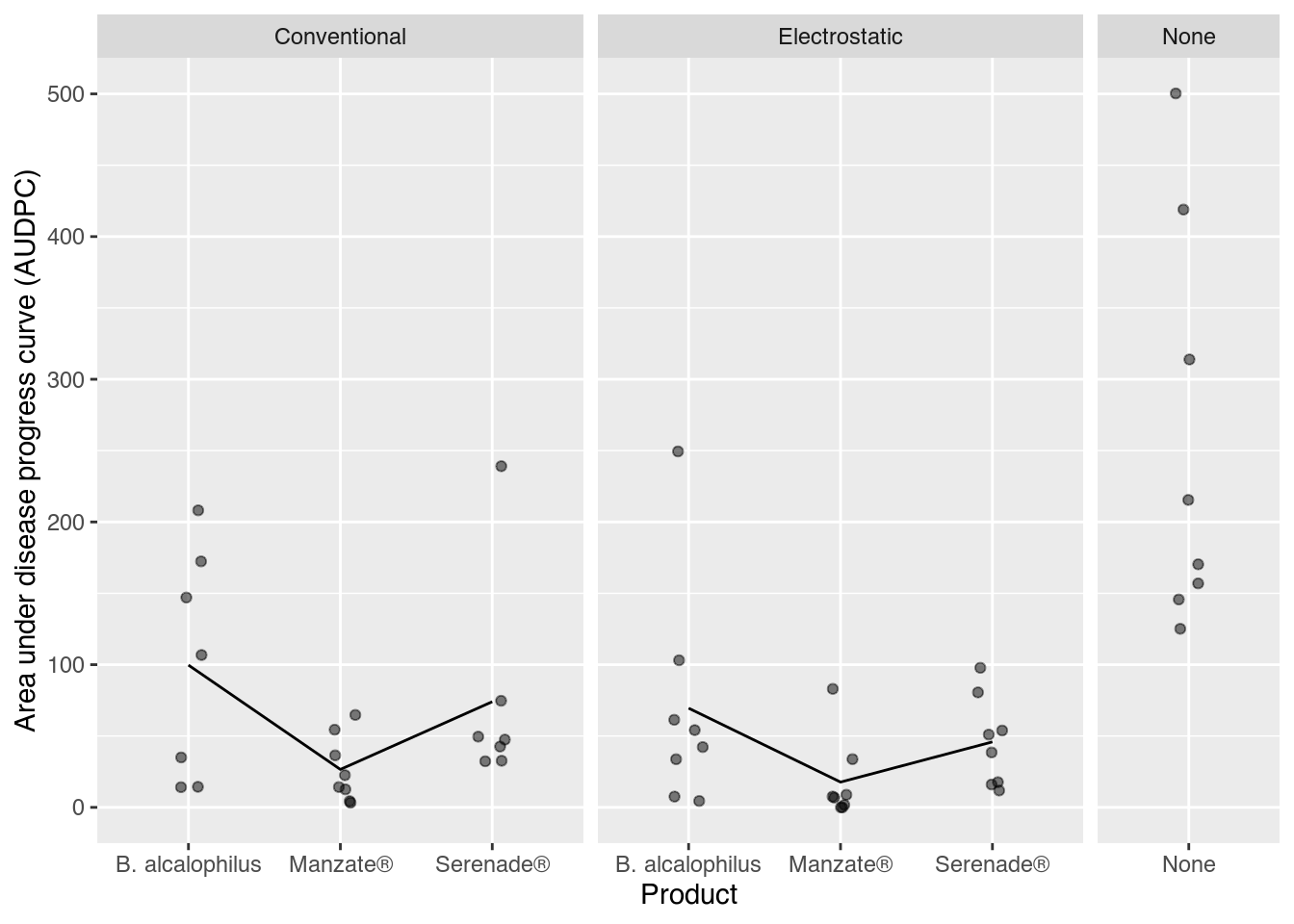
# Ajuste do modelo. -------------------------
# Ajuste do modelo via especificação por fórmula.
m0 <- lm(audpc + 0.1 ~ block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)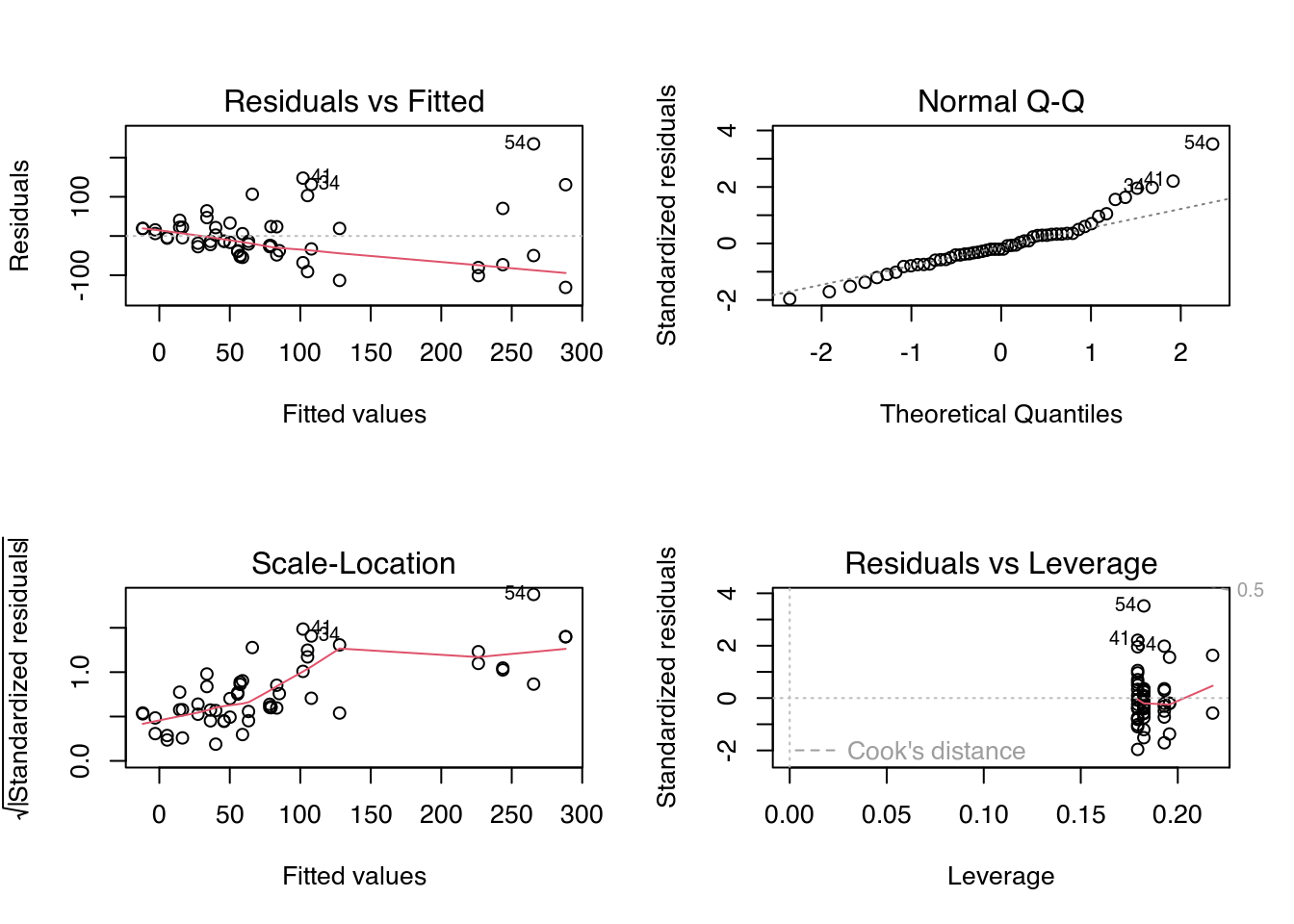
layout(1)
# Verifica necessidade de transformação.
MASS::boxcox(m0)
abline(v = 1/3, col = "red")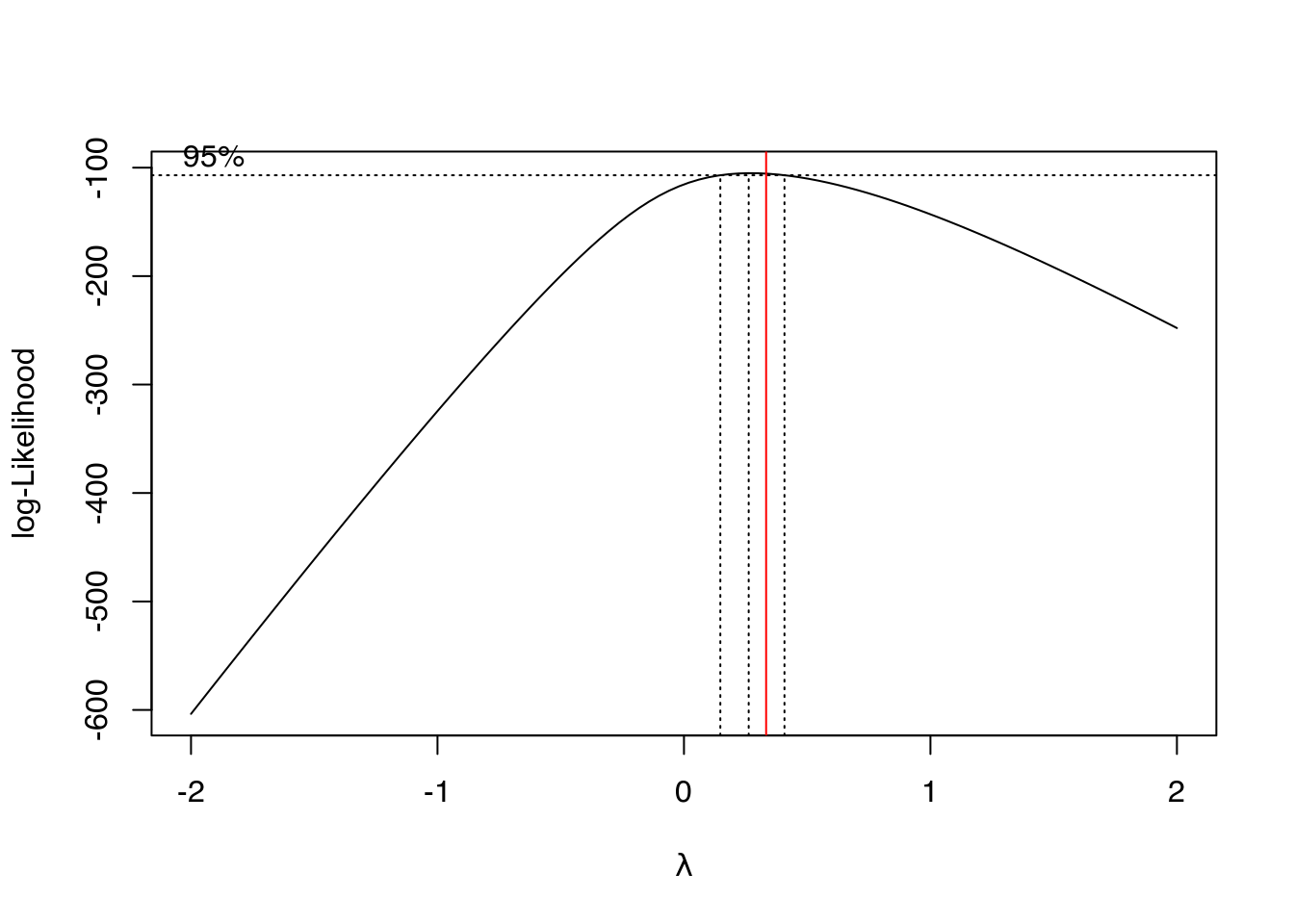
# Atualiza o modelo.
m0 <- update(m0, .^(1/3) ~ .)
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)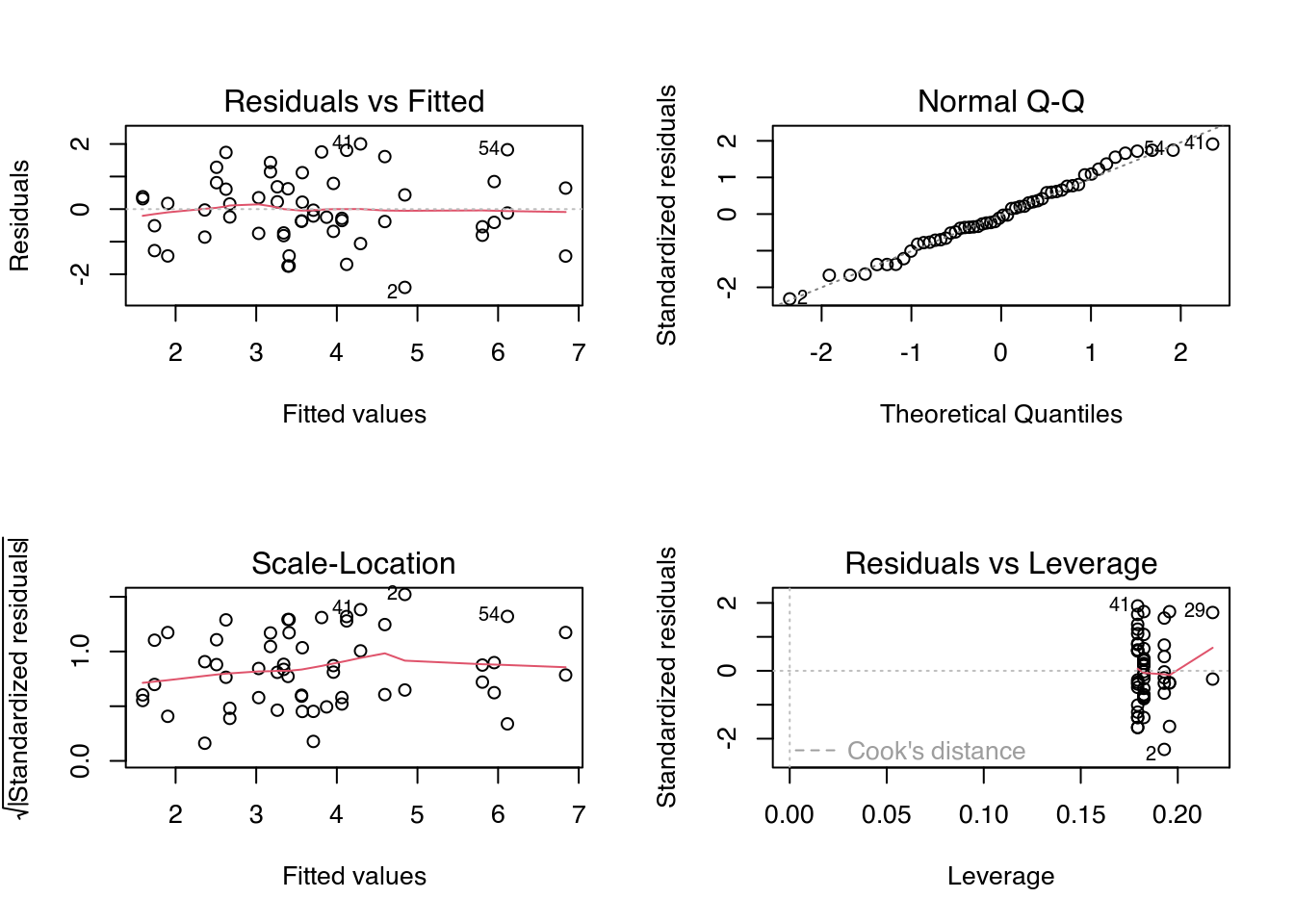
layout(1)
# Quadro de análise de variância.
anova(m0)Analysis of Variance Table
Response: (audpc + 0.1)^(1/3)
Df Sum Sq Mean Sq F value Pr(>F)
block 3 9.060 3.0199 2.2604 0.09464 .
product 3 78.867 26.2891 19.6779 3.071e-08 ***
spray 1 4.412 4.4117 3.3023 0.07600 .
product:spray 2 0.137 0.0683 0.0511 0.95022
Residuals 44 58.783 1.3360
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Decomposição da SQ. -----------------------
# Cria a matriz do delineamento completa.
X_full <- model.matrix(~block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Modelo ajustado passando a matriz completa.
m1 <- aov(audpc^(1/3) ~ 0 + X_full, data = tb)
# coef(m1)
# Efeitos.
ef <- list("bloc" = 2:4,
"FvsA" = 7,
"prod" = 5:6,
"spray" = 8,
"prod:spray" = 9:10)
# Desdobramento das somas de quadrados.
summary(m1, split = list("X_full" = ef)) Df Sum Sq Mean Sq F value Pr(>F)
X_full 10 832.9 83.29 59.315 < 2e-16 ***
X_full: bloc 3 9.4 3.13 2.227 0.098313 .
X_full: FvsA 1 57.4 57.45 40.913 8.81e-08 ***
X_full: prod 2 24.2 12.11 8.624 0.000692 ***
X_full: spray 1 5.0 5.02 3.575 0.065234 .
X_full: prod:spray 2 0.3 0.15 0.110 0.896235
Residuals 44 61.8 1.40
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Comparações de médias. --------------------
# Matriz do modelo descartadas as linhas repetidas, caso hajam.
X_un <- unique(X_full)
# Fatores do modelo, removidas as linhas repetidas também.
pts <- subset(tb, select = names(attr(X_full, "contrasts"))) |>
unique()
# Testa se os tamanhos são iguais.
stopifnot(nrow(pts) == nrow(X_un))
# Cria um objeto agregado considerando os níveis de fonte e modo.
grp <- pts |>
group_by(product, spray)
# Matriz com termos para determinar médias ajustadas para os pontos
# experimentais.
pts <- group_keys(grp)
X_ls <-
by(X_un,
INDICES = group_indices(grp),
FUN = colMeans) %>%
do.call(what = rbind)
# X_ls
# Remove efeitos não estimados.
X_ls <- X_ls[, !is.na(coef(m0))]
# Entenda os pesos, principalmente para os níveis de bloco.
MASS::fractions(t(X_ls)) 1 2 3 4 5 6 7
(Intercept) 1 1 1 1 1 1 1
block2 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
product1 -1 -1 1 1 0 0 0
product2 -1 -1 -1 -1 2 2 0
product3 -1 -1 -1 -1 -1 -1 3
spray1 -1 1 -1 1 -1 1 0
product1:spray1 1 -1 -1 1 0 0 0
product2:spray1 1 -1 1 -1 -2 2 0# Médias amostrais e ajustadas apenas para conferir.
tb |>
group_by(product, spray) |>
summarise("média amostral" = mean(audpc^(1/3), na.rm = TRUE),
.groups = "drop") |>
bind_cols("média ajustada" = c(X_ls %*% na.omit(coef(m0))))# A tibble: 7 × 4
product spray `média amostral` `média ajustada`
<fct> <fct> <dbl> <dbl>
1 B. alcalophilus Conventional 4.23 4.18
2 B. alcalophilus Electrostatic 3.63 3.63
3 Manzate® Conventional 2.73 2.73
4 Manzate® Electrostatic 1.84 1.96
5 Serenade® Conventional 3.94 3.93
6 Serenade® Electrostatic 3.40 3.40
7 None None 6.18 6.18# Comparação dos produtos. ------------------
# Funções lineares para média ajustada para níveis de produto.
L_prod <- by(X_ls, INDICES = pts$product, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_prod)) B. alcalophilus Manzate® Serenade® None
(Intercept) 1 1 1 1
block2 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4
product1 -1 1 0 0
product2 -1 -1 2 0
product3 -1 -1 -1 3
spray1 0 0 0 0
product1:spray1 0 0 0 0
product2:spray1 0 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
# c(deviance(m2), deviance(m0))
results <-
apmc(X = L_prod,
model = m2,
focus = "product",
cld2 = TRUE) %>%
mutate(product = factor(product, levels = levels(tb$product)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
results product fit lwr upr cld
1 B. alcalophilus 59.70892 36.081151 91.89545 b
2 Manzate® 12.96704 5.516942 25.19775 c
3 Serenade® 49.37648 28.780153 78.00630 b
4 None 235.75305 153.474733 343.17251 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = product, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Product",
y = "Area under disease progress curve")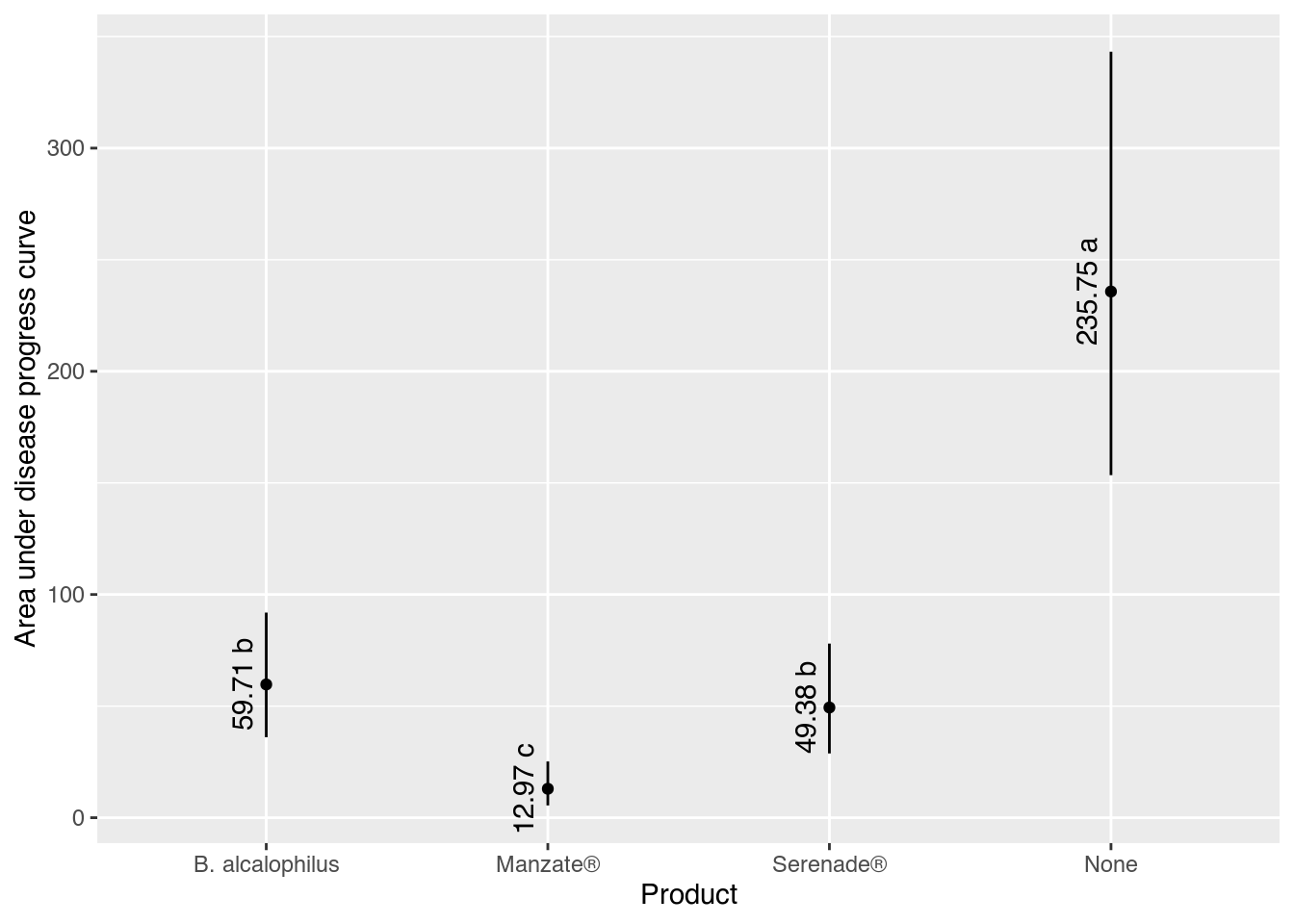
# Comparação dos sprays. --------------------
# Funções lineares para média ajustada para níveis de produto.
L_spray <- by(X_ls, INDICES = pts$spray, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_spray)) Conventional Electrostatic None
(Intercept) 1 1 1
block2 1/4 1/4 1/4
block3 1/4 1/4 1/4
block4 1/4 1/4 1/4
product1 0 0 0
product2 0 0 0
product3 -1 -1 3
spray1 -1 1 0
product1:spray1 0 0 0
product2:spray1 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
results <-
apmc(X = L_spray,
model = m2,
focus = "spray",
cld2 = TRUE) %>%
mutate(spray = factor(spray, levels = levels(tb$spray)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
results spray fit lwr upr cld
1 Conventional 47.33657 30.33853 69.72917 b
2 Electrostatic 27.00713 16.09402 41.99034 b
3 None 235.75305 153.47473 343.17251 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = spray, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Spray",
y = "Area under disease progress curve")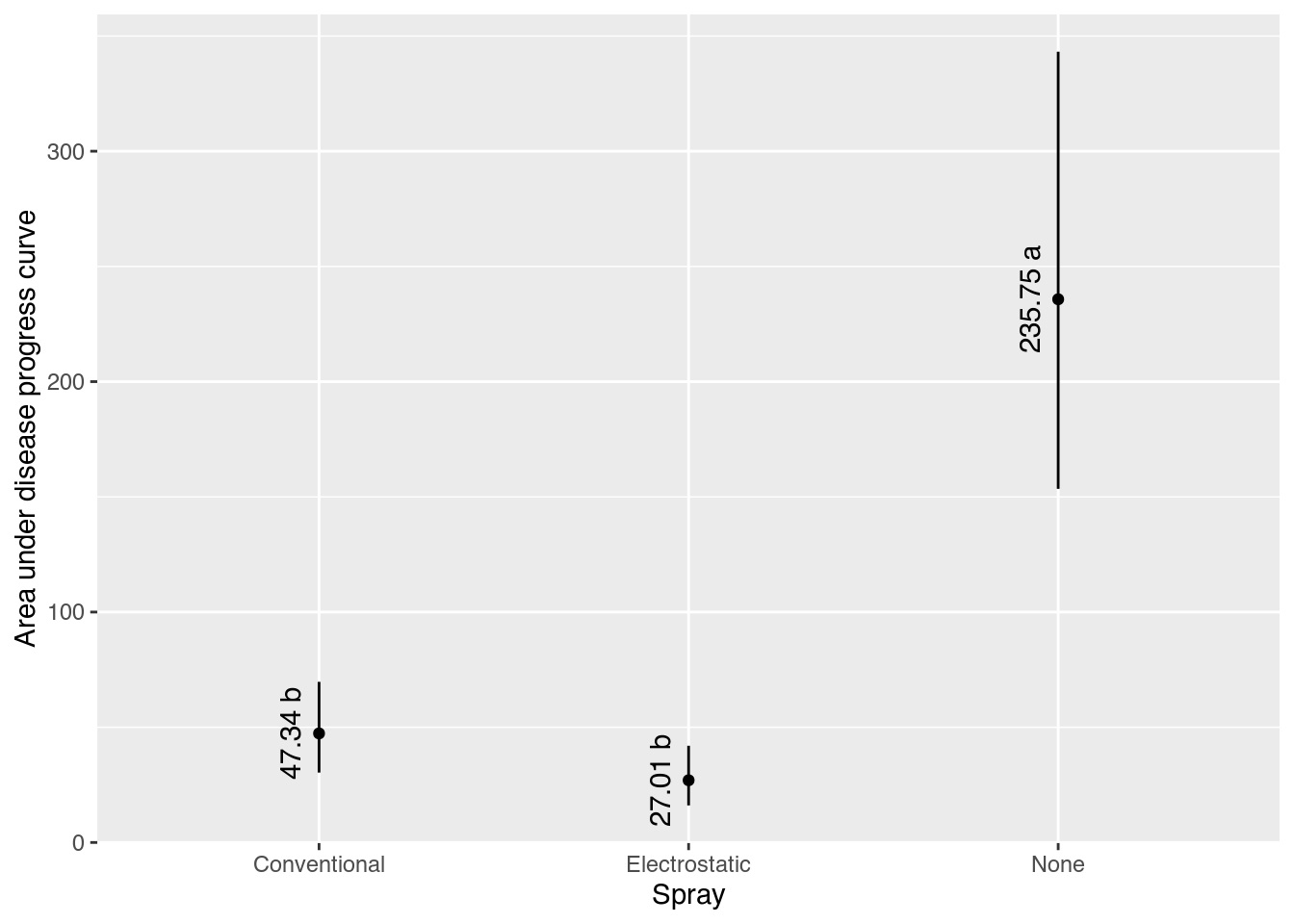
2 Desfolha
ggplot(data = tb,
mapping = aes(x = product,
y = defol)) +
facet_grid(facets = . ~ spray,
scale = "free",
space = "free") +
geom_jitter(alpha = 0.5,
width = 0.1,
height = 0) +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun = "mean") +
labs(x = "Product",
y = "Defoliation")`geom_line()`: Each group consists of only one observation.
ℹ Do you need to adjust the group aesthetic?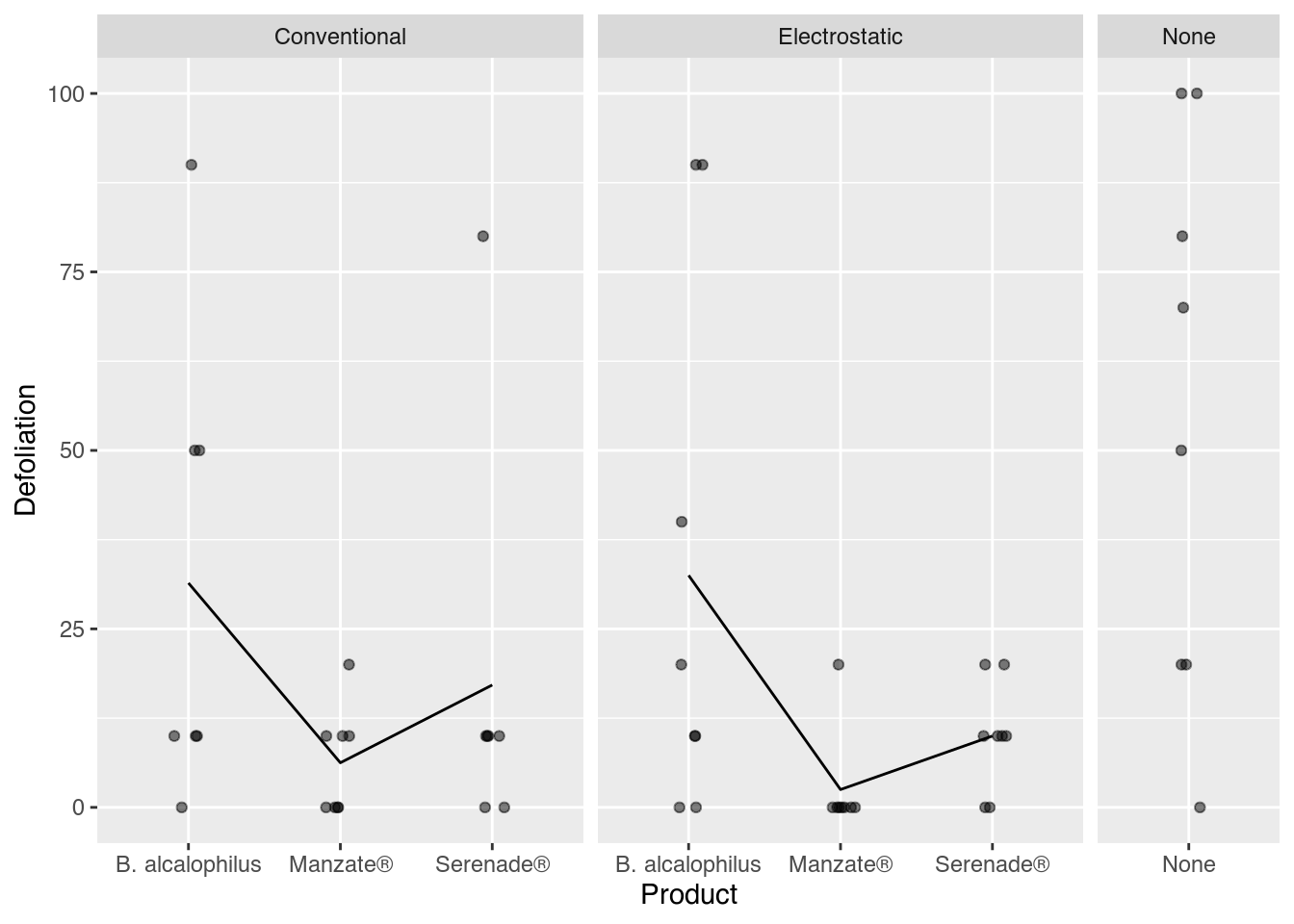
# Ajuste do modelo. -------------------------
# Ajuste do modelo via especificação por fórmula.
m0 <- lm(defol + 0.1 ~ block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)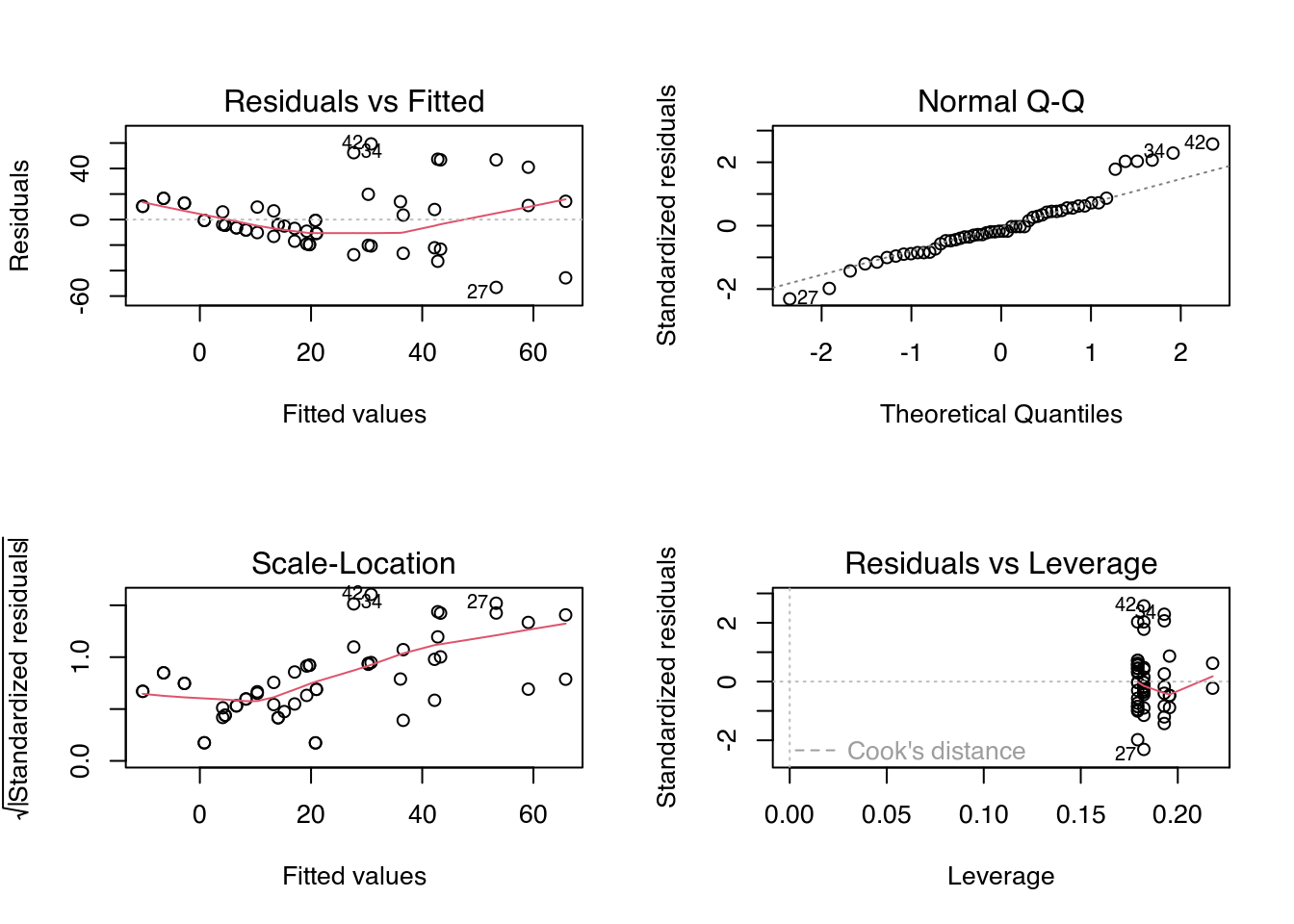
layout(1)
# Verifica necessidade de transformação.
MASS::boxcox(m0)
abline(v = 1/3, col = "red")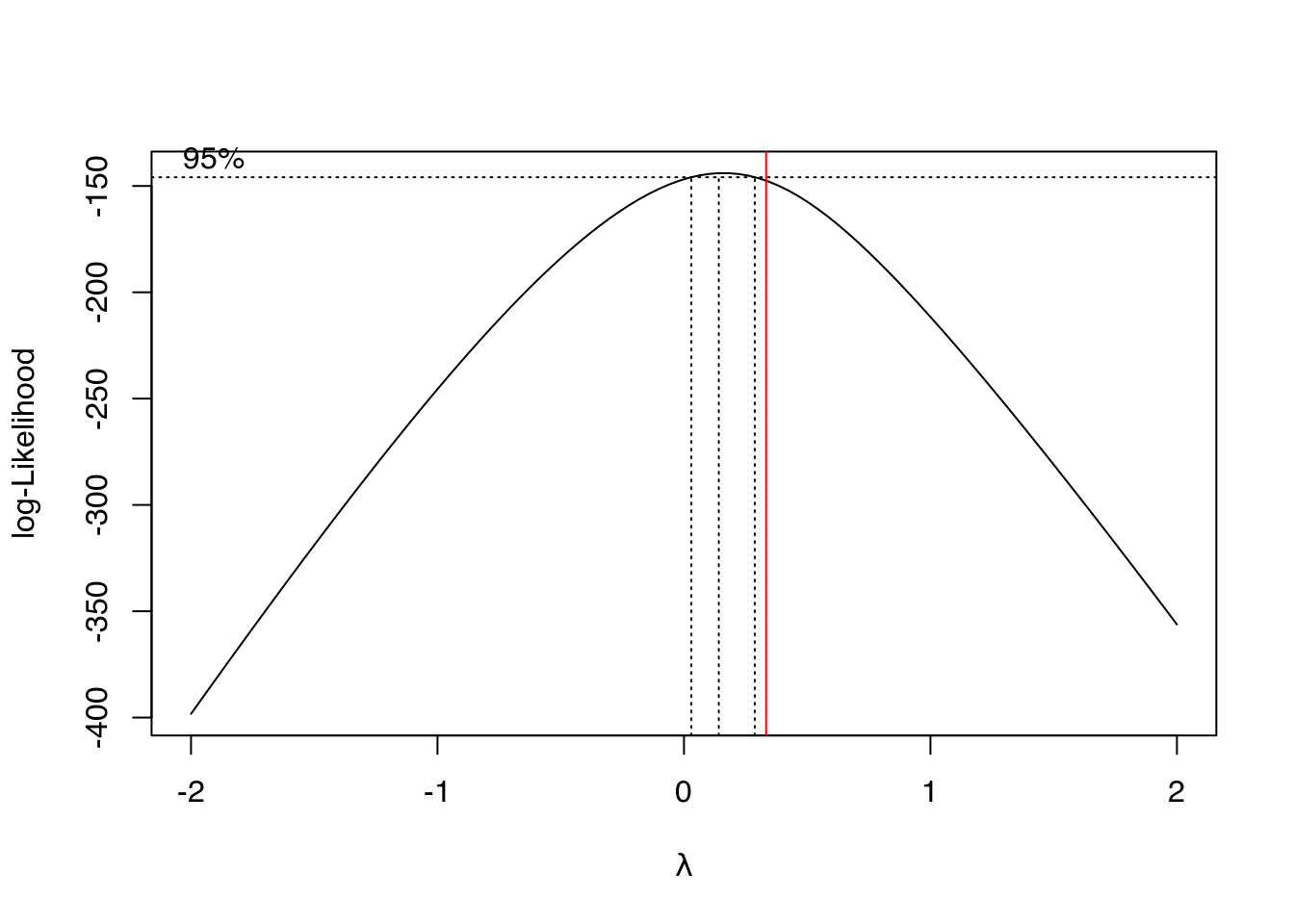
# Atualiza o modelo.
m0 <- update(m0, defol^(1/3) ~ .)
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)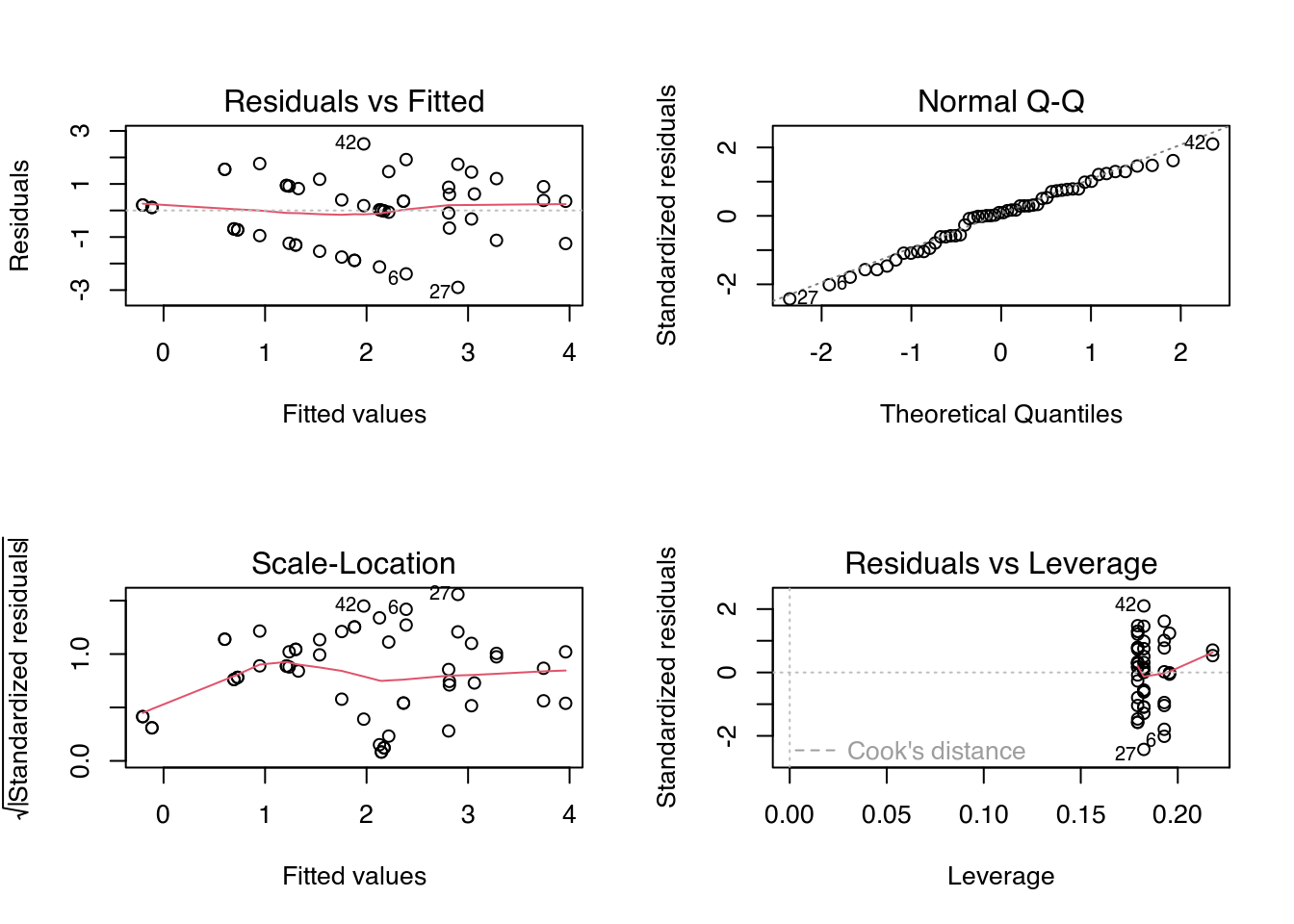
layout(1)
# Quadro de análise de variância.
anova(m0)Analysis of Variance Table
Response: defol^(1/3)
Df Sum Sq Mean Sq F value Pr(>F)
block 3 13.311 4.4369 2.5402 0.0685876 .
product 3 44.675 14.8917 8.5257 0.0001415 ***
spray 1 1.574 1.5743 0.9013 0.3476212
product:spray 2 1.264 0.6322 0.3619 0.6983764
Residuals 44 76.854 1.7467
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Decomposição da SQ. -----------------------
# Cria a matriz do delineamento completa.
X_full <- model.matrix(~block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Modelo ajustado passando a matriz completa.
m1 <- aov(defol^(1/3) ~ 0 + X_full, data = tb)
# coef(m1)
# Efeitos.
ef <- list("bloc" = 2:4,
"FvsA" = 7,
"prod" = 5:6,
"spray" = 8,
"prod:spray" = 9:10)
# Desdobramento das somas de quadrados.
summary(m1, split = list("X_full" = ef)) Df Sum Sq Mean Sq F value Pr(>F)
X_full 10 258.86 25.886 14.820 4.56e-11 ***
X_full: bloc 3 13.31 4.437 2.540 0.06859 .
X_full: FvsA 1 18.99 18.986 10.870 0.00194 **
X_full: prod 2 25.69 12.845 7.354 0.00176 **
X_full: spray 1 1.57 1.574 0.901 0.34762
X_full: prod:spray 2 1.26 0.632 0.362 0.69838
Residuals 44 76.85 1.747
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Comparações de médias. --------------------
# Matriz do modelo descartadas as linhas repetidas, caso hajam.
X_un <- unique(X_full)
# Fatores do modelo, removidas as linhas repetidas também.
pts <- subset(tb, select = names(attr(X_full, "contrasts"))) |>
unique()
# Testa se os tamanhos são iguais.
stopifnot(nrow(pts) == nrow(X_un))
# Cria um objeto agregado considerando os níveis de fonte e modo.
grp <- pts |>
group_by(product, spray)
# Matriz com termos para determinar médias ajustadas para os pontos
# experimentais.
pts <- group_keys(grp)
X_ls <-
by(X_un,
INDICES = group_indices(grp),
FUN = colMeans) %>%
do.call(what = rbind)
# X_ls
# Remove efeitos não estimados.
X_ls <- X_ls[, !is.na(coef(m0))]
# Entenda os pesos, principalmente para os níveis de bloco.
MASS::fractions(t(X_ls)) 1 2 3 4 5 6 7
(Intercept) 1 1 1 1 1 1 1
block2 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
product1 -1 -1 1 1 0 0 0
product2 -1 -1 -1 -1 2 2 0
product3 -1 -1 -1 -1 -1 -1 3
spray1 -1 1 -1 1 -1 1 0
product1:spray1 1 -1 -1 1 0 0 0
product2:spray1 1 -1 1 -1 -2 2 0# Médias amostrais e ajustadas apenas para conferir.
tb |>
group_by(product, spray) |>
summarise("média amostral" = mean(defol^(1/3), na.rm = TRUE),
.groups = "drop") |>
bind_cols("média ajustada" = c(X_ls %*% na.omit(coef(m0))))# A tibble: 7 × 4
product spray `média amostral` `média ajustada`
<fct> <fct> <dbl> <dbl>
1 B. alcalophilus Conventional 2.62 2.67
2 B. alcalophilus Electrostatic 2.43 2.43
3 Manzate® Conventional 1.15 1.15
4 Manzate® Electrostatic 0.339 0.339
5 Serenade® Conventional 1.85 1.78
6 Serenade® Electrostatic 1.76 1.76
7 None None 3.35 3.35 # Comparação dos produtos. ------------------
# Funções lineares para média ajustada para níveis de produto.
L_prod <- by(X_ls, INDICES = pts$product, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_prod)) B. alcalophilus Manzate® Serenade® None
(Intercept) 1 1 1 1
block2 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4
product1 -1 1 0 0
product2 -1 -1 2 0
product3 -1 -1 -1 3
spray1 0 0 0 0
product1:spray1 0 0 0 0
product2:spray1 0 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
# c(deviance(m2), deviance(m0))
results <-
apmc(X = L_prod,
model = m2,
focus = "product",
cld2 = TRUE) %>%
mutate(product = factor(product, levels = levels(tb$product)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
results product fit lwr upr cld
1 B. alcalophilus 16.558338 6.414660e+00 33.999157 ab
2 Manzate® 0.410601 4.631824e-04 2.798127 c
3 Serenade® 5.534275 1.252960e+00 14.879672 bc
4 None 37.705740 1.402488e+01 79.228912 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = product, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Product",
y = "Defoliation")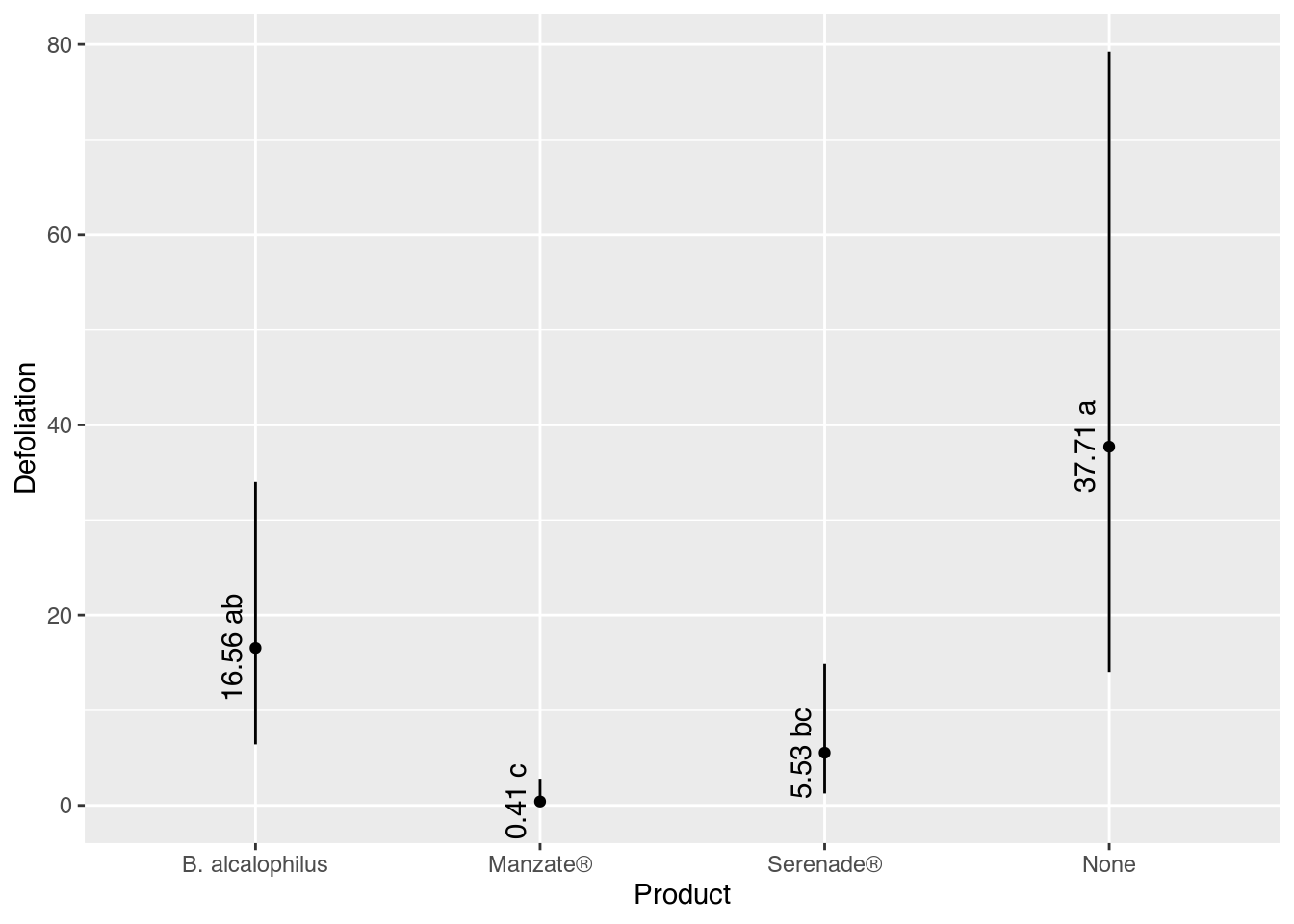
# Comparação dos sprays. --------------------
# Funções lineares para média ajustada para níveis de produto.
L_spray <- by(X_ls, INDICES = pts$spray, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_spray)) Conventional Electrostatic None
(Intercept) 1 1 1
block2 1/4 1/4 1/4
block3 1/4 1/4 1/4
block4 1/4 1/4 1/4
product1 0 0 0
product2 0 0 0
product3 -1 -1 3
spray1 -1 1 0
product1:spray1 0 0 0
product2:spray1 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
results <-
apmc(X = L_spray,
model = m2,
focus = "spray",
cld2 = TRUE) %>%
mutate(spray = factor(spray, levels = levels(tb$spray)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
results spray fit lwr upr cld
1 Conventional 6.507576 2.1814868 14.473763 b
2 Electrostatic 3.422198 0.8937961 8.623375 b
3 None 37.705740 14.0248762 79.228912 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = spray, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Spray",
y = "Defoliation")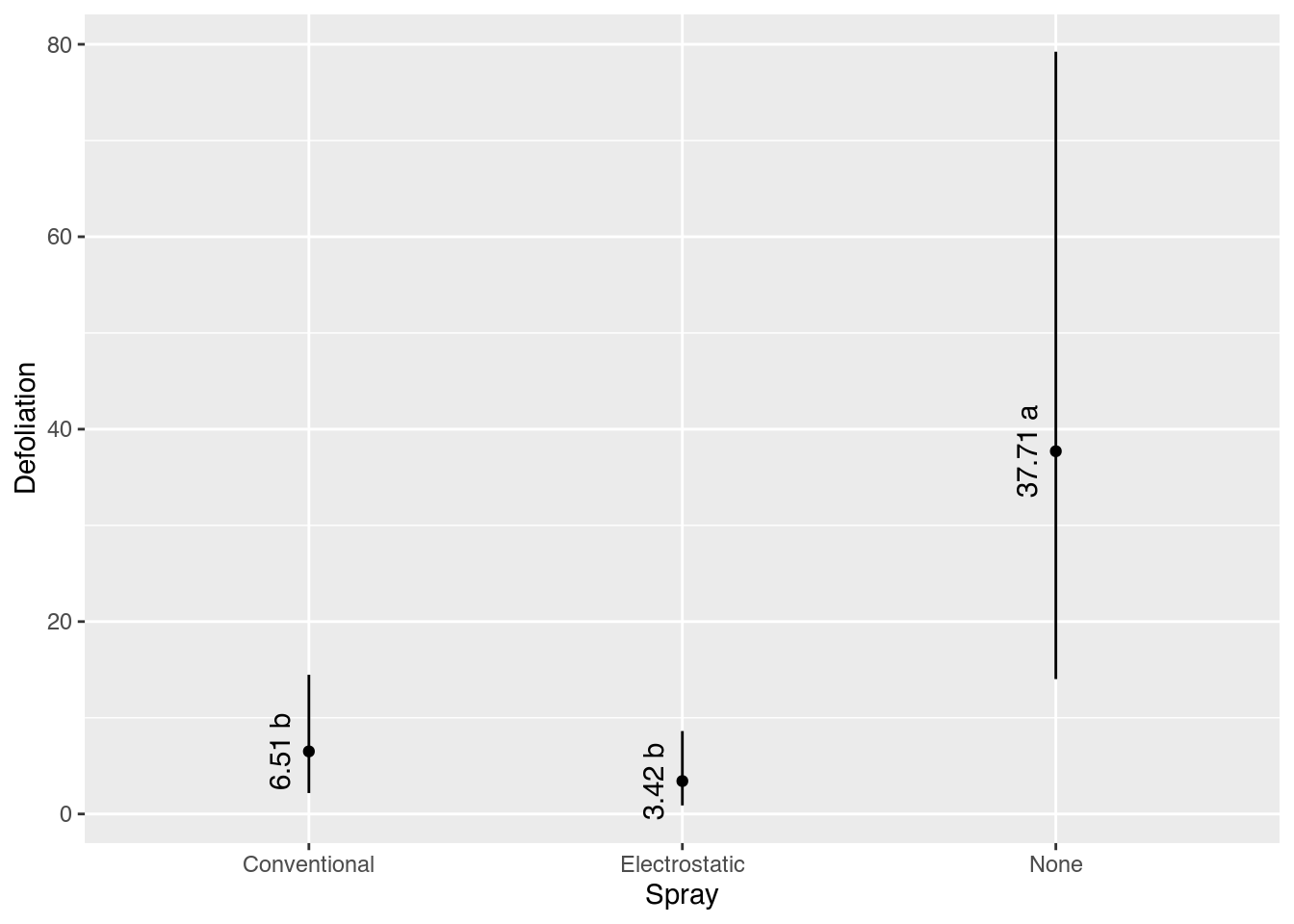
3 Severidade máxima
ggplot(data = tb,
mapping = aes(x = product,
y = sevmax)) +
facet_grid(facets = . ~ spray,
scale = "free",
space = "free") +
geom_jitter(alpha = 0.5,
width = 0.1,
height = 0) +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun = "mean") +
labs(x = "Product",
y = "Maximum severity")`geom_line()`: Each group consists of only one observation.
ℹ Do you need to adjust the group aesthetic?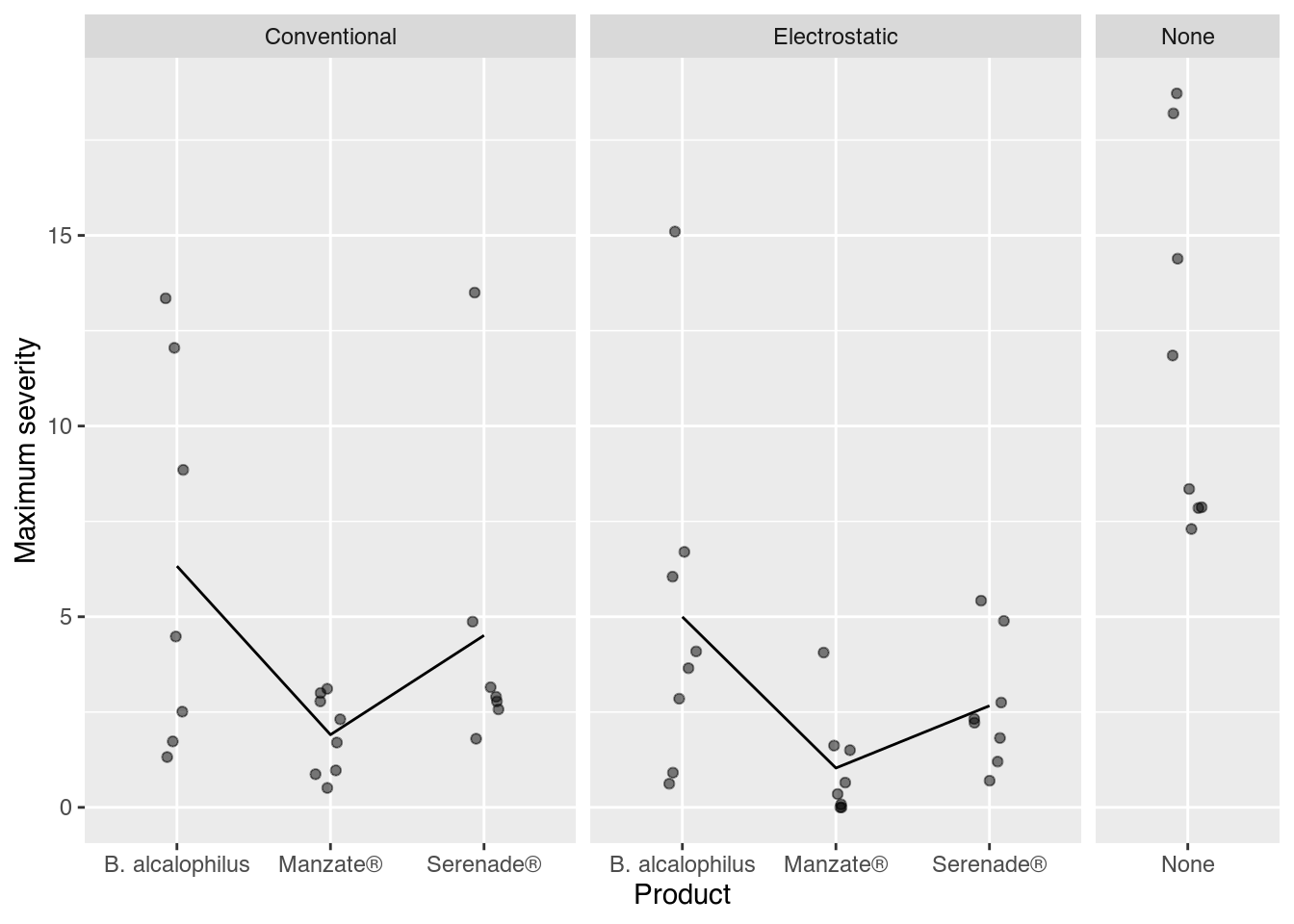
# Ajuste do modelo. -------------------------
# Ajuste do modelo via especificação por fórmula.
m0 <- lm(sevmax + 0.1 ~ block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)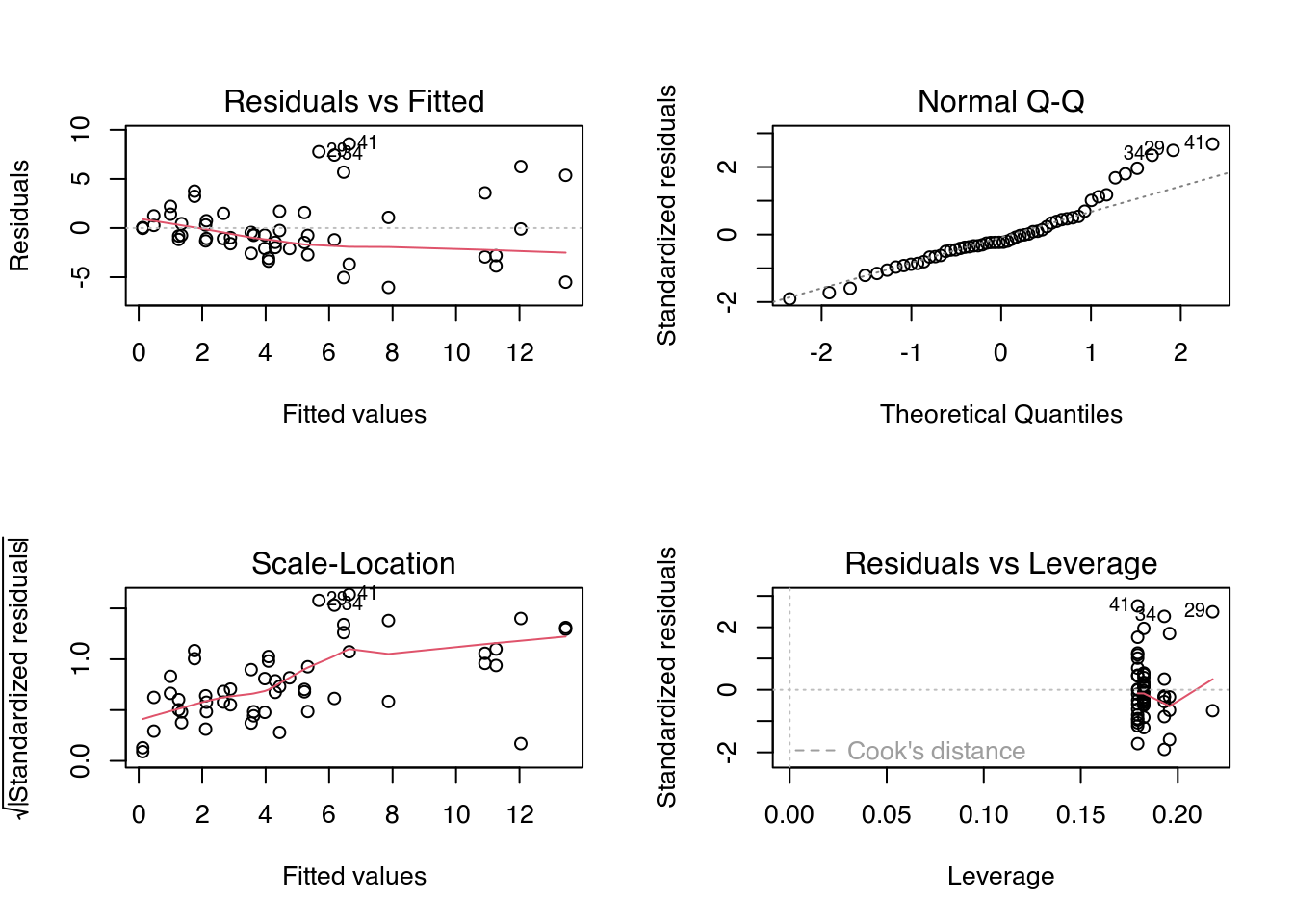
layout(1)
# Verifica necessidade de transformação.
MASS::boxcox(m0)
abline(v = 1/3, col = "red")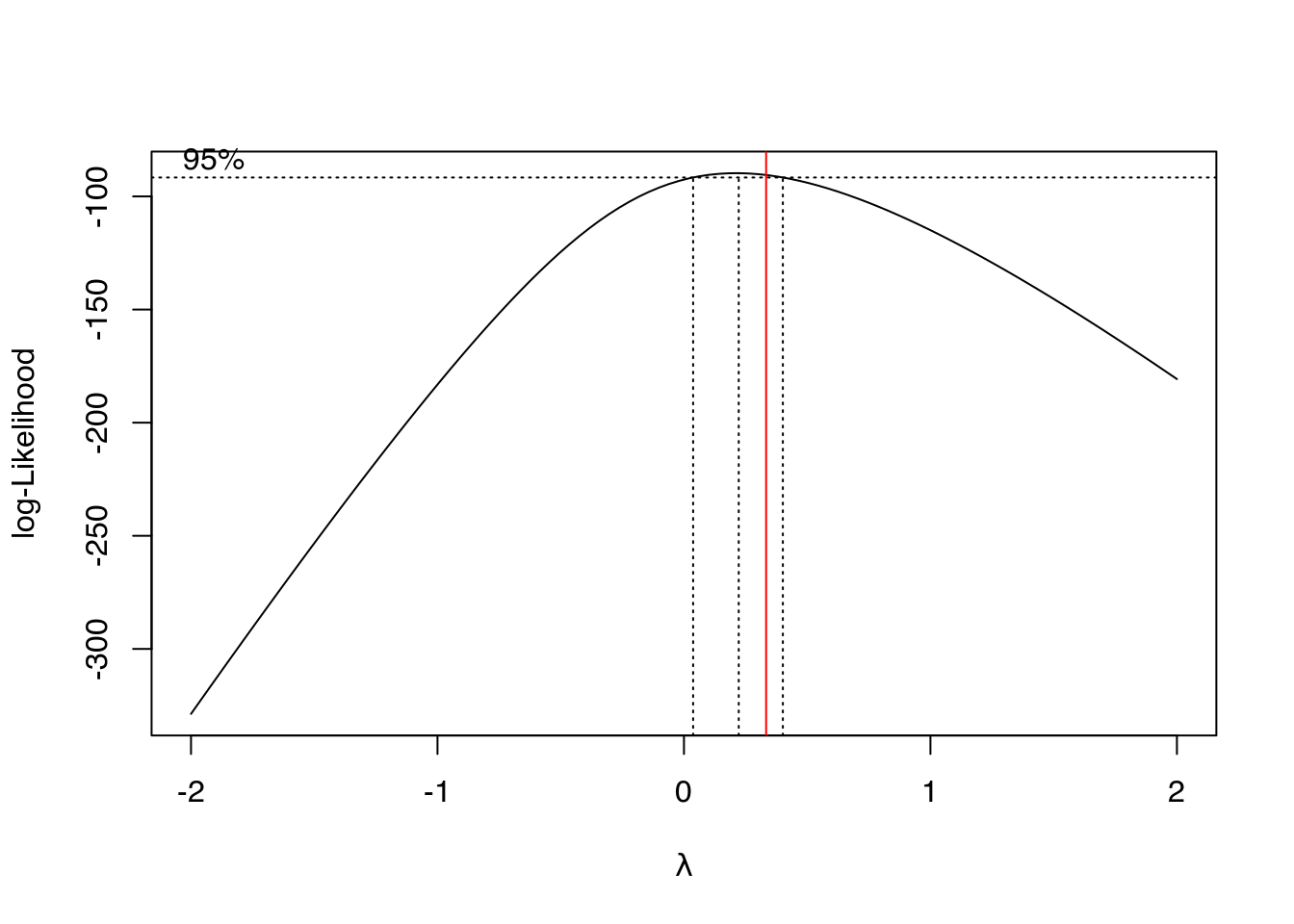
# Atualiza o modelo.
m0 <- update(m0, sevmax^(1/3) ~ .)
# Diagnóstico do modelo.
par(mfrow = c(2, 2))
plot(m0)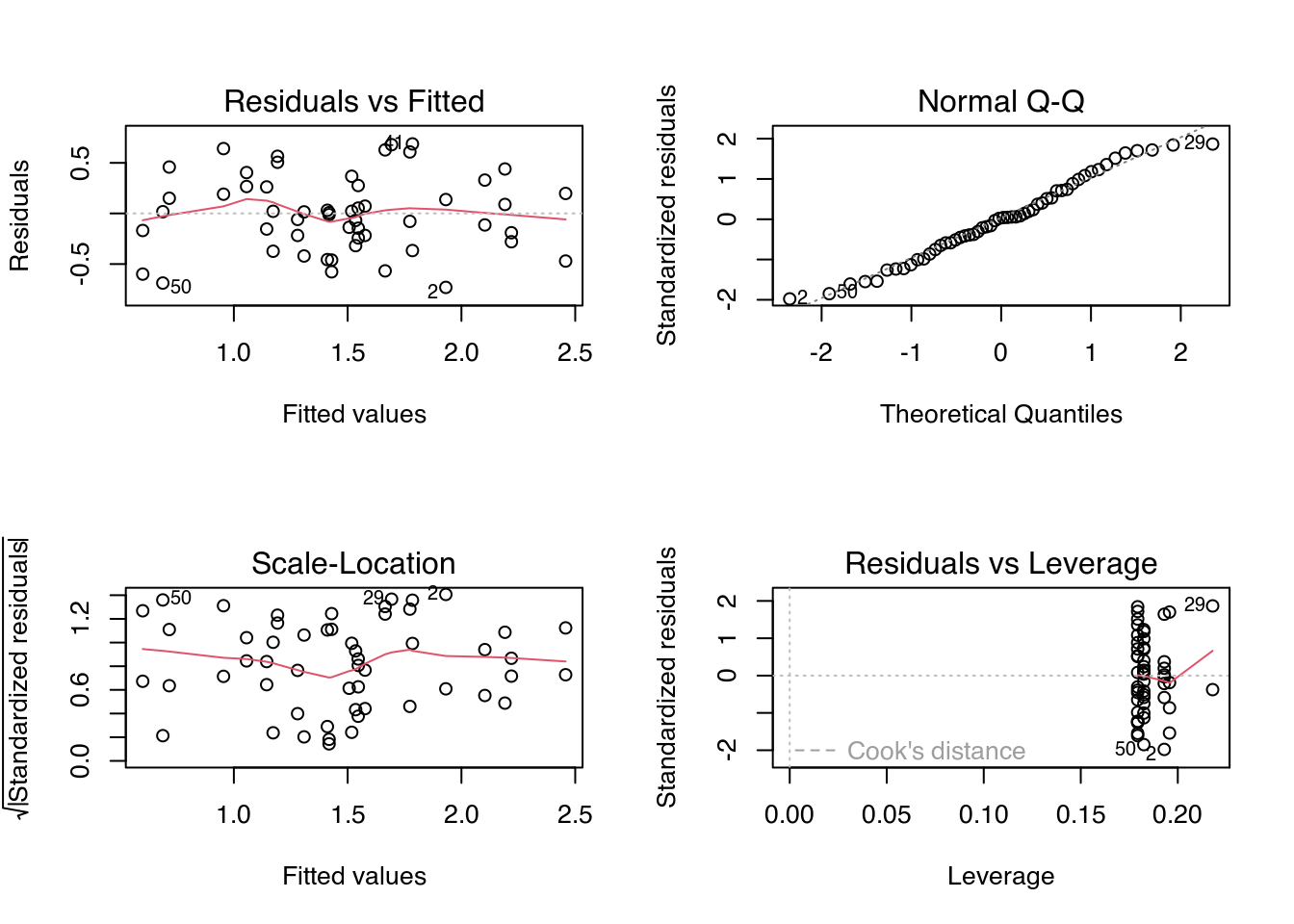
layout(1)
# Quadro de análise de variância.
anova(m0)Analysis of Variance Table
Response: sevmax^(1/3)
Df Sum Sq Mean Sq F value Pr(>F)
block 3 0.9866 0.32886 1.9435 0.13649
product 3 9.2527 3.08425 18.2269 7.816e-08 ***
spray 1 0.9030 0.90301 5.3365 0.02563 *
product:spray 2 0.2011 0.10054 0.5941 0.55641
Residuals 44 7.4454 0.16921
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Decomposição da SQ. -----------------------
# Cria a matriz do delineamento completa.
X_full <- model.matrix(~block + product * spray,
data = tb,
contrasts = list(product = "contr.helmert",
spray = "contr.helmert"))
# Modelo ajustado passando a matriz completa.
m1 <- aov(sevmax^(1/3) ~ 0 + X_full, data = tb)
# coef(m1)
# Efeitos.
ef <- list("bloc" = 2:4,
"FvsA" = 7,
"prod" = 5:6,
"spray" = 8,
"prod:spray" = 9:10)
# Desdobramento das somas de quadrados.
summary(m1, split = list("X_full" = ef)) Df Sum Sq Mean Sq F value Pr(>F)
X_full 10 128.79 12.879 76.110 < 2e-16 ***
X_full: bloc 3 0.99 0.329 1.943 0.136490
X_full: FvsA 1 5.46 5.460 32.264 9.94e-07 ***
X_full: prod 2 3.79 1.897 11.208 0.000116 ***
X_full: spray 1 0.90 0.903 5.337 0.025634 *
X_full: prod:spray 2 0.20 0.101 0.594 0.556408
Residuals 44 7.45 0.169
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Comparações de médias. --------------------
# Matriz do modelo descartadas as linhas repetidas, caso hajam.
X_un <- unique(X_full)
# Fatores do modelo, removidas as linhas repetidas também.
pts <- subset(tb, select = names(attr(X_full, "contrasts"))) |>
unique()
# Testa se os tamanhos são iguais.
stopifnot(nrow(pts) == nrow(X_un))
# Cria um objeto agregado considerando os níveis de fonte e modo.
grp <- pts |>
group_by(product, spray)
# Matriz com termos para determinar médias ajustadas para os pontos
# experimentais.
pts <- group_keys(grp)
X_ls <-
by(X_un,
INDICES = group_indices(grp),
FUN = colMeans) %>%
do.call(what = rbind)
# X_ls
# Remove efeitos não estimados.
X_ls <- X_ls[, !is.na(coef(m0))]
# Entenda os pesos, principalmente para os níveis de bloco.
MASS::fractions(t(X_ls)) 1 2 3 4 5 6 7
(Intercept) 1 1 1 1 1 1 1
block2 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
product1 -1 -1 1 1 0 0 0
product2 -1 -1 -1 -1 2 2 0
product3 -1 -1 -1 -1 -1 -1 3
spray1 -1 1 -1 1 -1 1 0
product1:spray1 1 -1 -1 1 0 0 0
product2:spray1 1 -1 1 -1 -2 2 0# Médias amostrais e ajustadas apenas para conferir.
tb |>
group_by(product, spray) |>
summarise("média amostral" = mean(sevmax^(1/3), na.rm = TRUE),
.groups = "drop") |>
bind_cols("média ajustada" = c(X_ls %*% na.omit(coef(m0))))# A tibble: 7 × 4
product spray `média amostral` `média ajustada`
<fct> <fct> <dbl> <dbl>
1 B. alcalophilus Conventional 1.72 1.72
2 B. alcalophilus Electrostatic 1.57 1.57
3 Manzate® Conventional 1.20 1.20
4 Manzate® Electrostatic 0.740 0.740
5 Serenade® Conventional 1.57 1.56
6 Serenade® Electrostatic 1.33 1.33
7 None None 2.24 2.24 # Comparação dos produtos. ------------------
# Funções lineares para média ajustada para níveis de produto.
L_prod <- by(X_ls, INDICES = pts$product, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_prod)) B. alcalophilus Manzate® Serenade® None
(Intercept) 1 1 1 1
block2 1/4 1/4 1/4 1/4
block3 1/4 1/4 1/4 1/4
block4 1/4 1/4 1/4 1/4
product1 -1 1 0 0
product2 -1 -1 2 0
product3 -1 -1 -1 3
spray1 0 0 0 0
product1:spray1 0 0 0 0
product2:spray1 0 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
# c(deviance(m2), deviance(m0))
results <-
apmc(X = L_prod,
model = m2,
focus = "product",
cld2 = TRUE) %>%
mutate(product = factor(product, levels = levels(tb$product)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
results product fit lwr upr cld
1 B. alcalophilus 4.4356238 2.9122653 6.414692 b
2 Manzate® 0.9062024 0.4397456 1.622071 c
3 Serenade® 3.0179078 1.8613726 4.575254 b
4 None 11.2827702 7.4122330 16.309447 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = product, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Product",
y = "Maximum severity")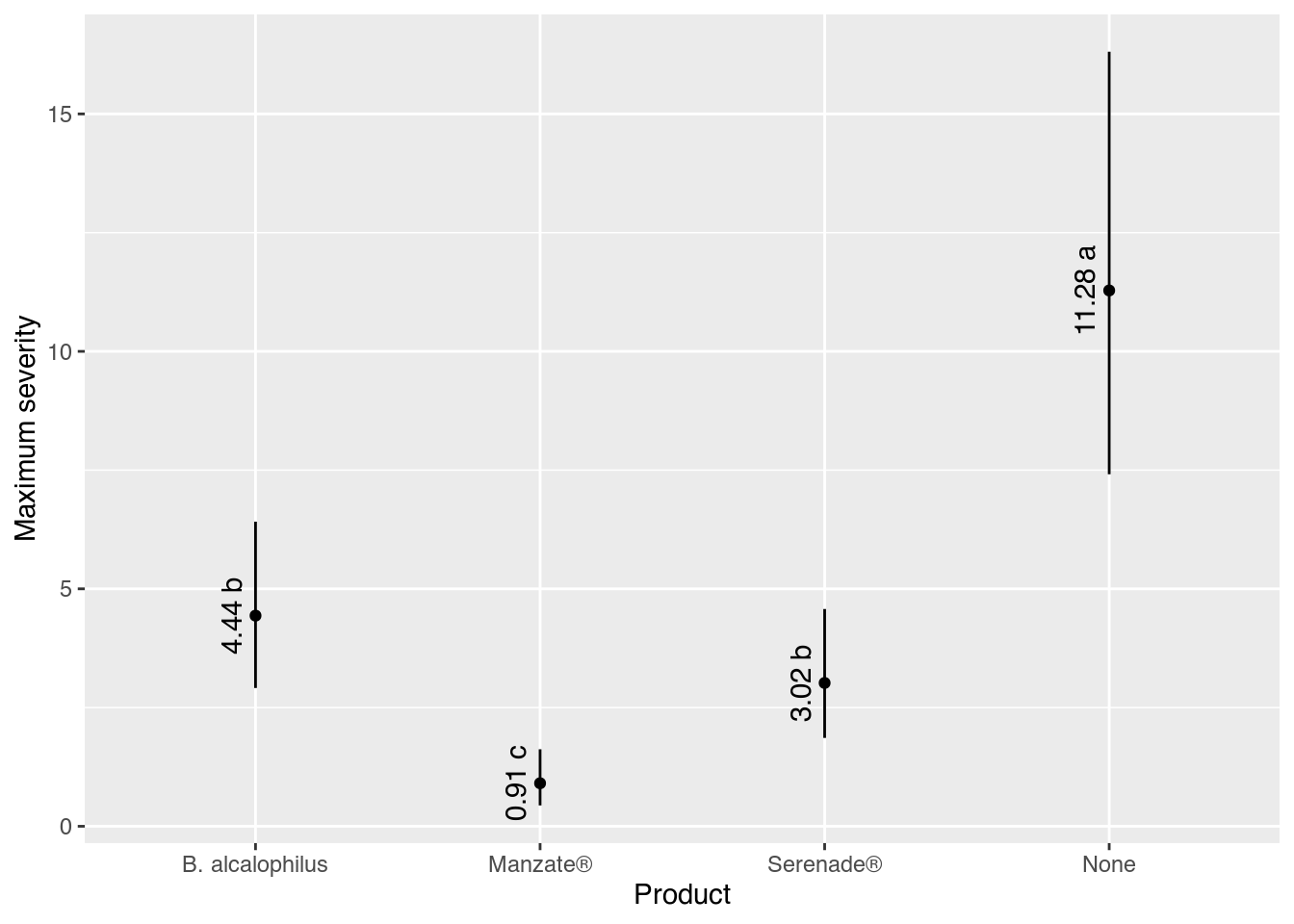
# Comparação dos sprays. --------------------
# Funções lineares para média ajustada para níveis de produto.
L_spray <- by(X_ls, INDICES = pts$spray, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_spray)) Conventional Electrostatic None
(Intercept) 1 1 1
block2 1/4 1/4 1/4
block3 1/4 1/4 1/4
block4 1/4 1/4 1/4
product1 0 0 0
product2 0 0 0
product3 -1 -1 3
spray1 -1 1 0
product1:spray1 0 0 0
product2:spray1 0 0 0 X <- model.matrix(m0)[, colnames(X_ls)]
# colnames(X)
# Atualiza com a matriz sem particionar.
m2 <- update(m0, . ~0 + X) |>
suppressWarnings()
results <-
apmc(X = L_spray,
model = m2,
focus = "spray",
cld2 = TRUE) %>%
mutate(spray = factor(spray, levels = levels(tb$spray)),
cld = ordered_cld(cld, fit))
# Volta para a escala original.
results <-
results |>
mutate_at(c("fit", "lwr", "upr"), ~ .^3)
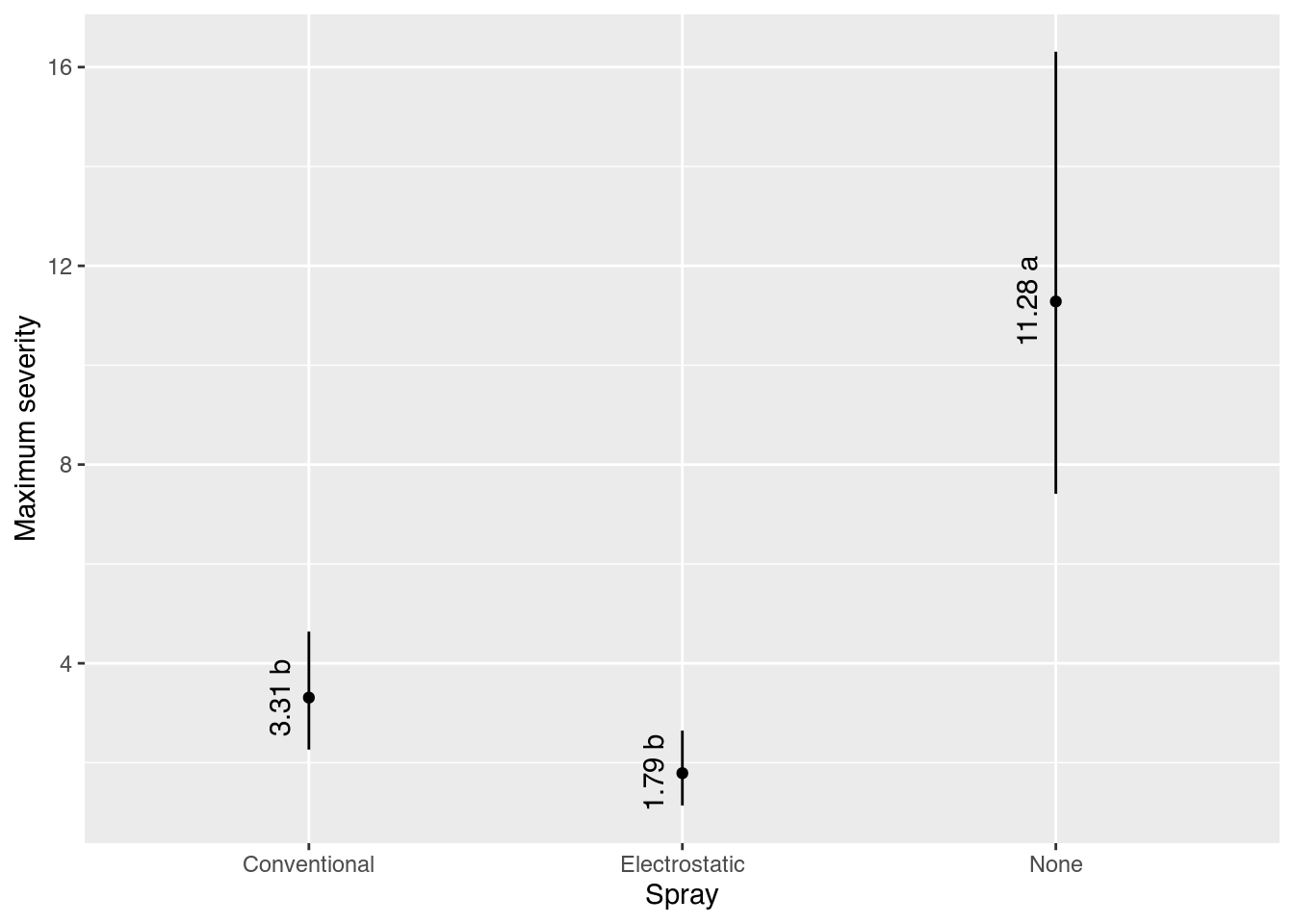
results spray fit lwr upr cld
1 Conventional 3.309423 2.262501 4.637824 b
2 Electrostatic 1.787824 1.139416 2.644773 b
3 None 11.282770 7.412233 16.309447 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = spray, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Spray",
y = "Maximum severity")