29 Agrupando comandos, execução condicional, controle de fluxo, "loops" e a "família" *apply
29.1 Agrupando comandos
O R é uma linguagem que interpreta expressões, o que implica que o único tipo de comando usado é
uma expressão ou função que executa o processamento da requisição e retorna algum resultado. Nesta
sessão vamos alguns formatos para facilitar/agilizar o uso de comandos.
É possível atribuir os mesmos valores a vários objetos de uma só vez utilizando atribuições
múltiplas de valores.
> x <- y <- z <- numeric(5)
> x
Um grupo de comandos podem ser agrupado com "{ }" e separados por ";" para digitação em
uma mesma linha. Em certas situações, como no "prompt"do R as chaves são opcionais.
> {
+ x <- 1:3
+ y <- x + 4
+ z <- y/x
+ }
> x <- 1:3
> y <- x + 4
> z <- y/x
> x
[1] 5.000000 3.000000 2.333333
29.2 Execução condicional
Execuções condicionais são controladas por funções especiais que verificam se uma condição é
satisfeita para permitir a execução de um comando. As seguintes funções e operadores podem ser
usadas para controlar execução condicional.
- if() (opcionalmente) acompanhado de else
- &, ∥, && e ∥∥
- ifelse()
- switch()
A estrutura if() else é comumente usada, em especial dentro de funções. Quando aplicada
diretamente na linha de comando, é uma prática recomendada colocar chaves marcando o início e
fim dos comandos de execução condicional. Quando a expressão que segue o if() e/ou
else tem uma única linha ela pode ser escrita diretamente, entretando, caso sigam-se
mais de duas linhas deve-se novamente usar chaves, agora também depois destes de forma
que todos os comandos da execução condicional fiquem contidos na chave, caso contrário
apenas a primeira linha será considerada para execução condicional e todas as demais são
processadas normalmente. Inspecione os comandos a seguir que ilustram os diferentes usos.
> x <- 10
> y <- 15
> {
+ if (x > 8)
+ z <- 2 * x
+ }
> z
> rm(x, y, z)
> x <- 10
> y <- 15
> {
+ if (x > 12)
+ z <- 2 * x
+ else z <- 5 * x
+ }
> z
> rm(x, y, z)
> x <- 10
> y <- 15
> {
+ if (x > 8) {
+ z <- 2 * x
+ w <- z + y
+ }
+ else {
+ z <- 5 * x
+ w <- z - y
+ }
+ }
> z
> rm(x, y, z, w)
> x <- 10
> y <- 15
> {
+ if (x > 8)
+ z <- 2 * x
+ w <- z + y
+ if (x <= 8)
+ z <- 5 * x
+ w <- z - y
+ }
> z
Um comando útil para manipulação de dados é o split() que permite separa dados por grupos.
Por exemplo considere o conjunto de dados codemtcars, que possui várias variáveis relacionadas a
características de veículos. Entre as variáveis estão as que indicam o consumo (mpg - miles per
gallon) e o tipo de câmbio, manual ou automático (am). Para separar os dados da variável mpg para
cada tipo de câmbio, podemos usar:
> data(mtcars)
> with(mtcars, split(mpg, am))
$‘0‘
[1] 21.4 18.7 18.1 14.3 24.4 22.8 19.2 17.8 16.4 17.3 15.2 10.4 10.4 14.7 21.5
[16] 15.5 15.2 13.3 19.2
$‘1‘
[1] 21.0 21.0 22.8 32.4 30.4 33.9 27.3 26.0 30.4 15.8 19.7 15.0 21.4
Outro comando com funcionalidade similar é agregate().
29.3 Controle de fluxo
O controle de fluxo no R é implementado pelas funções for(), while() e repeat(). A escolha de
qual usar vai depender do contexto e objetivo do código e em geral não existe solução única, sendo
que uma mesma tarefa pode ser feita por uma ou outra.
Apenas para ilustração considere o seguinte exemplo resolvido de três formas diferentes com cada
uma destas funções:
Dado um valor de n gerar amostrar de tamanho 1, 2,…,n e para calcule a média de cada amostra, com
3 casas decimais.
Primeiro vamos implementar uma solução usando for().
> f1 <- function(n) {
+ medias <- numeric(n)
+ for (i in 1:n) {
+ am <- rnorm(i)
+ medias[i] <- round(mean(am), dig = 3)
+ }
+ return(medias)
+ }
> set.seed(283)
> f1(10)
[1] 1.007 -0.063 -0.392 1.546 0.341 -0.514 -0.086 -0.224 0.137 0.138
Agora vamos executar a mesma tarefa com while()
> f2 <- function(n) {
+ medias <- numeric(n)
+ i <- 1
+ while (i <= n) {
+ am <- rnorm(i)
+ medias[i] <- round(mean(am), dig = 3)
+ i <- i + 1
+ }
+ return(medias)
+ }
> set.seed(283)
> f2(10)
[1] 1.007 -0.063 -0.392 1.546 0.341 -0.514 -0.086 -0.224 0.137 0.138
E finalmente a mesma tarefa com repeat()
> f3 <- function(n) {
+ medias <- numeric(n)
+ i <- 1
+ repeat {
+ am <- rnorm(i)
+ medias[i] <- round(mean(am), dig = 3)
+ if (i == n)
+ break
+ i <- i + 1
+ }
+ return(medias)
+ }
> set.seed(283)
> f3(10)
[1] 1.007 -0.063 -0.392 1.546 0.341 -0.514 -0.086 -0.224 0.137 0.138
NOTA: as soluções acima são apenas ilustrativas e não representam a forma mais eficiente de
efetuar tal operação o R. Na verdade, para este tipo de cálculo recomenda-se o uso de funções do tipo
*apply que veremos no restante desta sessão.
29.4 Alguns comentários adicionais
Nas soluções acima as amostras foram usadas para calcular as médias e depois descartadas. Suponha
agora que queremos preservar e retornar também os dados simulados. Para ilustrar vamos mostrar
como fazer isto modificando um pouco a primeira função.
> f1a <- function(n) {
+ res <- list()
+ res$amostras <- list()
+ res$medias <- numeric(n)
+ for (i in 1:n) {
+ res$amostras[[i]] <- rnorm(i)
+ res$medias[i] <- round(mean(res$amostras[[i]]), dig = 3)
+ }
+ return(res)
+ }
> set.seed(283)
> ap <- f1a(4)
> names(ap)
$amostras
$amostras[[1]]
[1] 1.00687
$amostras[[2]]
[1] 0.2003886 -0.3257288
$amostras[[3]]
[1] 0.4913491 -1.0009700 -0.6665789
$amostras[[4]]
[1] 2.035963 1.174572 1.214059 1.761383
$medias
[1] 1.007 -0.063 -0.392 1.546
Vamos agora ver uma outra modificação. Nas funções acima geravamos amostras com tamanhos
sequênciais com incremento de 1 elemento no tamanho da amostra. A função a seguir mostra como
gerar amostras de tamanhos especificados pelo usuário e para isto toma como argumento um vetor de
tamanhos de amostra.
> f5 <- function(ns) {
+ medias <- numeric(length(ns))
+ j <- 1
+ for (i in ns) {
+ am <- rnorm(i)
+ medias[j] <- round(mean(am), dig = 3)
+ j <- j + 1
+ }
+ return(medias)
+ }
> set.seed(231)
> f5(c(2, 5, 8, 10))
[1] -1.422 -0.177 0.056 0.158
29.5 Evitando "loops" — a "família" *apply
O R é uma linguagem vetorial e "loops"podem e devem ser substituídos por outras formas de
cálculo sempre que possível. Usualmente usamos as funções apply(), sapply(), tapply() e
lapply() para implementar cálculos de forma mais eficiente. Vejamos algums exemplos.
- apply() para uso em matrizes, arrays ou data-frames
- tapply() para uso em vetores, sempre retornando uma lista
- sapply() para uso em vetores, simplificando a estrutura de dados do resultado se possível
(para vetor ou matriz)
- mapply() para uso em vetores, versão multivariada de sapply()
- lapply() para ser aplicado em listas
-
1.
- Seja o problema mencionado no início desta sessão de gerar amostras de tamanhos sequenciais e
calcular a média para cada uma delas. Uma alternativa aos códigos apresentados seria:
> set.seed(283)
> sapply(1:10, function(x) round(mean(rnorm(x)), dig = 3))
[1] 1.007 -0.063 -0.392 1.546 0.341 -0.514 -0.086 -0.224 0.137 0.138
-
2.
- Considere agora a modificação mencionado anteriormente de calcular médias de amostras com
tamanho fornecidos pelo usuário
> vec <- c(2, 5, 8, 10)
> f6 <- function(n) round(mean(rnorm(n)), dig = 3)
> set.seed(231)
> sapply(vec, f6)
[1] -1.422 -0.177 0.056 0.158
-
3.
- No próximo exemplo consideramos uma função que simula dados e calcula medidas de posição e
dispersão associadas utilizando para cada uma delas duas medidas alternativas. Inicialmente
definimos a função:
> proc <- function(...) {
+ x <- rnorm(500)
+ modA <- list(pos = mean(x), disp = sd(x))
+ modB <- list(pos = mean(x, trim = 0.1), disp = mad(x))
+ return(list(A = modA, B = modB))
+ }
Agora vamos rodar a função 10 vezes.
> set.seed(126)
> res <- lapply(1:10, proc)
O resultado está armazanado no objeto res, que neste caso é uma lista. Agora vamos
extrair desta lista as médias aritméticas e depois ambas, média aritmética e aparada:
> mediaA <- function(x) x$A$pos
> mA <- sapply(res, mediaA)
> mediaAB <- function(x) c(x$A$pos, x$B$pos)
> mAB <- sapply(res, mediaAB)
> rownames(mAB) <- paste("modelo", LETTERS[1:2], sep = "")
> mAB
[,1] [,2] [,3] [,4] [,5] [,6]
modeloA 0.02725767 -0.01017973 0.0958355 0.02058979 0.04582751 0.07898205
modeloB 0.01706928 -0.02781770 0.1023454 0.02210935 0.06210404 0.05914628
[,7] [,8] [,9] [,10]
modeloA 0.06122656 -0.05981805 0.006781871 -0.02798788
modeloB 0.04085053 -0.05680834 -0.020411456 -0.02029610
Os comandos acima podem ser reescritos em versões simplificadas:
> mA <- sapply(res, function(x) x$A$pos)
> mA
[1] 0.027257675 -0.010179733 0.095835502 0.020589788 0.045827513
[6] 0.078982050 0.061226561 -0.059818054 0.006781871 -0.027987878
> mAB <- sapply(res, function(x) sapply(x, function(y) y$pos))
> mAB
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
A 0.02725767 -0.01017973 0.0958355 0.02058979 0.04582751 0.07898205 0.06122656
B 0.01706928 -0.02781770 0.1023454 0.02210935 0.06210404 0.05914628 0.04085053
[,8] [,9] [,10]
A -0.05981805 0.006781871 -0.02798788
B -0.05680834 -0.020411456 -0.02029610
E para obter as médias das médias de cada medida:
A B
0.02385153 0.01782913
-
4.
- A função tapply() pode ser usada para calcular o resultado de uma operação sobre dados, para
cada um dos níveis de uma segunda variável No primeiro exemplo consideramos novamente o
conjunto de dados mtcars mencionado anteriormente. Os comandos abaixo calculam
média, variância e coeficinte de variação do consumo para cada tipo de cambio.
> with(mtcars, tapply(mpg, am, mean))
> with(mtcars, tapply(mpg, am, var))
> with(mtcars, tapply(mpg, am, function(x) 100 * sd(x)/mean(x)))
Vejamos ainda um outro exemplo onde definimos 50 dados divididos em 5 grupos.
> x <- rnorm(50, mean = 50, sd = 10)
> y <- rep(LETTERS[1:5], each = 10)
> x
[1] 55.66788 43.71391 42.78483 50.28745 40.77170 62.06800 60.53166 51.90432
[9] 56.41214 65.46560 35.99390 42.67566 40.26776 47.61359 57.92209 60.69673
[17] 48.80234 44.29422 44.48886 39.02277 60.93054 32.73959 39.37867 56.89312
[25] 37.06637 61.45986 44.66166 50.60778 37.78913 39.13208 48.53931 43.29661
[33] 66.03602 65.55652 58.05864 55.21829 45.90542 45.01864 37.73984 38.00313
[41] 34.85114 34.24760 65.07629 49.01286 62.37572 38.36997 57.93003 39.72861
[49] 66.62899 45.37572
[1] "A" "A" "A" "A" "A" "A" "A" "A" "A" "A" "B" "B" "B" "B" "B" "B" "B" "B" "B"
[20] "B" "C" "C" "C" "C" "C" "C" "C" "C" "C" "C" "D" "D" "D" "D" "D" "D" "D" "D"
[39] "D" "D" "E" "E" "E" "E" "E" "E" "E" "E" "E" "E"
> gM <- tapply(x, y, mean)
> gM
A B C D E
52.96075 46.17779 46.06588 50.33724 49.35969
> gCV <- tapply(x, y, function(z) 100 * sd(z)/mean(z))
> gCV
A B C D E
16.19106 17.17599 23.08328 20.65681 25.77284
Para organizar os dados em um data-frame:
> xy <- data.frame(x = rnorm(50, mean = 50, sd = 10), y = rep(LETTERS[1:5],
+ each = 10))
> gM <- with(xy, tapply(x, y, mean))
> gM
A B C D E
49.91571 51.03091 45.26204 47.45439 47.25661
> gCV <- with(xy, tapply(x, y, function(z) 100 * sd(z)/mean(z)))
> gCV
A B C D E
16.13100 11.97707 17.35279 18.67300 16.03077
-
5.
- Considere gerarmos uma matrix 1000 × 300 representando 1000 amostras de tamanho 300. O
que desejamos é calcular a média de cada uma das amostras Os códigos a seguir mostras três
formas alternativas de fazer isto. Encapsulamos os comandos com a função system.time() que
compara os tempos de execução.
> x <- matrix(rnorm(1000 * 300), nc = 300)
> system.time({
+ f <- function(x) {
+ mx <- numeric(1000)
+ for (i in 1:1000) mx[i] <- mean(x[i, ])
+ mx
+ }
+ mx <- f(x)
+ })
user system elapsed
0.076 0.008 0.085
> system.time(mx <- apply(x, 1, mean))
user system elapsed
0.100 0.000 0.103
> system.time(mx <- rowMeans(x))
user system elapsed
0.004 0.000 0.003
A função rowMeans() é substancialmente mais eficiente (menos tempo de execução. Outras
funções simulares são colMeans(), rowSums() e colSums().
-
6.
- Considere o seguinte problema:
Sejam li e ls vetores com os limites superiores e inferiores definindo intervalos. Inicialmente
vamos simular estes valores.
> li <- round(rnorm(500, m = 70, sd = 10))
> ls <- li + rpois(li, lam = 5)
O que queremos montar um vetor com os valores únicos que definem estes intervalos, e testar a
pertinência de cada elemento a cada um dos intervalos. Ao final teremos uma matrix
indicando, para cada elemento do vetor de valores únicos, a pertinência a cada intervalo.
Inicialmente vamos fazer um código usando "loops"guardando os resultados no objeto B.
> system.time({
+ aux <- sort(c(li, ls))
+ m <- length(table(aux))
+ all <- rep(min(aux), m)
+ for (j in 1:(m - 1)) {
+ all[j + 1] <- min(aux[aux > all[j]])
+ }
+ n <- length(li)
+ aij <- matrix(0, nrow = n, ncol = m)
+ for (i in 1:n) {
+ for (j in 1:m) {
+ aij[i, j] <- ifelse(all[j] >= li[i] & all[j] <= ls[i],
+ 1, 0)
+ }
+ B <- aij
+ }
+ })
user system elapsed
2.536 0.032 2.790
Agora, usando a estrutura vetorial da linguagem R vamos reimplementar este código de
maneira mais eficiente e adequada para a linguagem, usando sapply(), guardando os
resultados no objeto A. Ao final usamos identical() para testar se os resultados
numéricos são exatamente os mesmos. Note a diferença nos tempos de execução.
> system.time({
+ all <- sort(unique(c(li, ls)))
+ interv1 <- function(x, inf, sup) ifelse(x >= inf & x <= sup,
+ 1, 0)
+ A <- sapply(all, interv1, inf = li, sup = ls)
+ })
user system elapsed
0.032 0.004 0.037
-
7.
- Considere agora uma extensão do problema anterior. Queremos montar o vetor com os valores
únicos que definem estes intervalos como no caso anterior, e depois usar este vetor montar
intervalos com pares de elementos consecutivos deste vetor e testar se cada um destes intervalos
está contido em cada um dos intervalos originais. O resultado final é uma matrix indicando para
cada intervalo obtido desta forma a sua pertinência a cada um dos intervalos originais. Da
mesma forma que no caso anterior implementamos com um "loop"e depois usando a estrutura
vetorial da linguagem, e testando a igualdade dos resultados com identical().
> li <- round(rnorm(500, m = 70, sd = 10))
> ls <- li + rpois(li, lam = 5)
> system.time({
+ aux <- sort(c(li, ls))
+ m <- length(table(aux))
+ all <- rep(min(aux), m)
+ for (j in 1:(m - 1)) {
+ all[j + 1] <- min(aux[aux > all[j]])
+ }
+ n <- length(li)
+ aij <- matrix(0, nrow = n, ncol = m - 1)
+ for (i in 1:n) {
+ for (j in 1:m - 1) {
+ aij[i, j] <- ifelse(all[j] >= li[i] & all[j + 1] <=
+ ls[i], 1, 0)
+ }
+ B <- aij
+ }
+ })
user system elapsed
2.892 0.040 3.150
> system.time({
+ all <- sort(unique(c(li, ls)))
+ all12 <- cbind(all[-length(all)], all[-1])
+ interv1 <- function(x, inf, sup) ifelse(x[1] >= inf & x[2] <=
+ sup, 1, 0)
+ A <- apply(all12, 1, interv1, inf = li, sup = ls)
+ })
user system elapsed
0.032 0.000 0.050
Uso da família *apply – outros exemplos
Os exemplos a seguir foram retirados de mensagens enviada à lista R_STAT.
-
1.
- adapdato de mensagem enviada por Silvano C Costa
Tenho uma pergunta onde pode haver mais de uma resposta, por exemplo:
Q1 - Qual Esporte você pratica:
1.()Futebol 2.()Volei 3.() Natação 4.()Atletismo
Q2 - Sexo:
1.Masculino() 2.Feminino()
Então teria os dados dessa forma:
Q1.1 Q1.2 Q1.3 Q1.4 Q2
1 0 1 0 1 => Homem Praticante de Futebol,Natacao
0 1 1 0 2 => Mulher praticante de Volei e Natação
0 0 0 1 2 => Mulher praticante de Atletismo
Gostaria de criar uma tabela cruzada entre essas variáveis:
M F
Futebol 21 10
Natação 13 20
Volei 5 2
Atletismo 10 10
Para mostrar como obter a solução, como não temos o questionário aqui vamos primeiro simular
dados deste tipo como se tivéssemos 75 questionários.
> esportes <- as.data.frame(matrix(sample(c(0, 1), 300, rep = TRUE),
+ nc = 4))
> names(esportes) <- c("Futebol", "Natacao", "Volei", "Atletismo")
> esportes$S <- sample(c("M", "F"), 75, rep = TRUE)
> dim(esportes)
Futebol Natacao Volei Atletismo S
1 1 1 0 0 F
2 0 0 1 0 F
3 1 1 1 0 M
4 1 0 1 0 F
5 1 1 1 0 F
6 0 1 1 1 F
Solução 1: Para cada esporte podemos contar os praticantes de cada sexo em cada esporte,
separadamente utilizando table() e verificando a segunda linha da tablea a seguir.
> with(esportes, table(Futebol, S))
S
Futebol F M
0 18 19
1 19 19
Desta forma, podemos obter a tabela desejada combinando os resultados de tabelas para cada
esporte.
> with(esportes, rbind(table(Futebol, S)[2, ], table(Natacao, S)[2,
+ ], table(Volei, S)[2, ], table(Atletismo, S)[2, ]))
F M
[1,] 19 19
[2,] 19 22
[3,] 24 20
[4,] 20 21
Solução 2: alternativamente, podemos usar sapply() para tomar cada esporte e, como os
dados são codificados em 0/1, usar tapply() para somar os praticantes (1) de cada sexo.
> sapply(esportes[, 1:4], function(x) tapply(x, esportes$S, sum))
Futebol Natacao Volei Atletismo
F 19 19 24 20
M 19 22 20 21
-
2.
- Adaptado de mensagem enviada por André
Queria fazer uma amostragem e tirar as informações. Tenho duas amostras.
Aplico o teste t e tenho um P-valor.
a <-c(1,2,4,5,3,4,5,6,6,7,2)
b <-c(5,3,4,5,3,4,5,3,4,3,5)
Eu gostaria de juntar estes dois vetores num mesmo vetor e fazer 1000
reamostragens neste vetor de tamanho do vetor a e com reposição.
e <- c(5,3,4,5,3,4,5,3,4,3,5,1,2,4,5,3,4,5,6,6,7,2)
Depois eu queria ver aplicar o teste t(nas amostras) e ver como se
comportam estes p-valores.
Inicialmente vamos entrar com os dados e obter o teste-t.
> a <- c(1, 2, 4, 5, 3, 4, 5, 6, 6, 7, 2)
> b <- c(5, 3, 4, 5, 3, 4, 5, 3, 4, 3, 5)
> tt.ab <- t.test(a, b)
> tt.ab
Welch Two Sample t-test
data: a and b
t = 0.1423, df = 14.14, p-value = 0.8889
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-1.278239 1.460057
sample estimates:
mean of x mean of y
4.090909 4.000000
Agora obtemos as 1000 reamostras deste vetor cada uma com 11 × 2 = 22 valores utilizando
sample(). As reamostras serão arranjadas num array de dimensão 11 × 2 × 1000.
> e <- c(5, 3, 4, 5, 3, 4, 5, 3, 4, 3, 5, 1, 2, 4, 5, 3, 4, 5,
+ 6, 6, 7, 2)
> reamostras <- array(sample(e, length(e) * 1000, rep = T), dim = c(length(e)/2,
+ 2, 1000))
Portanto cada elemento da terceira dimensão corresponde a uma reamostra. Para fazer os
testes-t nas 1000 reamostras utilizamos apply() que vai gerar uma lista de 100 elementos com
resultados dos testes.
> TT <- apply(reamostras, 3, function(x) t.test(x[, 1], x[, 2]))
Para ver o resultado do teste em uma das amostras selecionamos um elemento da lista.
Welch Two Sample t-test
data: x[, 1] and x[, 2]
t = -1.8448, df = 19.886, p-value = 0.08001
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-1.7436506 0.1072869
sample estimates:
mean of x mean of y
4.000000 4.818182
Finalmente pode-se extrair uma quantidade de interesse dos resultados, como no exemplo a
seguir, extraímos os p-valores.
[1] "statistic" "parameter" "p.value" "conf.int" "estimate"
[6] "null.value" "alternative" "method" "data.name"
> pvals <- sapply(TT, function(x) x$p.value)
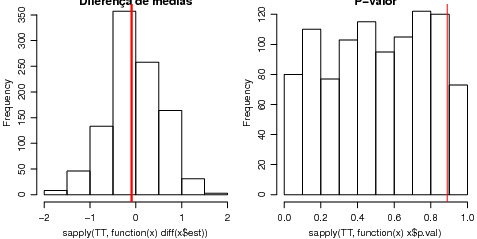
Os gráficos no Figura 2 mostram valores obtidos an amostra, comparados aos valores obtidos na
reamostragem. À esquerda é mostrado a estimativa da diferença de médias, e à direita os
p-valores.
> hist(sapply(TT, function(x) diff(x$est)), main = "Diferença de médias")
> abline(v = diff(tt.ab$est), col = 2, lwd = 3)
> hist(sapply(TT, function(x) x$p.val), main = "P-valor")
> abline(v = tt.ab$p.val, col = 2)
OBS: note que estão sendo usadas as opçõees default do teste-t para comparação de duas
amostras dadas em t.test() (bilateral, não pareado, variâncias diferentes, etc). Para alterar
algum argumento basta acrescentar o argumento desejados na chamada de apply().
> TT <- apply(reamostras, 3, function(x, ...) t.test(x[, 1], x[,
+ 2], ...), var.equal = TRUE)
29.6 Extensões da família *apply
As funções da família *apply tem o propósito geral de aplicar uma função repetidamente a diversas
partes de um conjunto de dados. Além das mencionadas acima várias outras tem sido implementadas.
Notadamente se destacam as do pacote plyr (a_ply, d_ply, l_ply, r_ply, dentre váris
outras).
Por exemplo considere a função l_ply que aplica uma função em elementos uma lista porém em
situações onde estamos interessados apenas em um efeito colateral (gráfico, arquivo gravado em disco,
etc) de função e não no resultado que ela retorna. Como exemplo considere com conjunto de dados
trees, uma data-frame que possui três colunas com diâmetros, alturas e volumes de árvores. Supunha
que desejamos fazer o histograma das três variáveis, e então uma possível forma seria utlizar o
comando lapply().
> data(trees)
> par(mfrow = c(1, 3))
> lapply(trees, hist)
Neste caso, além de produzir os gráficos as resultados dos cálculos do histograma são retornados na
tela, o que pode ser indesejável e/ou desnecessário. A função l_ply() produz o mesmo
resultado gráfico porém sem que os resultados dos cálculos do histograma sejam retornados.
> require(plyr)
> l_ply(trees, hist)
Para mais informações sobre o pacote plyr consulte as informações e documentaçÕes em
http://had.co.nz/plyr/.