O problema que ilustra este tópico consiste em encontrar valores "padrão"(ou de referência) para a teores de elementos químicos Para isto, amostras de referência supostamente de teores iguais foram enviadas a diferentes laboratórios nos quais determinações de teores foram feitas com replicações.
Como exemplo, considere os dados dos teores medidos de um único elemento mostrados a seguir e que podem ser obtidos em http://www.leg.ufpr.br/~paulojus/dados/MgO.xls
Pode-se identificar duas fontes de variação nos valores medidos, uma devido à variabilidade entre laboratórios e outra devida à variabilidade das replicações feitas nos laboratórios. O objetivo é encontrar um valor "característico"para as amostras, que seria dado por alguma "média"adequada, associada a uma medida de variabilidade desta média, por exemplo dada por um intervalo de confiança. Além disto deseja-se estimar os "componentes de variância", isto é, medidas da variabilidade entre e dentro de laboratórios.
Nos resultados a seguir ajustamos um modelo ajustado com a função lme() do pacote nlme.
O summary() mostra um resumo dos resultados mais importantes do ajuste do modelo incluindo as estimativas da média (Fixed Effects: Intercept) e dos desvios padrão entre laboratórios (Random Effects: Intercept) e das replicatas (Random Effects: Residual).
O intervalo de confiança para média e as estimativas das variâncias e desvios padrões podem ser obtidos como mostrado a seguir.
Os resultados mostrados anteriormente devem ser vistos apenas como uma ilustração dos comandos básicos para obtenção dos resultados. Entretanto, não deve-se tomar os resultados obtidos como corretos ou definitivos pois uma análise criteriosa deve verificar anomalias dos dados e adequação a pressupostos do modelo.
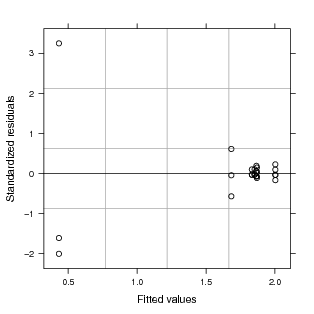
Os gráficos de resíduos das figuras 69 e 70 mostram observações discrepantes e pode-se detectar que ocorrem nos dados do Laboratório E. No primeiro desses todos os resíduos são visualizados conjuntamente, enquanto que no segundo usa-se gráficos condicionais do sistema gráfico fornecido pelo pacote lattice para separar os resíduos de cada laboratório.
A observação de valor 0.68 do laboratório E é bastante diferente das demais replicatas deste laboratório (0.28 e 0.31), sendo que este dado também foi considerado suspeito pela fonte dos dados. Uma possível alternativa é, em acordo com o responsável pelos dados, optar por remover este dado da análise o que pode ser feito com o comando a seguir.
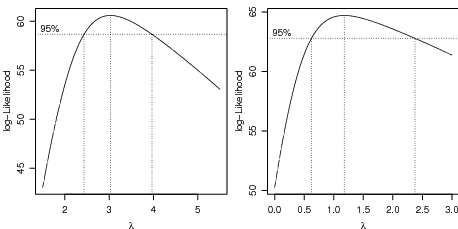
O modelo ajustado assume que os dados possuem distribuição normal e os gráficos de perfil de verossimilhança do parâmetro da transformação Box-Cox na figura 71 mostram que, excluindo-se o dado atípico, a transformação não é necessária.
O modelo ajustado com o novo conjunto de dados apresenta resultados diferentes do anterior, reduzindo a estimativa de variância entre as replicatas.
Além disto, nota-se que na verdade todas as observações do Laboratório E parecem atípicas com valores inferiores aos obtidos nos demais laboratórios. Poderia-se então considerar ainda remover todas as observações deste laboratório.
Os resultados são substancialmente diferentes e a decisão de exclusão on não dos dados deste Laboratório deve ser cuidadosamente investigada dentro do contexto destes dados e conjunto com especialista da área.
Assumindo que efeitos aleatórios podem ser usados para descrever o efeito de laboratórios, podemos descrever os teores por um modelo de efeitos aleatórios:
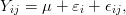
em que yij são valores observados na j-ésima medida feita no i-ésimo laboratório, μ é o valor real do elemento na amostra padrão, εi ~ N(0,σε2) é o efeito aleatório do i-ésimo laboratório e σ ε2 que representa a variabilidade de medidas fornecidas por diferentes laboratórios (entre laboratórios) e ϵij ~ N(0,σϵ2) é o termo associado à j-ésima medida feita no i-ésimo laboratório e σ ϵ2 é a variabilidade das medidas de replicatas dentro dos laboratórios.
O problema então consiste em estimar μ e a variância associada à esta estimativa, que por sua vez está associada aos valores dos parâmetros de variância do modelo σε2 e σ ϵ2. Esses últimos parâmetros são chamados de componentes de variância. Diferentes métodos de estimação são propostos na literatura tais como estimadores de momentos baseados na análise de variância, estimadores minque (estimadores de norma quadrática mínima), estimadores de máxima verossimilhança e máxima verossimilhança restrita.
Sob o modelo assumido os observações tem distribuição normal
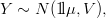
Considerando os recursos computacionais atualmente disponíveis e as propriedades dos diferentes estimadores, nossa preferência é pelo uso de estimadores de máxima verossimilhança restrita. Estes estimadores são obtidos maximizando-se a função de verossimilhaça de uma projeção do vetor dos dados no espaço complementar os definido pela parte fixa do modelo. Tipicamente, os estimadores de σε2 e σ ϵ2 são obtidos por maximização numérica de tal função e o estimador do parâmetro de interesse e sua variância são então obtidos por:
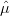 | = (1l′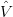 -11l)-11l′ -11l)-11l′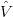 -1y -1y | (6) |
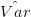 ( (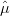 ) ) | = (1l′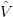 -11l)-1 -11l)-1 | (7) |
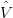 é a matrix de variâncias e covariâncias estimada das observações obtida a partir das
estimativas
é a matrix de variâncias e covariâncias estimada das observações obtida a partir das
estimativas 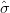 ε2 e
ε2 e 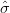 ϵ2.
ϵ2.
No exemplo em questão são estes os estimadores utilizados para obter as estimativas mostradas na Sessão anterior (ver o resultado de summary(mgo1.lme) e com valores mostrados novamente a seguir.
O intervalo de confiança para média pode então ser obtido por:
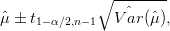
. Nos comandos a seguir mostramos a obtenção do intervalo segundo cálculos dessa expressão e a equivalência com o informado pela funçãom intervals.lme().
Para uma observação individual o intervalo é dado por
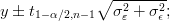
e as estimativas 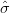 ε2 e
ε2 e 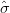 ϵ2 podem obtidas da seguinte forma.
ϵ2 podem obtidas da seguinte forma.
O coeficiente de correlação intraclasse reflete a relação entre a variabilidade das observações dentro dos laboratórios em relação a variabilidade total. É definido ela expressão a seguir e calculado como mostrado nas linhas de comando.
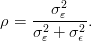
O pacote lme4 reimplementa algumas funcionalidades do nlme onde o modelo é definido indicando os termos aleatórios entre parênteses na fórmula e eliminando o uso do argumento random.
O comando para se obter uma análise equivalente à anterior é mostrado a seguir. Os resultados são apresentados de forma diferente, prém os elementos são equivalentes.
A opção padrão é o ajuste por máxima verossimilhança restrita. Estimativas de máxima verossimilhança podem ser obtidas usando o argumento REML=FALSE.