![[ ]1-(1∕n)
------1-----
θ = θR + (θS - θR ) 1 + (α Ψm )n](Rembrapa197x.png)
Modelos não lineares permitem o ajuste de relações mais complexas que relações lineares ou linearizáveis entre quantidades de interesse. Em muitos casos tais modelos tem a sua forma funcional específica para o prolema sendo tratado, relacionada a algum mecanismo (biológico, físico, etc) inerente ao processo em questão.
Nesta seção vamos ilustrar com dados da área de física de solos o ajuste de modelos não lineares utilizando a função nls(), cujo é um acrônimo para non-linear least squares. Esta função é bastante flexível e incorpora diversas opções para fazer ajustes incluindo características do modelo, tipo e controle dos algorítmos disponíveis.
Diferentemente dos modelos lineares, o ajuste de modelos não lineares não permite que as expressões dos estimadores dos parâmetros desconhecidos do modelo sejam obtidas analiticamente sendo portanto necessário o uso de métodos númericos. Inicialmente mostramos um ajuste feito de forma "ingênua"(naïve), declarando apenas a função e valores iniciais. Tal procedimento, embora simples, pode se ineficiente para o uso de métodos numéricos. Entretanto, o ajuste com nls() pode incorporar procedimentos que tendem a aprimorar o comportamento dos métodos numéricos tais como o fornecimento de funções que informem sobre a derivada do modelo sendo ajustado, inicialização automática com valores iniciais obtidos automaticamente, linearização parcial do modelo, além da escolha e calibragem dos algorítmos. O objetivo destas notas não é o de investigar todas estas opções, mas apenas fornecer os elementos iniciais para ilustrar a possibilidade de se obter tais resultados usando o R.
Este exemplo mostra o ajuste de um modelo não linear. Primeiro discutimos como efetuar um único ajuste para um conjunto de dados e algumas sugestões para examinar resultados. Ao final mostramos como efetuar vários ajustes de uma só vez de forma eficiente e extrair alguns resultados de particular interesse.
O exemplo mostrado aqui foi motivado por um questão levantada pelo Prof. Álvaro Pires da Silva do Departamento de Ciência do Solo da esalq/usp e refere-se ao ajuste da equação de van Genutchen para a curva de retenção de água no solo (ou curva de retenção de água no solo).
Informalmente falando, a equação de van Genutchen é um dos modelos matemáticos utilizados para descrever a curva característica de água no solo que caracteriza a armazenagem de água através de relação entre a umidade e o potencial matricial. Para determinação da curva característica de água o procedimento usual é o de se tomar uma amostra que é submetida a diferentes tensões em condições de laboratório. Para cada tensão aplicada a amostra perde parte do conteúdo de água e mede-se a umidade residual na amostra. A partir dos pares pontos com valores medidos de tensão e umidade, obtem-se a curva de retenção de água no solo que descreve a variação da umidade em função dos valores de tensão. O modelo de van Genutchen é dado pela seguinte equação:
![[ ]1-(1∕n)
------1-----
θ = θR + (θS - θR ) 1 + (α Ψm )n](Rembrapa197x.png) | (8) |
em que Ψm é o potencial matricial aplicado à amostra e θ é a umidade volumétrica medida na amostra. O parâmetros desconhecidos do modelo modelo são θS e θR que correspondem à umidade volumétrica na saturação e residual, respectivamente, α e n que definem o formato da curva sendo que o primeiro representa o inverso do potencial de entrada de ar e o segundo é um índice da distribuição dos tamanhos de poros. Portanto são obtidos dados para os pares de pontos (Ψm,θ) e (θS,θR,α,n) são parâmetros desconhecidos a serem estimados e que caracterizam a curva de retenção.
Para exemplificar o ajuste utilizamos dados cedidos pelo Prof. Álvaro que podem ser obtidos usando o comando mostrado a seguir. Este conjunto de dados refere-se a apenas duas amostras que são um subconjunto dos de dados original que contém diversas amostras. O objetivo é determinar da curva de retenção de água no solo estimada segundo modelo de van Genutchen para cada uma das amostras. No objeto cra a primeira coluna (am) indica o número da amostra, a segunda (pot) o potencial aplicado e a terceira (u) a umidade do solo. Vemos a seguir que dispomos de 15 pontos medidos da curva de retenção da primeira amostra e 13 para a segunda.
Inicialmente vamos nos concentrar na discussão do ajuste do modelo e para isto, vamos isolar os dados referentes a uma única amostra.
No gráfico à esquerda da Figura 30.1 visualizamos os dados de umidade versus pressão aplicada na amostra.
Uma melhor visualização é obtida utilizando-se no eixo horizontal o logarítmo (base 10) dos valores das pressões aplicadas conforme mostrado no gráfico à direita.
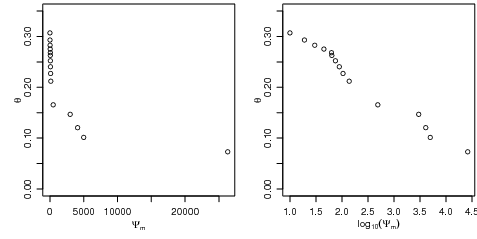
Portanto, os dados nas colunas u e pot do objeto de dados correspondem à θ e ψm na equação 8, e as demais quantidades (θR,θR,n,α) são parâmetros (coeficientes) a serem estimados a partir do ajuste do modelo teórico aos dados. Este é um modelo não linear pode ser ajustado utilizando o método de mínimos quadrados conforme implementado em nls(). A função possui três argumentos obrigatórios: (i) o primeiro é utilizado para declarar a expressão do modelo a ser ajustado, (ii) o segundo informa o objeto contendo o conjunto de dados cujas nomes das colunas relevantes devem ter o mesmo nome utilizado na declaração do modelo e, (iii) valores iniciais para os parâmetros a serem ajustados que devem ser passados por uma named list, isto é, uma lista com nomes dos elementos, e estes nomes também devem coincidir com os utilizados na declaração do modelo. Há argumentos adicionais para controlar o comportamento algorítimo, tal como critério de convergência. A documentação de nls() fornece mais detalhes.
A escolha dos valores iniciais é crucial e pode influenciar nos resultados do ajuste utilizando métodos numéricos, especialmente em exemplos como este com um pequeno número de dados. Os valores iniciais para θS e θR foram escolhidos inspecionando-se o gráfico e considerando a interpretação destes como valores de saturação e residual de umidade, pontanto considerando-se máximos e mínimos assintóticos para a função. A escolha de valores iniciais para os demais parâmetros é menos óbvia. Uma das formas de se obter tais valores é efetuar um ajuste aproximado, visual por tentativa e erro, traçando-se curvas sobre o gráfico dos dados. O comando a seguir ilustra como fazer tal procedimento a partir do gráfico dos dados originais mostrado anteriormente definindo uma expressão para curve() com o modelo de van Genutchen. Os valores foram escolhidos após uma séria de tentativas.
Definidos os valores iniciais prossegue-se com o ajuste do modelo conforme os comandos a seguir.
A partir do modelo ajustado pode-se calcular quantidades de interesse. Neste particular exemplo calculamos uma quantidade de interesse prático denotada por S que é um indicador da qualidade física do solo. Quanto maior o valor de S, melhor a sua qualidade física.
Os valores preditos são obtidos de forma direta com fitted(fit30) ou predict(fit30). Para visualização e avaliação do modelo ajustado podemos fazer diferentes gráficos. A Figura 30.1 mostra os pontos ajustados no gráfico da esquerda, e a união destes pontos no gráfico da direita. Gráficos de resíduos semelhantes aos obtidos para avaliar ajuste de modelos lineares podem e devem também ser investivados em uma análise. Neste exemplo mostramos o qq-plot dos resíduos e o gráfico dos resíduos versus valores preditos.
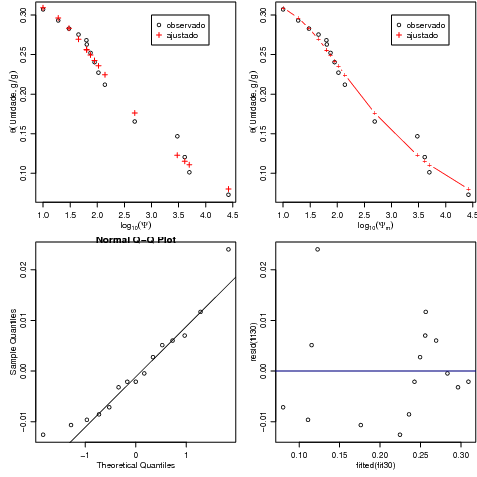
Para obter uma melhor visualização do modelo ajustado pode-se obter valores na curva ajustada não apenas nos pontos observados, mas em uma sequência de valores ao longo do gráfico como ilustrado a seguir. A Figura 30.1 mostra à direita o modelo definido pelos valores iniciais e o modelo ajustado, na escala original. o modelo ajustado na escala original Note que neste exemplo em geral prefere-se a visualização na escala logarítmica do potencial conforme gráfico da direita. A curva com o modelo ajustado a serem desenhadas sobre o gráfico dos dados são obtidas com comandos a seguir.
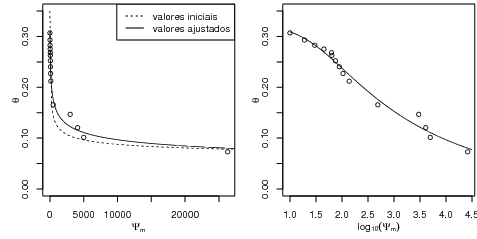
Comentários: é importante lembrar que certos modelos não lineares são parcialmente linearizáveis e neste caso o ajuste pode ser mais preciso e numericamente estável se beneficiando disto para reduzir a dimensão do problema de otimização numérica. Para isto é necessário redefinir a especicifação do modelo e utilizar o argumento method="plinear" em nls(). Neste exemplo em particilar pode-se considerar fazer o ajuste na escala de log10(Ψm) já que os resultados são tipicamente visualizados desta forma. Isto reduz a escala dos valores das variáveis e também torna o problema mais estável numericamente. Por outro lado, em geral reparametrizações podem mudar a interpretação de alguns parâmetros de modelo. Finalmente cuidados usuais com ajuste de modelos utilizando métodos iterativos devem ser observados, tais como sensibilidade a valores iniciais e verificação de convergência do algorítmo numérico.
Vamos considerar uma situação comum na prática onde em geral tem-se várias amostras para as quais deseja-se fazer ajuste individuais como ilustrado anteriormente É portanto conveniente que isto seja feito de forma automática, sem a necesidade e repetir os passos acima a cada ajuste. Neste exemplo vamos considerar duas amostras, mas o procedimento demostrado a seguir é geral e funcionará igualmente para um maior número de amostras.
Serão mostradas duas soluções. Nesta sessão o ajuste é feito para cada amostra individualmente automatizando várias chamadas à função nls() através de lapply() emulando o comportamento das várias chamadas em um loop. Na próxima sessão será mostrado como obter os todos os ajustes com uma única chamada à nls(). Ilustramos ambos casos porque a forma mais adequada vai depender de situação em questão e dos objetivos da análise.
Começamos definindo uma função que contém uma chamada à nls() como acima. Neste função estamos incluindo um argumento ini para passar valores iniciais que caso não fornecido assumirá os valores indicados. A seguir utilizamos a função by() para proceder o ajuste para cada amostra individualmente. Esta função retorna uma lista com dois elementos, um para cada amostra, sendo que cada um deles contém o ajuste do modelo não linear.
Neste caso, o objeto resultante allfits é uma lista de listas e portanto podemos usar funções como lapply(), sapply() ou similares para extrair resultados de interesse. Note que a primeira retorna sempre uma lista, enquanto que a segunda "simplifica"o objeto resultante se possível. Por exemplo, quando extraindo coeficientes a função retorna uma matrix 4 × 2, já que para cada uma das duas amostras são extraidos quatro coeficientes.
Quando ajustamos o modelo apenas para uma das amostras mostramos como calcular o índice S de qualidade física do solo a partir dos coeficientes estimados. Vamos então aqui obter este índice para cada uma das amostra. Para isto simplesmente definimos uma função que recebe o modelo ajustado e usa os coeficiente para calcular o valor de S. Passamos o objeto (lista) contendo todos os ajustes e a função que calcula S para sapply() que neste caso vai simplificar o resultado para formato de um vetor, já que a função calculaS retorna um escalar para cada amostra.
Finalmente, para encerrar este exemplo, vamos mostrar uma possível forma de combinar a visualização dos ajustes em em um único gráfico. Começamos definindo uma sequência de valores para os quais queremos visualizar os ajustes. Armazenamos os valores preditos para cada amostra no objeto allpred e optamos aqui por mostrar os ajustes para as duas amostras no mesmo gráfico.
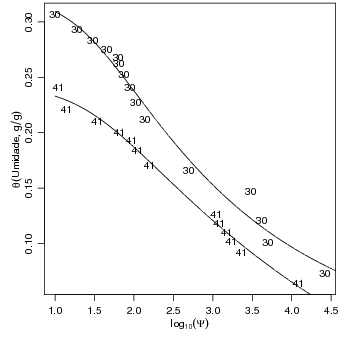
Na sessão anterior obtivemos o ajusta para cada amostra separadamente fazendo várias chamadas à função nls(). Isto pode ser adequado quando deseja-se de fato ajustes individuais e se, por um lado são efetuadas várias chamadas à função, por outro o número de dados em cada uma delas é pequeno. Uma forma alternativa de obter parâmetros para cada amostra, e talvez mais eficiente que a mostrada anteriormente é discutida a seguir.
Nesta sessão vamos considerar fazer todos os ajustes de só vez, isto é em uma única chamada à nls() que portanto vai utilizar todos os dados de todas as amostras. Além do aspecto computacional, isto pode ser interessante pois permite comparar e testar hipóteses para escolha entre diferentes modelos alternativos para explicar os dados Exemplificamos tal procedimento a seguir iniciando com um modelo para cada amostra e comparando com um modelo assume que os parâmetros (α, n) são comuns entre as amostras. Neste caso interpreta-se que cada amostra informa sobre os respectivos valores para (θS, θR) enquanto que todas as amostrs conjuntamente informam sobre (α, n). Após ajustar os modelos "candidatos"podemos fazer uma comparação formal dos ajustes atravez de anova(), o que não seria possível ajustando os modelos separadamente como mostrado sessão anterior. Os dois ajustes são mostrados a seguir o seletor [] é usado para indicar que os dados são tratados em grupos definidos por am. No caso do modelo com parâmetros distintos informamos oito valores iniciais para os parâmetros.
Para ajuste assumindo valores comuns para os parâmetros α e n não utilizamos o indicados de grupos para estes parâmetros e informamos apenas um valor inicial para cada um deles.
Neste exemplo temos então um modelo inicial com oito e outro mais parcimonioso com apenas seis parâmetros e utilizamos um teste formal para orientar a escolha de modelo, que neste caso indica que o modelo mais parcimonioso com parâmetros comuns explica os dados satisfatóriamente.