Essa é uma revisão anterior do documento!
Tabela de conteúdos
CE-225 - Segundo semestre de 2012
No quadro abaixo será anotado o conteúdo dado em cada aula do curso.
É indicado material para leitura correspondente ao conteúdo da aula nas referências bibliográficas básicas do curso:
- T & S: M. Antónia Amaral Turkman & Giovani Loiola Silva
- C & D: Gauss M. Cordeiro & Clarice G. B. Demétrio
- P: Gilberto A. Paula
| T & S | C & D | P | ||||||
|---|---|---|---|---|---|---|---|---|
| Data | Local | Conteúdo | Leitura | Exercícios | Leitura | Exercícios | Leitura | Exercícios |
| Introdução | ||||||||
| 31/10 | PA-03 | Introdução geral à disciplina | ||||||
| 07/11 | PA-03 | Modelo Linear. Método dos mínimos quadrados. Método da máxima verossimilhança. | ||||||
| 09/11 | PA-03 | Propriedades dos estimadores de máxima verossimilhança | ||||||
| 14/11 | PA-03 | Família Exponencial de distribuições | ||||||
| 21/11 | PA-03 | Definição dos Modelos lineares generalizados | ||||||
| 23/11 | LABEST | Modelo linear: aplicação a peso de nascidos vivos em 2010 (script) | ||||||
| 28/11 | PA-03 | Revisão | ||||||
| 30/11 | LABEST | Algoritmo de Fisher e aplicação (script) e contas matriciais em R (|script) | ||||||
| 05/12 | PA-03 | Avaliação 1 | ||||||
| 07/12 | PA-03 | Correção da avaliação 1 | ||||||
| 12/12 | PA-03 | Estimação: Algoritmo IWLS - definições | ||||||
| 14/12 | LABEST | Estimação: Algoritmo IWLS - exemplos (script) | ||||||
| 23/01 | PA-03 | Modelagem estatística: de LM's a GLM's | ||||||
| 25/01 | PA-03 | Comparando modelos e estratégias de modelagem em determinados problemas - em quais aspectos os modelos são diferentes? Modelos lineares, linearizáveis, não lineares, normais e não normais. Avaliação e comparação de ajustes de modelos. | ||||||
| 30/01 | LABEST (VER ABAIXO) | Exemplos de problemas sob a forma de GLM's | ||||||
| 01/02 | LABEST (VER ABAIXO) | Interpretações de resultados das análises | ||||||
| 06/02 | PA-03 (VER ABAIXO) | Testes de hipótese em GLM e tipos de resíduos, com ênfase em de Pearson e de Deviance | ||||||
| 08/02 | LABEST (VER ABAIXO) | |||||||
30/01 e 01/02
Considere o conjunto de dados "heart" Sobre número de pacientes com e sem ataque cardíaco para diferentes níveis da enzima creatinina kinase no sangue. O objetivo é examinar se os níveis da enzima podem ser utilizados como auxiliares de diagnóstico.
Ataque cardíaco
------------------
CK Sim Nao
-----------------------------------
20 2 88
60 13 26
100 30 8
140 30 5
180 21 0
220 19 1
260 18 1
300 13 1
340 19 1
380 15 0
420 7 0
460 8 0
----------------------------------
Analise os dados. A seguir são dadas algumas (mas não todas!) questões que podem ser consideradas nas análises.
- Qual modelo (GLM) poderia ser adotado?
- Que gráficos exploratórios poderiam ser feitos?
- Como pode-se investigar o efeito da CK ?
- O modelo NULO é suficiente para explicar os dados?
- O modelo COMPLETO superior ao NULO para explicar os dados?
- Como podemos avaliar a qualidade do ajuste do modelo completo?
- Como podemos detarminar se a covariável é realmente relevante?
- Como obter intervalos para os parâmetros?
- Como obter, avaliar e fazer gráficos do modelo predito?
- Como avaliar diferentes opções para função de ligação?
- Como avaliar se a linearidade das covariáveis selecionadas? (por exemplo, uma função quadrática ou cúbica no modelo linear seria melhor?)
infarto <- read.table("clipboard")
infarto
names(infarto) <- c("CK","S","N")
infarto
infarto <- transform(infarto, prop = S/(S+N))
with(infarto, plot(prop ~ CK))
mod0 <- glm(cbind(S,N) ~ 1, family=binomial, data=infarto)
mod0
summary(mod0)
logLik(mod0)
deviance(mod0)
mod1 <- glm(cbind(S,N) ~ CK, family=binomial, data=infarto)
mod1
summary(mod1)
logLik(mod1)
deviance(mod1)
anova(mod1)
anova(mod1, test="Chisq")
-2*(logLik(mod0) - logLik(mod1))
(pred1 <- predict(mod1))
(pl <- with(infarto, coef(mod1)[1] + coef(mod1)[2] * CK))
with(infarto,plot(prop/(1-prop), pred1, asp=1)); abline(0,1)
(pred1p <- predict(mod1, type="response"))
exp(pl)/(1+exp(pl))
with(infarto,plot(prop , pred1p, asp=1)); abline(0,1)
(pred1y <- with(infarto, (S+N)*pred1p))
cbind(infarto$S, pred1y)
plot(infarto$S, pred1y, asp=1); abline(0,1)
with(infarto, plot(prop/(1-prop) ~ CK))
with(infarto, lines(pred1 ~ CK, type="b"))
with(infarto, plot(prop/(1-prop), pred1)); abline(0,1)
with(infarto, plot(prop ~ CK))
with(infarto, lines(pred1y ~ CK, type="b", col=2, pch=19, cex=0.5))
with(infarto, plot(prop, pred1y)); abline(0,1)
mod2 <- glm(cbind(S,N) ~ CK + I(CK^2), family=binomial, data=infarto)
mod3 <- glm(cbind(S,N) ~ CK + I(CK^2) + I(CK^3), family=binomial, data=infarto)
mod12 <- glm(cbind(S,N) ~ as.factor(CK), family=binomial, data=infarto)
(pred12p <- predict(mod12, type="response"))
infarto$prop
c(logLik(mod0), logLik(mod1), logLik(mod2), logLik(mod3), logLik(mod12))
anova(mod0, mod1, mod2, mod3, mod12)
anova(mod0, mod1, mod2, mod3, mod12, test="Chisq")
mod1b <- glm(cbind(S,N) ~ CK, family=binomial(link="probit"), data=infarto)
mod1c <- glm(cbind(S,N) ~ CK, family=binomial(link="cauchit"), data=infarto)
logLik(mod1a)
logLik(mod1b)
logLik(mod1c)
mod3a <- glm(cbind(S,N) ~ poly(CK, 3), family=binomial, data=infarto)
mod3
mod3a
logLik(mod3)
logLik(mod3a)
deviance(mod3)
deviance(mod3a)
summary(mod3)
summary(mod3a)
anova(mod3)
anova(mod3a)
mod3b <- glm(cbind(S,N) ~ poly(CK, 3), family=binomial(link="probit"), data=infarto)
mod3c <- glm(cbind(S,N) ~ poly(CK, 3), family=binomial(link="cauchit"), data=infarto)
logLik(mod3a)
logLik(mod3b)
logLik(mod3c)
deviance(mod0)
-2*(logLik(mod0) - logLik(mod12))
deviance(mod1)
-2*(logLik(mod1) - logLik(mod12))
deviance(mod2)
-2*(logLik(mod2) - logLik(mod12))
deviance(mod3)
-2*(logLik(mod3) - logLik(mod12))
require(mgcv)
modgam <- gam(cbind(S,N) ~ s(CK), family=binomial, data=infarto)
modgam
logLik(modgam)
deviance(modgam)
plot(modgam)
with(infarto, plot(predict(modgam) ~ CK))
with(infarto, points(log(prop/(1-prop)) ~ CK))
## Fazer graficos de ajuste para o modelo 3 !!!
–
Considere o conjunto de dados "gala" com pacote "faraway"
require(faraway) data(gala) head(gala) help(gala)Nosso objetivo é montar um modelo para tentar explicar a variável resposta "número de especies" pelas demais variáveis disponíveis.
Vamos utilizar a função glm() do R, e seguem algumas sugestões (não exaustivas!!!) de tópicos a serem explorados pelas análises.
- Qual modelo (GLM) poderia ser adotado?
- Que gráficos exploratórios poderiam ser feitos?
- O modelo NULO é suficiente para explicar os dados?
- O modelo COMPLETO superior ao NULO para explicar os dados?
- Como podemos avaliar a qualidade do ajuste do modelo completo?
- Como podemos selecionar as covariáveis realmente relevantes?
- Como obter intervalos para os parâmetros?
- Como obter, avaliar e fazer gráficos do modelo predito?
- Como avaliar diferentes opções para função de ligação?
- Como avaliar se a linearidade das covariáveis selecionadas? (por exemplo um modelo com log(Area) seria melhor?)
06/02 e 08/02
Exemplo 1
A tabela abaixo apresenta dados de um estudo sobre "Acreditar em Vida Após a Morte" retirados de Wood (2006).
O interesse aqui é utilizar estes dados para investigar se a crença está associada com o sexo.
Embora várias abordagens e testes estatísticos sejam possíveis, nosso interesse investigar o problema a partir da
especificação de GLM. Procure propor um GLM para o estudo, interpretar o(s) modelo(s) proposto(s), o nulo, o saturado,
interpretando as quantidades de interesse nas análises e ainda comparando com resultados de outro(s) procedimento(s).
Acredita
-------------------
Sexo Sim Não
------------------------------
F 435 147
M 375 134
------------------------------
Exemplo 2
Novos casos de AIDS na Bélgica de 1981 a 1993.
Modelo Epidêmico (Venables & Ripley, MASS).
Interesse: A taxa de novos casos está reduzindo?
Considere um modelo em que o número esperado de novos casos por ano seja:
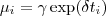
aids <- data.frame(
y = c(12,14,33,50,67,74,123,141,165,204,253,246,240),
ano = 1:13
)
aids
with(aids, plot(y ~ I(1980 + ano)))