|
Modelos de Regressão Não LinearFundamentos e Aplicações em R |
Acomodar estruturas de covariância nos erros
1 Variância heterogênea
library(latticeExtra)
library(nlme)
llayer <- latticeExtra::layer
source("https://raw.githubusercontent.com/walmes/wzRfun/master/R/panel.cbH.R")#-----------------------------------------------------------------------
# Leitura dos dados de peso de frutos de goiaba em função de dias após
# a antese (abertura da flor).
goi <- read.table("http://www.leg.ufpr.br/~walmes/data/goiaba.txt",
header = TRUE, sep = "\t")
str(goi)## 'data.frame': 174 obs. of 7 variables:
## $ daa : int 13 13 13 13 13 13 13 13 13 13 ...
## $ coleta: int 1 1 1 1 1 1 1 1 1 1 ...
## $ rep : int 1 2 3 4 5 6 7 8 9 10 ...
## $ long : num 29.1 32.9 36.3 34.7 33.3 ...
## $ trans : num 20.5 22.8 22.9 22.7 21.4 ...
## $ peso : num 6.66 8.51 9.93 9.14 7.6 7.54 6.27 9.94 7.95 7.79 ...
## $ volume: int 6 9 11 10 7 8 6 9 8 8 ...p0 <-
xyplot(peso ~ daa, data = goi, type = c("p", "a"), col = 1,
xlab = "Dias após a antese",
ylab = "Massa fresca do fruto (g)")
p0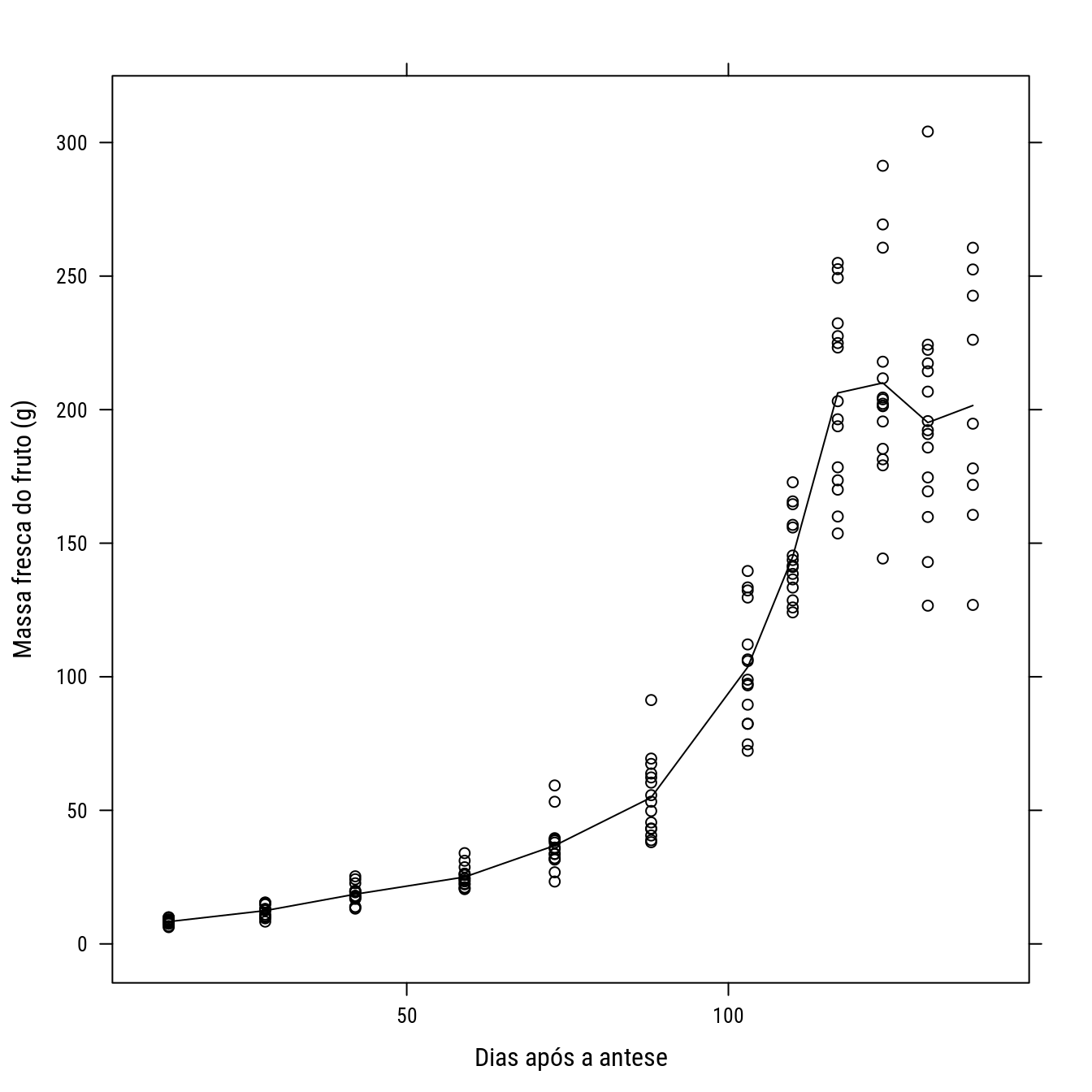
#-----------------------------------------------------------------------
# Gráfico das variâncias amostrais como função das médias.
# Relação log-log.
m <- with(goi, tapply(peso, daa, mean))
s <- with(goi, tapply(peso, daa, var))
# scatter.smooth(s ~ m)
plot(s ~ m, log = "xy")
#-----------------------------------------------------------------------
# Ajuste de modelos com mesma expressão para a média mas diferentes
# especificações para a variância.
# Modelo ordinário (erros homocedáticos).
# var(Y|x) ~ 1
m0 <- nls(peso ~ A - (A - D) * exp(-exp(C * (daa - B))),
data = goi,
start = c(A = 200, C = 0.09, B = 105, D = 7))
summary(m0)##
## Formula: peso ~ A - (A - D) * exp(-exp(C * (daa - B)))
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## A 205.76227 4.19618 49.036 < 2e-16 ***
## C 0.09367 0.01015 9.230 < 2e-16 ***
## B 107.35178 0.87439 122.773 < 2e-16 ***
## D 18.70608 3.17908 5.884 2.08e-08 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 24.94 on 170 degrees of freedom
##
## Number of iterations to convergence: 12
## Achieved convergence tolerance: 5.029e-06# var(Y|x) = sigma^2 * abs(daa)^{2 * delta}
m1 <- gnls(peso ~ A - (A - D) * exp(-exp(C * (daa - B))), data = goi,
start = c(A = 225, C = 0.05, B = 109, D = 7),
weights = varPower(value = 0.1, form = ~daa))
summary(m1)## Generalized nonlinear least squares fit
## Model: peso ~ A - (A - D) * exp(-exp(C * (daa - B)))
## Data: goi
## AIC BIC logLik
## 1418.027 1436.981 -703.0136
##
## Variance function:
## Structure: Power of variance covariate
## Formula: ~daa
## Parameter estimates:
## power
## 1.636154
##
## Coefficients:
## Value Std.Error t-value p-value
## A 225.63349 12.091086 18.66114 0
## C 0.04937 0.002928 16.85781 0
## B 109.96894 2.498714 44.01022 0
## D 6.74212 0.529455 12.73406 0
##
## Correlation:
## A C B
## C -0.643
## B 0.925 -0.720
## D -0.460 0.878 -0.485
##
## Standardized residuals:
## Min Q1 Med Q3 Max
## -2.56623148 -0.54890390 -0.03834664 0.64087134 2.66767440
##
## Residual standard error: 0.01342423
## Degrees of freedom: 174 total; 170 residual# log(unique(goi$daa))
# var(Y|x) = sigma^2 * abs(log(daa))^{2 * delta}
m2 <- gnls(peso ~ A - (A - D) * exp(-exp(C * (daa - B))),
data = goi,
start = c(A = 225, C = 0.05, B = 109, D = 7),
weights = varPower(value = 0.1, form = ~log(daa)))
summary(m2)## Generalized nonlinear least squares fit
## Model: peso ~ A - (A - D) * exp(-exp(C * (daa - B)))
## Data: goi
## AIC BIC logLik
## 1432.237 1451.191 -710.1183
##
## Variance function:
## Structure: Power of variance covariate
## Formula: ~log(daa)
## Parameter estimates:
## power
## 5.831728
##
## Coefficients:
## Value Std.Error t-value p-value
## A 218.30946 9.544151 22.87364 0
## C 0.05237 0.003337 15.69479 0
## B 108.28253 2.078245 52.10288 0
## D 7.10495 0.485460 14.63551 0
##
## Correlation:
## A C B
## C -0.595
## B 0.895 -0.653
## D -0.430 0.875 -0.429
##
## Standardized residuals:
## Min Q1 Med Q3 Max
## -2.68503221 -0.52857661 0.01298573 0.60922303 2.82847480
##
## Residual standard error: 0.0034759
## Degrees of freedom: 174 total; 170 residual# var(Y|x) = sigma^2 * exp(2 * delta * daa)
m3 <- gnls(peso ~ A - (A - D) * exp(-exp(C * (daa - B))),
data = goi,
start = c(A = 225, C = 0.05, B = 109, D = 7),
weights = varExp(value = 0.03, form = ~daa))
summary(m3)## Generalized nonlinear least squares fit
## Model: peso ~ A - (A - D) * exp(-exp(C * (daa - B)))
## Data: goi
## AIC BIC logLik
## 1402.368 1421.322 -695.184
##
## Variance function:
## Structure: Exponential of variance covariate
## Formula: ~daa
## Parameter estimates:
## expon
## 0.03010788
##
## Coefficients:
## Value Std.Error t-value p-value
## A 262.51053 28.861529 9.095517 0
## C 0.04446 0.002577 17.256077 0
## B 117.32303 4.541368 25.834291 0
## D 6.64837 0.721375 9.216258 0
##
## Correlation:
## A C B
## C -0.764
## B 0.973 -0.844
## D -0.520 0.840 -0.571
##
## Standardized residuals:
## Min Q1 Med Q3 Max
## -1.94206733 -0.64874567 -0.09044805 0.59309982 2.59031832
##
## Residual standard error: 1.070667
## Degrees of freedom: 174 total; 170 residual#-----------------------------------------------------------------------
# Grande impacto sobre a incerteza dos parâmetros.
summary(m0)$coeff[, 1:2]## Estimate Std. Error
## A 205.76226744 4.19618286
## C 0.09366616 0.01014825
## B 107.35178159 0.87438902
## D 18.70608471 3.17907987summary(m1)$tTable[, 1:2]## Value Std.Error
## A 225.63349086 12.091086102
## C 0.04936564 0.002928354
## B 109.96894480 2.498713695
## D 6.74211971 0.529455498summary(m2)$tTable[, 1:2]## Value Std.Error
## A 218.30945857 9.544151428
## C 0.05236619 0.003336532
## B 108.28252562 2.078244665
## D 7.10495060 0.485459621summary(m3)$tTable[, 1:2]## Value Std.Error
## A 262.51053141 28.861528693
## C 0.04446161 0.002576577
## B 117.32302543 4.541368109
## D 6.64837372 0.721374507#-----------------------------------------------------------------------
# Medidas da qualidade do ajuste.
# valor das log verossimilhanças
c(logLik(m0), logLik(m1), logLik(m2), logLik(m3))## [1] -804.5465 -703.0136 -710.1183 -695.1840# Teste da razão de verossimilhanças.
anova(m1, m0)## Model df AIC BIC logLik Test L.Ratio p-value
## m1 1 6 1418.027 1436.982 -703.0136
## m0 2 5 1619.093 1634.888 -804.5465 1 vs 2 203.0659 <.0001anova(m2, m0)## Model df AIC BIC logLik Test L.Ratio p-value
## m2 1 6 1432.237 1451.191 -710.1183
## m0 2 5 1619.093 1634.888 -804.5465 1 vs 2 188.8564 <.0001anova(m3, m0)## Model df AIC BIC logLik Test L.Ratio p-value
## m3 1 6 1402.368 1421.322 -695.1840
## m0 2 5 1619.093 1634.888 -804.5465 1 vs 2 218.725 <.0001#-----------------------------------------------------------------------
# Gráfico dos resíduos para os diferentes modelos.
residuos <- expand.grid(daa = goi$daa,
modelo = c("Constante",
"Potência",
"Potência do logaritmo",
"Exponencial"),
KEEP.OUT.ATTRS = FALSE)
residuos$respad <- c(residuals(m0, type = "p"),
residuals(m1, type = "p"),
residuals(m2, type = "p"),
residuals(m3, type = "p"))
residuos$fitted <- c(fitted(m0),
fitted(m1),
fitted(m2),
fitted(m3))
qqmath(~respad | modelo, data = residuos)
xyplot(respad ~ daa | modelo, data = residuos)
qqmath(~respad | modelo,
data = residuos, col = 1,
xlab = "Quantis teóricos esperados da distribuição normal padrão",
ylab = "Resíduos padronizados",
strip = strip.custom(bg = "gray90"),
layout = c(2, 2),
cex = 0.8)
xyplot(respad ~ daa | modelo,
data = residuos,
type = c("p", "smooth"),
col = c(1),
xlab = "Dias após a antese",
ylab = "Resíduos padronizados",
strip = strip.custom(bg = "gray90"),
layout = c(2, 2),
ylim = c(-4.5, 5.5))
#-----------------------------------------------------------------------
# Calculo das bandas de confiança.
model <- deriv3(~A - (A - D) * exp(-exp(C * (daa - B))),
c("A", "C", "B", "D"),
function(daa, A, C, B, D) {
NULL
})
l <- list(m0, m1, m2, m3)
names(l) <- levels(residuos$modelo)
L <- l
for(i in seq_along(L)){
m4 <- l[[i]]
U <- chol(vcov(m4))
pred <- data.frame(daa = seq(1, 140, 1))
m <- model(daa = pred$daa,
A = coef(m0)["A"],
C = coef(m0)["C"],
B = coef(m0)["B"],
D = coef(m0)["D"])
F0 <- attr(m, "gradient")
pred$y <- c(m)
pred$se <- sqrt(apply(F0 %*% t(U), 1, function(x) sum(x^2)))
z <- qnorm(0.975)
pred <- transform(pred, lwr = y - z * se, upr = y + z * se)
L[[i]] <- pred
}
str(L)## List of 4
## $ Constante :'data.frame': 140 obs. of 5 variables:
## ..$ daa: num [1:140] 1 2 3 4 5 6 7 8 9 10 ...
## ..$ y : num [1:140] 18.7 18.7 18.7 18.7 18.7 ...
## ..$ se : num [1:140] 3.17 3.17 3.17 3.17 3.17 ...
## ..$ lwr: num [1:140] 12.5 12.5 12.5 12.5 12.5 ...
## ..$ upr: num [1:140] 24.9 24.9 24.9 24.9 24.9 ...
## $ Potência :'data.frame': 140 obs. of 5 variables:
## ..$ daa: num [1:140] 1 2 3 4 5 6 7 8 9 10 ...
## ..$ y : num [1:140] 18.7 18.7 18.7 18.7 18.7 ...
## ..$ se : num [1:140] 0.528 0.528 0.527 0.527 0.527 ...
## ..$ lwr: num [1:140] 17.7 17.7 17.7 17.7 17.7 ...
## ..$ upr: num [1:140] 19.7 19.7 19.8 19.8 19.8 ...
## $ Potência do logaritmo:'data.frame': 140 obs. of 5 variables:
## ..$ daa: num [1:140] 1 2 3 4 5 6 7 8 9 10 ...
## ..$ y : num [1:140] 18.7 18.7 18.7 18.7 18.7 ...
## ..$ se : num [1:140] 0.483 0.483 0.483 0.483 0.482 ...
## ..$ lwr: num [1:140] 17.8 17.8 17.8 17.8 17.8 ...
## ..$ upr: num [1:140] 19.7 19.7 19.7 19.7 19.7 ...
## $ Exponencial :'data.frame': 140 obs. of 5 variables:
## ..$ daa: num [1:140] 1 2 3 4 5 6 7 8 9 10 ...
## ..$ y : num [1:140] 18.7 18.7 18.7 18.7 18.7 ...
## ..$ se : num [1:140] 0.721 0.721 0.721 0.721 0.721 ...
## ..$ lwr: num [1:140] 17.3 17.3 17.3 17.3 17.3 ...
## ..$ upr: num [1:140] 20.1 20.1 20.1 20.1 20.1 ...L <- dplyr::bind_rows(L, .id = "model")
L$model <- factor(L$model, levels = levels(residuos$model))
str(L)## 'data.frame': 560 obs. of 6 variables:
## $ model: Factor w/ 4 levels "Constante","Potência",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ daa : num 1 2 3 4 5 6 7 8 9 10 ...
## $ y : num 18.7 18.7 18.7 18.7 18.7 ...
## $ se : num 3.17 3.17 3.17 3.17 3.17 ...
## $ lwr : num 12.5 12.5 12.5 12.5 12.5 ...
## $ upr : num 24.9 24.9 24.9 24.9 24.9 ...#-----------------------------------------------------------------------
# Gráfico com as bandas de confiança.
#pdf("../figuras/goiaba.pdf", w=8, h=4)
ylim <- range(goi$peso)
xyplot(y ~ daa | model,
data = L,
type = "l",
col = 1,
ylab = "Massa fresca dos frutos (g)",
xlab = "Dias após a antese",
strip = strip.custom(bg = "gray90"),
prepanel = prepanel.cbH,
cty = "bands",
ly = L$lwr,
uy = L$upr,
panel = panel.cbH) +
as.layer(xyplot(peso ~ daa, data = goi, col = 1, cex = 0.2))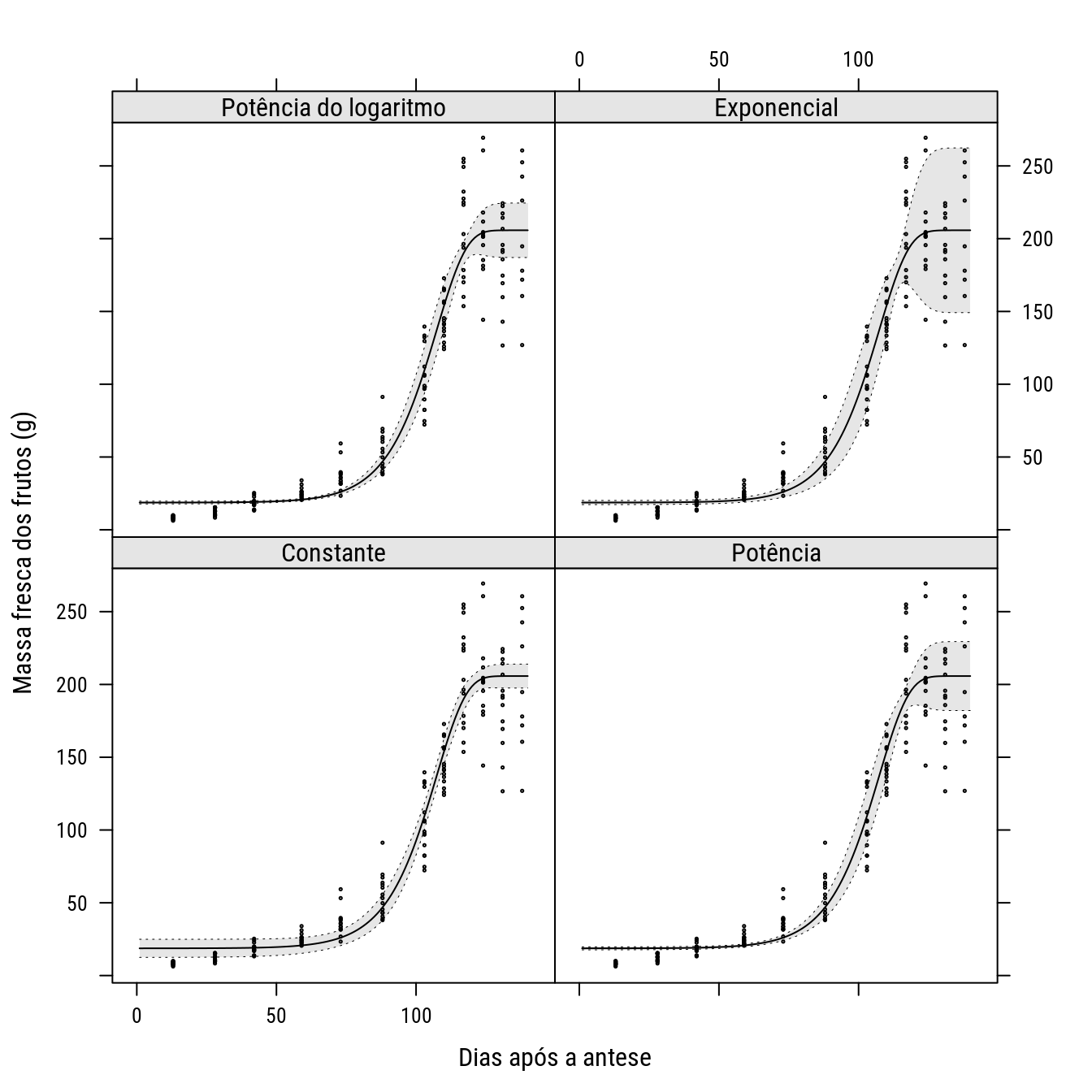
#-----------------------------------------------------------------------
# O modelo para a média pode ainda não ser o modelo adequado mas
# certamente qualquer um dos modelos que modelam a variância são mais
# adequados que o modelo ordinário. Se as inferências são para a parte
# final da curva então não existe tanta conseguência do não adequado
# ajuste à porção inicial dos dados. No entanto, deve-se procurar um
# modelo para a média mais próximo do comportamento dos dados.
#-----------------------------------------------------------------------
# Veja que curioso!
xyplot(log10(peso) ~ daa,
data = goi,
type = c("p", "a"), col = 1,
xlab = "Dias após a antese",
ylab = "Massa fresca do fruto (g)")
# A transformação tanto estabiliza a variância quanto torna a relação da
# média de Y com x mais simples. Pode-se usar um modelo resposta-platô.
#-----------------------------------------------------------------------2 Autocorrelação
|
Modelos de Regressão Não Linear: Fundamentos e Aplicações em R leg.ufpr.br/~walmes/cursoR/mrnl |
Prof. Walmes M. Zeviani Departamento de Estatística · UFPR |
