|
Modelos de Regressão Não LinearFundamentos e Aplicações em R |
Métodos de inferência para modelos não lineares
Exemplos com o modelo Van Genuchten
1 Assintóticos ou baseados na verossimilhança
rm(list = objects())
library(lattice)
library(latticeExtra)
library(rootSolve)
library(nlstools)
library(car)
llayer <- latticeExtra::layer
source("https://raw.githubusercontent.com/walmes/wzRfun/master/R/panel.cbH.R")1.1 Ajuste do modelo
O ajuste foi feito considerando a seguinte parametrização do modelo van Genuchten \[ U(x) = U_r + \frac{U_s - U_r}{(1 + \exp\{n(\alpha + x)\})^{1 - 1/n}} \] em que \(U\) é umidade (m3 m-3) do solo, \(x\) é o log na base 10 da tensão matricial aplicada (kPa), \(U_r\) é a umidade residual (assíntota inferior), \(U_s\) é a umidade de satuação (assíntota superior), \(\alpha\) e \(n\) são parâmetros empíricos de forma da curva de retenção de água.
Uma vez conhecido valores para as quatidades mencionadas, são obtidos \[ \begin{align*} S &= -n\cdot \frac{U_s - U_r}{(1 + 1/m)^{m + 1}} \newline I &= -\alpha - \log(m)/n \newline U_I &= U(x = I) \end{align*} \] em que \(S\) é a taxa no ponto de inflexão, parâmetro que é tido como central para avaliação da qualidade física do solo, bem como \(I\) que corresponde ao log da tensão no ponto de inflexão da curva de retenção de água do solo. A umidade correspondente a tensão no ponto de inflexão é representada por \(U_I\). Pela restrição Mualem (1976), \(m = 1 - 1/n\).
#-----------------------------------------------------------------------
# Importação dos dados.
cra <- read.table("http://www.leg.ufpr.br/~walmes/data/cra_manejo.txt",
header = TRUE, sep = "\t")
cra$tens[cra$tens == 0] <- 0.1
cras <- subset(cra, condi == "LVA3,5")
cras <- aggregate(umid ~ posi + prof + tens, data = cras, FUN = mean)
cras$caso <- with(cras, interaction(posi, prof))
cras$ltens <- log(cras$tens)
xyplot(umid ~ ltens | posi,
groups = prof, data = cras,
type = c("p", "a"))
# Escolhe apenas uma curva.
cra <- subset(cras, caso == "EL.20")
cra## posi prof tens umid caso ltens
## 1 EL 20 0.1 0.5733333 EL.20 -2.3025851
## 11 EL 20 1.0 0.5466667 EL.20 0.0000000
## 21 EL 20 2.0 0.5233333 EL.20 0.6931472
## 31 EL 20 4.0 0.4800000 EL.20 1.3862944
## 41 EL 20 6.0 0.4633333 EL.20 1.7917595
## 51 EL 20 10.0 0.4466667 EL.20 2.3025851
## 61 EL 20 33.0 0.4200000 EL.20 3.4965076
## 71 EL 20 100.0 0.3933333 EL.20 4.6051702
## 81 EL 20 500.0 0.3733333 EL.20 6.2146081
## 91 EL 20 1500.0 0.3700000 EL.20 7.3132204#-----------------------------------------------------------------------
# Ajuste.
# Expressão do modelo van Genuchten.
model <- umid ~ Ur + (Us - Ur)/(1 + exp(n * (alp + ltens)))^(1 - 1/n)
# Valores iniciais para os parâmetros da curva.
start <- list(Ur = 0.3, Us = 0.6, alp = -0.5, n = 4)
# Ajuste.
n0 <- nls(model, data = cra, start = start)
coef(n0)## Ur Us alp n
## 0.3605170 0.5765758 -0.2693376 1.4397566# Resumo do ajuste.
summary(n0)##
## Formula: umid ~ Ur + (Us - Ur)/(1 + exp(n * (alp + ltens)))^(1 - 1/n)
##
## Parameters:
## Estimate Std. Error t value Pr(>|t|)
## Ur 0.36052 0.00686 52.553 3.19e-09 ***
## Us 0.57658 0.00520 110.885 3.63e-11 ***
## alp -0.26934 0.18554 -1.452 0.197
## n 1.43976 0.05795 24.846 2.80e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.00504 on 6 degrees of freedom
##
## Number of iterations to convergence: 8
## Achieved convergence tolerance: 4.353e-06# Verificando.
xyplot(umid ~ ltens, data = cra, col = "black", pch = 19) +
llayer(panel.curve(Ur + (Us - Ur)/(1 + exp(n * (alp + x)))^(1 - 1/n),
col = "orange", lwd = 2),
data = as.list(coef(n0)))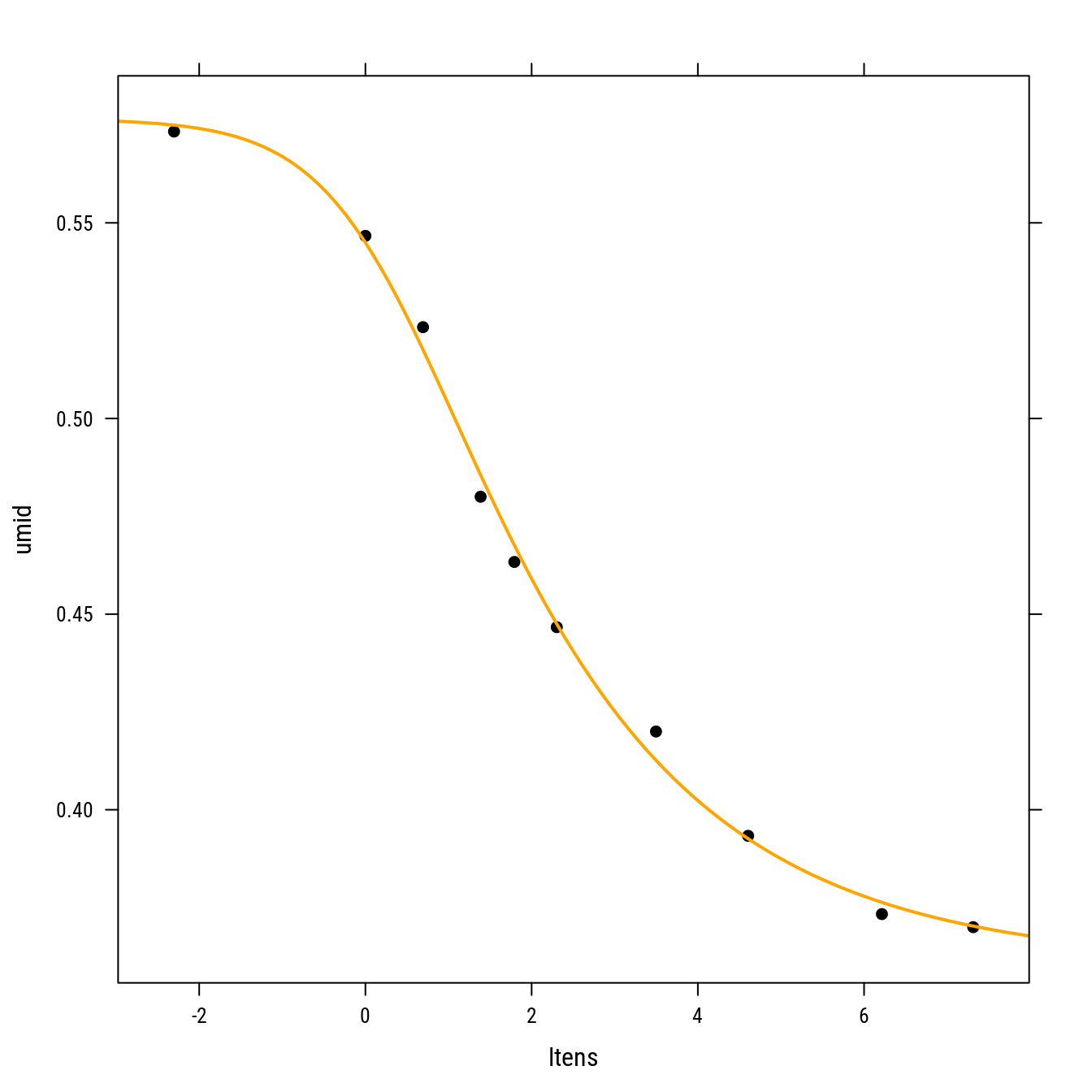
1.2 Intervalos de confiança para os parâmetros
#-----------------------------------------------------------------------
# Intervalos de confiança.
# Intervalos de Wald: são baseado na aproximação quadrática da
# verossimilhança, conhecido como intervalos de Wald ou
# assintóticos. São simétricos por construção.
wci <- confint.default(n0)
wci## 2.5 % 97.5 %
## Ur 0.3470715 0.37396258
## Us 0.5663844 0.58676714
## alp -0.6329936 0.09431849
## n 1.3261809 1.55333229pfl <- profile(n0)
# Intervalos de confiança baseados no perfil da log-verossimilhança.
# pci <- confint(n0)
# pci
confint(pfl)## 2.5% 97.5%
## Ur 0.3388796 0.3750252
## Us 0.5645621 0.5893191
## alp -0.6724646 0.2163408
## n 1.3104970 1.6003692# Perfil.
par(mfrow = c(2, 2))
plot(pfl)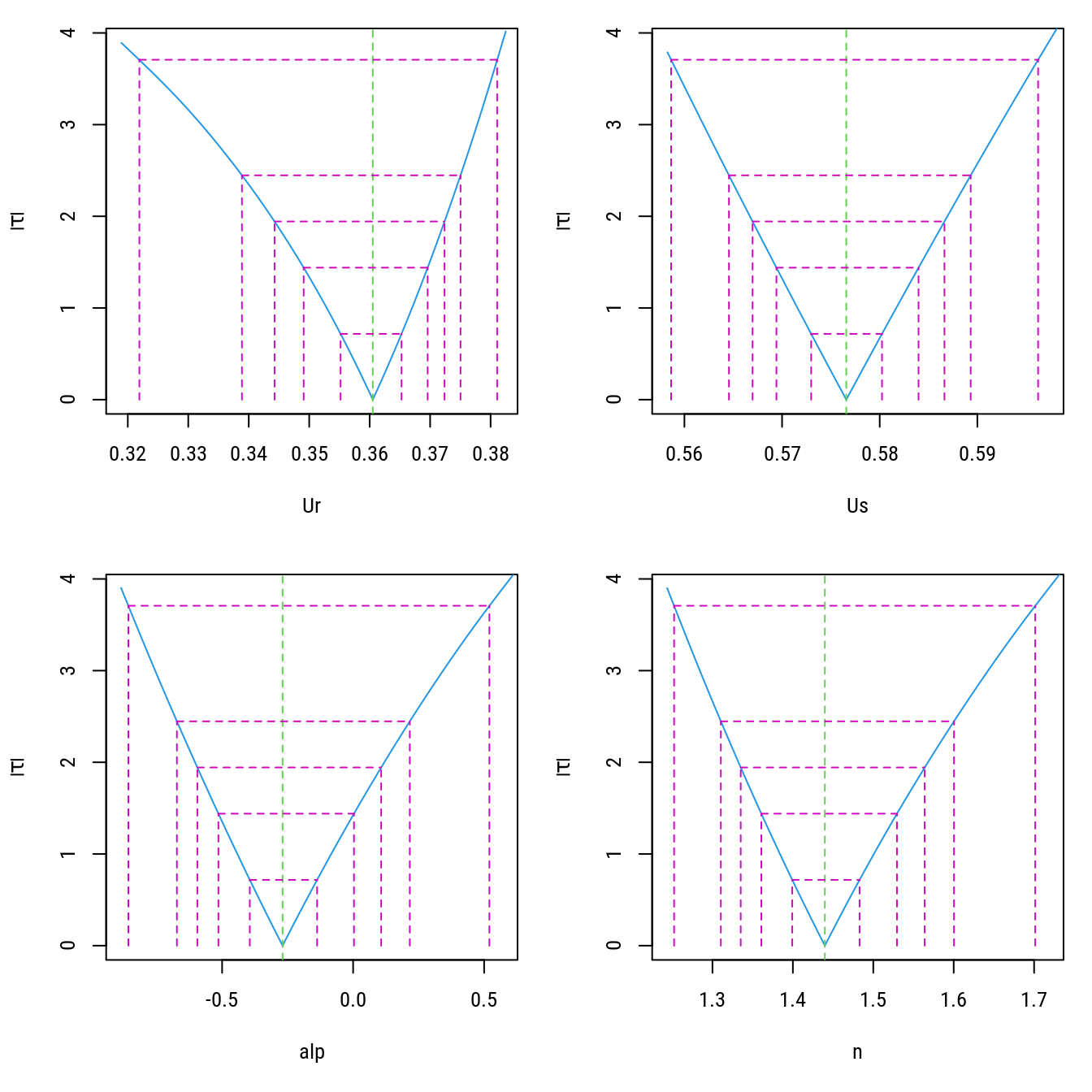
layout(1)1.3 Inferência assintótica para funções dos parâmetros
Método Delta é um procedimento para aproximar a distribuição de uma função de um vetor aleatório através de uma expansão de Taylor. É simples, mas útil, para deduzir a distribuição assintótica de variáveis aleatórias. O Teorema do Limite Central pode ser considerado como um caso particular do Método Delta. Para aplicar o método delta precisamos da função \(f(\theta)\), sua deriavada de primeira order \(f'(\theta)\), das estimativas \(\hat{\theta}\) e matriz de covariância das estimativas \(\text{Var}(\hat{\theta})\).
Vamos considerar para demostração que desejamos inferir sobre o parâmetro \(S\) acima definido como \[ \begin{aligned} S &= -n\cdot \frac{U_s - U_r}{(1 + 1/m)^{m + 1}}, \quad m = 1 - 1/n\\ &= -n\cdot \frac{U_s - U_r}{\left(\frac{2 - 1/n}{1 - 1/n}\right)^{2 - 1/n}}. \end{aligned} \]
Portanto, as funções necessárias são \[ \begin{align*} g(\theta) &= S = -n\cdot \frac{U_s - U_r}{ \left(\frac{2 - 1/n}{1 - 1/n}\right)^{2 - 1/n}}\\ g'(\theta) &= \text{vetor gradiente}. \end{align*} \]
#-----------------------------------------------------------------------
# Método delta.
# Derivadas parciais.
g_theta <- "-n * (Us - Ur)/((2 - 1/n)/(1 - 1/n))^(2 - 1/n)"
S_expr <- parse(text = g_theta)
# D(S_expr, "n")
# D(S_expr, "Ur")
# D(S_expr, "Us")
S_grad <- sapply(names(coef(n0)), D, expr = S_expr)
S_grad## $Ur
## n/((2 - 1/n)/(1 - 1/n))^(2 - 1/n)
##
## $Us
## -(n/((2 - 1/n)/(1 - 1/n))^(2 - 1/n))
##
## $alp
## [1] 0
##
## $n
## -((Us - Ur)/((2 - 1/n)/(1 - 1/n))^(2 - 1/n) + -n * (Us - Ur) *
## (((2 - 1/n)/(1 - 1/n))^((2 - 1/n) - 1) * ((2 - 1/n) * (1/n^2/(1 -
## 1/n) - (2 - 1/n) * (1/n^2)/(1 - 1/n)^2)) + ((2 - 1/n)/(1 -
## 1/n))^(2 - 1/n) * (log(((2 - 1/n)/(1 - 1/n))) * (1/n^2)))/(((2 -
## 1/n)/(1 - 1/n))^(2 - 1/n))^2)# Usando função pronta do pacote `car`.
dm <- deltaMethod(n0, g = g_theta)
rownames(dm) <- "S"
dm## Estimate SE 2.5 % 97.5 %
## S -0.0467033 0.0027953 -0.0521820 -0.0412# Fazendo passo a passo.
# Matriz de covariâncias.
v <- vcov(n0)
# Valor de g().
g_val <- eval(S_expr, envir = as.list(coef(n0)))
g_val## [1] -0.04670329# Valor de g'().
g_grad <- sapply(S_grad,
FUN = eval,
envir = as.list(coef(n0)))
g_grad <- rbind(g_grad)
g_grad## Ur Us alp n
## g_grad 0.2161601 -0.2161601 0 -0.07347609# Erro padrão de g(theta).
sqrt(g_grad %*% v %*% t(g_grad))## g_grad
## g_grad 0.0027953381.4 Bandas de confiança
Bandas de confiança são um caso particular de função das estimativas dos parâmetros. A forma de obter uma região de confiança é via aproximação linear, ou seja, método delta.
# Modelo escrito como função dos parâmetros (theta).
formula(n0)## umid ~ Ur + (Us - Ur)/(1 + exp(n * (alp + ltens)))^(1 - 1/n)f <- function(theta, ltens) {
with(as.list(theta),
Ur + (Us - Ur)/(1 + exp(n * (alp + ltens)))^(1 - 1/n))
}
# Matriz de derivadas parciais em \hat{\theta} (n x p).
gradient(f, x = coef(n0), ltens = log10(c(1, 3)))## Ur Us alp n
## [1,] 0.1463190 0.8536810 -0.03278927 -0.03995177
## [2,] 0.2295688 0.7704312 -0.04203480 -0.07463418# IMPORTANT: esse gradiente é obtido numericamente. Você pode definir
# uma função que retorne o gradiante usando as derivadas
# analíticas. Isse deve ser mais seguro e mais barato
# computacionalmente.
xlim <- extendrange(log(cra$tens))
pred <- data.frame(ltens = seq(xlim[1], xlim[2], length.out = 31))
# Valores ajustados (g(\theta)).
# pred$fit <- f(theta = coef(n0), ltens = pred$ltens)
pred$fit <- predict(n0, newdata = pred)
# Matriz gradiente (um gradiente para cada observação, g'(\theta)).
grad <- gradient(f, x = coef(n0), ltens = pred$ltens)
head(grad)## Ur Us alp n
## [1,] 0.003738088 0.9962619 -0.001153555 1.172640e-03
## [2,] 0.006177758 0.9938223 -0.001896472 1.455047e-03
## [3,] 0.010175105 0.9898249 -0.003096889 1.595043e-03
## [4,] 0.016667123 0.9833329 -0.005002332 1.291439e-03
## [5,] 0.027062896 0.9729371 -0.007941536 -4.978341e-05
## [6,] 0.043346804 0.9566532 -0.012275403 -3.469687e-03# Etapas até o IC passando pelo erro padrão e margem de erro.
U <- chol(vcov(n0))
pred$se <- sqrt(apply(grad %*% t(U), 1, function(x) sum(x^2)))
tval <- qt(p = c(lwr = 0.025, upr = 0.975), df = df.residual(n0))
me <- outer(pred$se, tval, "*")
pred <- cbind(pred, sweep(me, 1, pred$fit, "+"))
str(pred)## 'data.frame': 31 obs. of 5 variables:
## $ ltens: num -2.78 -2.43 -2.08 -1.73 -1.37 ...
## $ fit : num 0.576 0.575 0.574 0.573 0.571 ...
## $ se : num 0.005 0.00489 0.00472 0.00448 0.00415 ...
## $ lwr : num 0.564 0.563 0.563 0.562 0.561 ...
## $ upr : num 0.588 0.587 0.586 0.584 0.581 ...# Observados, preditos e a banda de confiança.
xyplot(umid ~ ltens, data = cra) +
as.layer(xyplot(fit ~ ltens,
data = pred,
type = "l",
prepanel = prepanel.cbH,
cty = "bands",
ly = pred$lwr,
uy = pred$upr,
panel = panel.cbH))
2 Métodos computacionais
2.1 Bootstrap
Útil para obter intervalo de confiança para os parâmetros e funções de parâmetros.
#-----------------------------------------------------------------------
# Bootstrap.
# Intervalo de confiança bootstrap (bootstrap não paramétrico).
bci <- nlsBoot(nls = n0, niter = 1000)
summary(bci)##
## ------
## Bootstrap statistics
## Estimate Std. error
## Ur 0.3605058 0.005276492
## Us 0.5764996 0.004061391
## alp -0.2717622 0.143205605
## n 1.4442989 0.045319464
##
## ------
## Median of bootstrap estimates and percentile confidence intervals
## Median 2.5% 97.5%
## Ur 0.3605415 0.3501181 0.37054197
## Us 0.5760963 0.5701454 0.58485145
## alp -0.2750114 -0.5397752 0.02287787
## n 1.4420162 1.3629330 1.53749738# Distribuição conjunta das estimativas bootstrap.
# pairs(bci$coefboot)
plot(bci)
#-----------------------------------------------------------------------
# Usando bootstrap uma função de parâmetros.
# Função avaliada na estimativa bootstrap.
bci$estiboot[, "Estimate"]## Ur Us alp n
## 0.3605058 0.5764996 -0.2717622 1.4442989eval(S_expr, envir = as.list(bci$estiboot[, "Estimate"]))## [1] -0.04702236# Função avaliada em cada ajuste.
bg <- apply(bci$coefboot,
MARGIN = 1,
FUN = function(th_i) {
eval(S_expr, envir = as.list(th_i))
})
# Mediana e erro padrão.
median(bg)## [1] -0.04679502sd(bg)## [1] 0.002202843# IC percentil.
quantile(bg, probs = c(0.025, 0.975))## 2.5% 97.5%
## -0.05141595 -0.04281475#-----------------------------------------------------------------------
# Adiciona as bandas do bootstrap.
# Observados, preditos e a banda de confiança.
xyplot(umid ~ ltens, data = cra) +
llayer({
th <- bci$coefboot
for (i in 1:nrow(th)) {
with(as.list(th[i, ]), {
panel.curve(
Ur + (Us - Ur)/(1 + exp(n * (alp + x)))^(1 - 1/n),
col = rgb(0, 1, 0, 0.05))
})
}
}, under = TRUE) +
as.layer(xyplot(fit ~ ltens,
data = pred,
type = "l",
prepanel = prepanel.cbH,
cty = "bands",
ly = pred$lwr,
uy = pred$upr,
panel = panel.cbH))
2.2 Jackknife
Permite determinar o vício das estimativas e obter intervalo de confiança.
#-----------------------------------------------------------------------
# Jackknife.
# Intervalo de confiança bootstrap (bootstrap não paramétrico).
jci <- nlsJack(nls = n0)
summary(jci)##
## ------
## Jackknife statistics
## Estimates Bias
## Ur 0.3647461 -0.004229046
## Us 0.5571080 0.019467818
## alp -0.6789955 0.409657973
## n 1.4777074 -0.037950813
##
## ------
## Jackknife confidence intervals
## Low Up
## Ur 0.3479492 0.3815430
## Us 0.5037130 0.6105029
## alp -1.8574998 0.4995087
## n 1.2753712 1.6800436
##
## ------
## Influential values
## * Observation 1 is influential on Ur
## * Observation 1 is influential on Us
## * Observation 1 is influential on alp
## * Observation 3 is influential on alp
## * Observation 7 is influential on alp
## * Observation 1 is influential on n
## * Observation 7 is influential on n# Observações influentes.
plot(jci)
# Estimativas parciais (ajuste sem a i-ésima observação).
jci$coefjack[2, ]## Ur Us alp n
## 0.3593239 0.5760651 -0.2342528 1.4264996coef(update(n0, data = cra[-2, ]))## Ur Us alp n
## 0.3593238 0.5760652 -0.2342499 1.4264987# Estimativa a partir da estimativa Jackknife.
eval(S_expr,
envir = as.list(setNames(jci$estijack[, "Estimates"],
rownames(jci$estijack))))## [1] -0.04403122# Para obter a estimativa com erro padrão via Jackknife.
n <- length(fitted(n0))
# Estimativa com todas as observações.
full_est <- eval(S_expr,
envir = as.list(coef(n0)))
full_est## [1] -0.04670329# Estimativas parciais sem a i-ésima observação.
part_est <- apply(jci$coefjack,
MARGIN = 1,
FUN = function(th_i) {
eval(S_expr, envir = as.list(th_i))
})
# Vetor de pseudo valores.
pv_g <- n * full_est - (n - 1) * part_est
# Estimativa pontual e erro padrão de Jackknife.
mean(pv_g)## [1] -0.04518224sd(pv_g)/sqrt(n)## [1] 0.0037668862.3 Regiões de confiança
A região de confiança é obtida por simulação Monte Carlo.
#-----------------------------------------------------------------------
# Região de confiança para os parâmetros.
# Contornos para a região de confiança baseados na função residual sum
# of squares.
rss <- nlsContourRSS(n0)## 0%16%33%50%66%83%100%
## RSS contour surface array returnedplot(rss)
# Regiões baseadas em aceitação-rejeição.
cr <- nlsConfRegions(n0, exp = 3)## 0%1%2%3%4%5%6%7%8%9%10%11%12%13%14%15%16%17%18%19%20%21%22%23%24%25%26%27%28%29%30%31%32%33%34%35%36%37%38%39%40%41%42%43%44%45%46%47%48%49%50%51%52%53%54%55%56%57%58%59%60%61%62%63%64%65%66%67%68%69%70%71%72%73%74%75%76%77%78%79%80%81%82%83%84%85%86%87%88%89%90%91%92%93%94%95%96%97%98%99%100%
## Confidence regions array returnedplot(cr)
#-----------------------------------------------------------------------
# Usando os valores internos da região de confiança conjunta.
# DANGER: existe preocupação com relação ao uso dessa abordagem. A
# primeira refere-se a cobertura deste IC. Erro padrão não deve ser
# calculado pois a distribuição dos valores é uniforme dentro da RC, por
# conta do método de simulação, e portanto não representam a
# distribuição do estimador.
mc_g <- apply(cr$cr,
MARGIN = 1,
FUN = function(th_i) {
eval(S_expr, envir = as.list(th_i))
})
range(mc_g)## [1] -0.06012402 -0.036280063 Inferência Bayesiana
3.1 Ajuste do modelo
#-----------------------------------------------------------------------
# Modelo para o Stan.
library(rstan)
stanmodel <- "
data {
int<lower=0> N;
real x[N];
real Y[N];
}
parameters {
real<lower = 0, upper = 1> Ur;
real<lower = 0, upper = 1> Us;
real alp;
real n;
real<lower=0> tau;
}
transformed parameters {
real sigma;
sigma = 1/sqrt(tau);
}
model {
real m[N];
for (i in 1:N){
m[i] = Ur+(Us-Ur)/pow(1+exp(n*(alp+x[i])), 1-1/n);
};
Y ~ normal(m, sigma);
Ur ~ uniform(0, 1);
Us ~ uniform(0, 1);
alp ~ normal(0, 1);
n ~ normal(2, 2);
tau ~ gamma(.0001, .0001);
}
"
coef(n0)## Ur Us alp n
## 0.3605170 0.5765758 -0.2693376 1.4397566# Ajusta o modelo.
fit <- stan(model_code = stanmodel,
model_name = "cra",
chains = 4,
iter = 500,
seed = 2020,
data = list(N = nrow(cra),
x = cra$ltens,
Y = cra$umid))##
## SAMPLING FOR MODEL 'cra' NOW (CHAIN 1).
## Chain 1:
## Chain 1: Gradient evaluation took 2.4e-05 seconds
## Chain 1: 1000 transitions using 10 leapfrog steps per transition would take 0.24 seconds.
## Chain 1: Adjust your expectations accordingly!
## Chain 1:
## Chain 1:
## Chain 1: Iteration: 1 / 500 [ 0%] (Warmup)
## Chain 1: Iteration: 50 / 500 [ 10%] (Warmup)
## Chain 1: Iteration: 100 / 500 [ 20%] (Warmup)
## Chain 1: Iteration: 150 / 500 [ 30%] (Warmup)
## Chain 1: Iteration: 200 / 500 [ 40%] (Warmup)
## Chain 1: Iteration: 250 / 500 [ 50%] (Warmup)
## Chain 1: Iteration: 251 / 500 [ 50%] (Sampling)
## Chain 1: Iteration: 300 / 500 [ 60%] (Sampling)
## Chain 1: Iteration: 350 / 500 [ 70%] (Sampling)
## Chain 1: Iteration: 400 / 500 [ 80%] (Sampling)
## Chain 1: Iteration: 450 / 500 [ 90%] (Sampling)
## Chain 1: Iteration: 500 / 500 [100%] (Sampling)
## Chain 1:
## Chain 1: Elapsed Time: 0.087421 seconds (Warm-up)
## Chain 1: 0.076284 seconds (Sampling)
## Chain 1: 0.163705 seconds (Total)
## Chain 1:
##
## SAMPLING FOR MODEL 'cra' NOW (CHAIN 2).
## Chain 2:
## Chain 2: Gradient evaluation took 1.1e-05 seconds
## Chain 2: 1000 transitions using 10 leapfrog steps per transition would take 0.11 seconds.
## Chain 2: Adjust your expectations accordingly!
## Chain 2:
## Chain 2:
## Chain 2: Iteration: 1 / 500 [ 0%] (Warmup)
## Chain 2: Iteration: 50 / 500 [ 10%] (Warmup)
## Chain 2: Iteration: 100 / 500 [ 20%] (Warmup)
## Chain 2: Iteration: 150 / 500 [ 30%] (Warmup)
## Chain 2: Iteration: 200 / 500 [ 40%] (Warmup)
## Chain 2: Iteration: 250 / 500 [ 50%] (Warmup)
## Chain 2: Iteration: 251 / 500 [ 50%] (Sampling)
## Chain 2: Iteration: 300 / 500 [ 60%] (Sampling)
## Chain 2: Iteration: 350 / 500 [ 70%] (Sampling)
## Chain 2: Iteration: 400 / 500 [ 80%] (Sampling)
## Chain 2: Iteration: 450 / 500 [ 90%] (Sampling)
## Chain 2: Iteration: 500 / 500 [100%] (Sampling)
## Chain 2:
## Chain 2: Elapsed Time: 0.085823 seconds (Warm-up)
## Chain 2: 0.0707 seconds (Sampling)
## Chain 2: 0.156523 seconds (Total)
## Chain 2:
##
## SAMPLING FOR MODEL 'cra' NOW (CHAIN 3).
## Chain 3:
## Chain 3: Gradient evaluation took 1.3e-05 seconds
## Chain 3: 1000 transitions using 10 leapfrog steps per transition would take 0.13 seconds.
## Chain 3: Adjust your expectations accordingly!
## Chain 3:
## Chain 3:
## Chain 3: Iteration: 1 / 500 [ 0%] (Warmup)
## Chain 3: Iteration: 50 / 500 [ 10%] (Warmup)
## Chain 3: Iteration: 100 / 500 [ 20%] (Warmup)
## Chain 3: Iteration: 150 / 500 [ 30%] (Warmup)
## Chain 3: Iteration: 200 / 500 [ 40%] (Warmup)
## Chain 3: Iteration: 250 / 500 [ 50%] (Warmup)
## Chain 3: Iteration: 251 / 500 [ 50%] (Sampling)
## Chain 3: Iteration: 300 / 500 [ 60%] (Sampling)
## Chain 3: Iteration: 350 / 500 [ 70%] (Sampling)
## Chain 3: Iteration: 400 / 500 [ 80%] (Sampling)
## Chain 3: Iteration: 450 / 500 [ 90%] (Sampling)
## Chain 3: Iteration: 500 / 500 [100%] (Sampling)
## Chain 3:
## Chain 3: Elapsed Time: 0.082767 seconds (Warm-up)
## Chain 3: 0.060732 seconds (Sampling)
## Chain 3: 0.143499 seconds (Total)
## Chain 3:
##
## SAMPLING FOR MODEL 'cra' NOW (CHAIN 4).
## Chain 4:
## Chain 4: Gradient evaluation took 1.1e-05 seconds
## Chain 4: 1000 transitions using 10 leapfrog steps per transition would take 0.11 seconds.
## Chain 4: Adjust your expectations accordingly!
## Chain 4:
## Chain 4:
## Chain 4: Iteration: 1 / 500 [ 0%] (Warmup)
## Chain 4: Iteration: 50 / 500 [ 10%] (Warmup)
## Chain 4: Iteration: 100 / 500 [ 20%] (Warmup)
## Chain 4: Iteration: 150 / 500 [ 30%] (Warmup)
## Chain 4: Iteration: 200 / 500 [ 40%] (Warmup)
## Chain 4: Iteration: 250 / 500 [ 50%] (Warmup)
## Chain 4: Iteration: 251 / 500 [ 50%] (Sampling)
## Chain 4: Iteration: 300 / 500 [ 60%] (Sampling)
## Chain 4: Iteration: 350 / 500 [ 70%] (Sampling)
## Chain 4: Iteration: 400 / 500 [ 80%] (Sampling)
## Chain 4: Iteration: 450 / 500 [ 90%] (Sampling)
## Chain 4: Iteration: 500 / 500 [100%] (Sampling)
## Chain 4:
## Chain 4: Elapsed Time: 0.087144 seconds (Warm-up)
## Chain 4: 0.064707 seconds (Sampling)
## Chain 4: 0.151851 seconds (Total)
## Chain 4:# length(fit)
# class(fit)
# str(fit)
methods(class = "stanfit")## [1] as.array as.data.frame as.matrix constrain_pars
## [5] dim dimnames extract get_cppo_mode
## [9] get_inits get_logposterior get_num_upars get_posterior_mean
## [13] get_seed get_seeds get_stancode get_stanmodel
## [17] grad_log_prob is.array log_prob loo_moment_match
## [21] loo names names<- pairs
## [25] plot print show summary
## [29] traceplot unconstrain_pars
## see '?methods' for accessing help and source code# Métodos.
# summary(fit) # Resumo de cada cadeia.
# plot(fit) # Boxplot para os parâmetros.
traceplot(fit) # Traço de cada cadeia.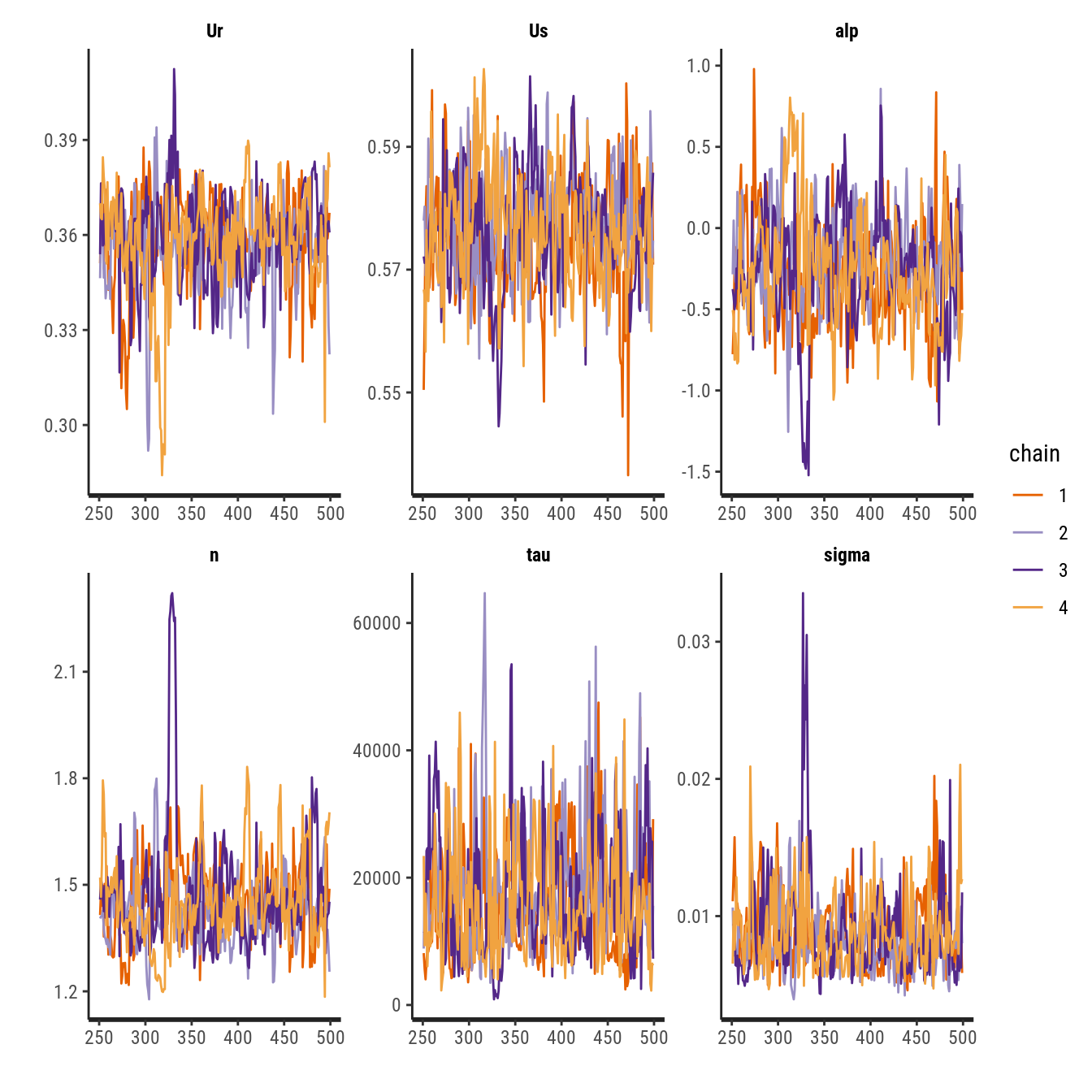
pairs(fit) # Matriz de diagramas de dispersão.
# Quantis.
show(fit)## Inference for Stan model: cra.
## 4 chains, each with iter=500; warmup=250; thin=1;
## post-warmup draws per chain=250, total post-warmup draws=1000.
##
## mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
## Ur 0.36 0.00 0.01 0.32 0.35 0.36 0.37 0.38 169 1.01
## Us 0.58 0.00 0.01 0.56 0.57 0.58 0.58 0.59 222 1.01
## alp -0.25 0.03 0.33 -0.87 -0.45 -0.26 -0.05 0.41 152 1.01
## n 1.45 0.01 0.13 1.25 1.38 1.44 1.50 1.72 154 1.02
## tau 17053.90 571.53 9252.23 4215.02 10188.78 15611.16 21788.53 39504.60 262 1.01
## sigma 0.01 0.00 0.00 0.01 0.01 0.01 0.01 0.02 176 1.02
## lp__ 39.99 0.17 2.13 34.81 38.78 40.43 41.55 42.76 155 1.02
##
## Samples were drawn using NUTS(diag_e) at Thu Mar 4 12:11:57 2021.
## For each parameter, n_eff is a crude measure of effective sample size,
## and Rhat is the potential scale reduction factor on split chains (at
## convergence, Rhat=1).cbind(summary(n0)$coeff, confint.default(n0))## Estimate Std. Error t value Pr(>|t|) 2.5 % 97.5 %
## Ur 0.3605170 0.006860092 52.552798 3.186065e-09 0.3470715 0.37396258
## Us 0.5765758 0.005199767 110.884932 3.626706e-11 0.5663844 0.58676714
## alp -0.2693376 0.185542216 -1.451624 1.968082e-01 -0.6329936 0.09431849
## n 1.4397566 0.057947850 24.845729 2.797466e-07 1.3261809 1.55333229summary(n0)$sigma## [1] 0.005039773logLik(n0)## 'log Lik.' 41.26869 (df=5)3.2 Inferência para uma função de parâmetros
# Distribuição a posteriori.
post <- as.data.frame(fit)
post <- cbind(expand.grid(KEEP.OUT.ATTRS = FALSE,
iter = 1:dim(fit)[1],
chain = 1:dim(fit)[2]),
post)
head(post)## iter chain Ur Us alp n tau sigma lp__
## 1 1 1 0.3510678 0.5504184 -0.7769515 1.414809 8146.775 0.01107917 36.53269
## 2 2 1 0.3653096 0.5625991 -0.6019667 1.485188 5660.427 0.01329154 38.46508
## 3 3 1 0.3636149 0.5784327 -0.2240589 1.441082 4031.372 0.01574975 37.80961
## 4 4 1 0.3696018 0.5835929 -0.1392925 1.497138 6812.774 0.01211541 39.25586
## 5 5 1 0.3493356 0.5689243 -0.1164449 1.354138 8803.245 0.01065807 39.99072
## 6 6 1 0.3616336 0.5719103 -0.4981739 1.502613 8375.534 0.01092682 40.29596# Inferência sobre o parâmetro S.
by_g <- apply(post[, names(coef(n0))],
MARGIN = 1,
FUN = function(th_i) {
eval(S_expr, envir = as.list(th_i))
})
c(mean = mean(by_g), median = median(by_g), sd = sd(by_g))## mean median sd
## -0.047039678 -0.046322968 0.005698393quantile(by_g, c(0.025, 0.975))## 2.5% 97.5%
## -0.05881039 -0.037880873.3 Bandas de confiança
#-----------------------------------------------------------------------
# Bandas de confiança para o ajuste.
meanCi <- function(x, conf = 0.975) {
alpha <- (1 - conf)/2
stat <- c(mean(x), quantile(x, c(alpha, 1 - alpha)))
names(stat) <- toupper(c("fit", "lwr", "upr"))
return(stat)
}
aux <- apply(post[, names(coef(n0))],
MARGIN = 1,
FUN = function(theta) {
eval(parse(text = as.character(model[3])),
envir = c(as.list(theta),
ltens = list(pred$ltens)))
})
pred <- cbind(pred[, 1:5], t(apply(aux, 1, FUN = meanCi)))
str(pred)## 'data.frame': 31 obs. of 8 variables:
## $ ltens: num -2.78 -2.43 -2.08 -1.73 -1.37 ...
## $ fit : num 0.576 0.575 0.574 0.573 0.571 ...
## $ se : num 0.005 0.00489 0.00472 0.00448 0.00415 ...
## $ lwr : num 0.564 0.563 0.563 0.562 0.561 ...
## $ upr : num 0.588 0.587 0.586 0.584 0.581 ...
## $ FIT : num 0.575 0.575 0.574 0.572 0.57 ...
## $ LWR : num 0.555 0.555 0.555 0.554 0.553 ...
## $ UPR : num 0.594 0.593 0.591 0.588 0.585 ...xyplot(umid ~ ltens, data = cra) +
as.layer(xyplot(fit + lwr + upr ~ ltens, data = pred,
type = "l", col = 1, lty = c(1, 2, 2))) +
as.layer(xyplot(FIT + LWR + UPR ~ ltens, data = pred,
type = "l", col = "red", lty = c(1, 2, 2)))
#-----------------------------------------------------------------------|
Modelos de Regressão Não Linear: Fundamentos e Aplicações em R leg.ufpr.br/~walmes/cursoR/mrnl |
Prof. Walmes M. Zeviani Departamento de Estatística · UFPR |
