Capítulo 4 Polinómios locais
Os estimadores de kernel sofrem de viés de limite e viés de projeto. Esses problemas podem ser aliviados usando uma generalização da regressão kernel chamada de regressão polinomial local.
Para motivar este estimador, primeiro considere escolher um estimador \(a = \widehat{r}_n(x)\) para minimizar as somas de quadrados \(\sum_{i=1}^n (Y_i-a)^2\). A solução é a função constante \(\widehat{r}_n(x)=\overline{Y}\) que obviamente não é um bom estimador de \(r(x)\). Agora defina a função de peso \(w_i(x)=K\big((x_i-x)/h\big)\) e escolha um \(a = \widehat{r}_n(x)\) para minimizar as somas de quadrados ponderadas \[\begin{equation} \sum_{i=1}^n w_i(x)\big(Y_i-a\big)^2\cdot \end{equation}\] Do cálculo, vemos que a solução é \[\begin{equation} \widehat{r}_n(x) \, = \, \frac{\displaystyle\sum_{i=1}^n w_i(x)Y_i}{\displaystyle\sum_{i=1}^n w_i(x)}, \end{equation}\] que é exatamente o estimador de regressão kernel. Isso nos fornece uma interpretação interessante do estimador kernel: este é um estimador localmente constante, obtido a partir de mínimos quadrados ponderados localmente.
Isso sugere que podemos melhorar o estimador usando um polinômio local de grau \(p\) em vez de uma constante local. Seja \(x\) algum valor fixo no qual queremos estimar \(r(x)\). Para valores \(u\) em uma vizinhança de \(x\), defina o polinômio \[\begin{equation} P_x(u;a) \, = \, a_0 \, + \, a_1(u-x) \, + \, \frac{a_2}{2!}(u-x)^2 \, + \, \cdots \, + \frac{a_p}{p!}(u-x)^p\cdot \end{equation}\] Podemos aproximar uma função de regressão suave \(r(u)\) em uma vizinhança do valor alvo \(x\) pelo polinômio: \[\begin{equation} r(u) \, = \, P_x(u;a)\cdot \end{equation}\] Estimamos \(a = (a_0,\cdots, a_p)^\top\) escolhendo \(\widehat{a} = (\widehat{a}_0,\cdots,\widehat{a}_p)^\top\) que minimize a soma de quadrados ponderadas localmente \[\begin{equation} \sum_{i=1}^n w_i(x)\big( Y_i \, - \, P_x(X_i;a)\big)^2\cdot \end{equation}\]
O estimador \(\widehat{a}\) depende do valor alvo \(x\), então escrevemos \(\widehat{a}(x) = (\widehat{a}_0(x),\cdots,\widehat{a}_p(x))^\top\) se quisermos tornar essa dependência explícita. A estimativa local de \(r\) é \[\begin{equation} \widehat{r}_n(x) \, = \, P_x(u;\widehat{a})\cdot \end{equation}\] Em particular, no valor alvo \(u = x\) temos \[\begin{equation} \widehat{r}_n(x) \, = \, P_x(x;\widehat{a}) \, = \, \widehat{a}_0(x)\cdot \end{equation}\] Embora \(\widehat{r}_n(x)\) dependa apenas de \(\widehat{a}_0(x)\), isso não é equivalente a simplesmente ajustar uma constante local.
Por exemplo, caso \(p=0\) retornamos ao estimador kernel. O caso especial em que \(p=1\) é chamado de regressão linear local e esta é a versão que recomendamos como uma opção padrão. Como veremos, estimadores polinomiais locais e, em particular, estimadores lineares locais possuem algumas propriedades notáveis como mostrado por Fan (1992) e Hastie and Loader (1993). Muitos dos resultados que se seguem são desses documentos.
Para encontrar \(\widehat{a}(x)\), será útil re-expressar o problema em notação vetorial. Definamos \[\begin{equation} X_x \, = \, \begin{pmatrix} 1 & (x_1-x) & \cdots & \frac{(x_1-x)^p}{p!} \\ 1 & (x_2-x) & \cdots & \frac{(x_2-x)^p}{p!} \\ \vdots & \vdots & \ddots & \vdots \\ 1 & (x_n-x) & \cdots & \frac{(x_n-x)^p}{p!} \end{pmatrix} \end{equation}\] e seja \(W_x\) a matriz diagonal \(n\times n\) cujo componente \((i, i)\) é \(w_i(x)\). Podemos então reescrever a soma de quadrados ponderadas localmente como \[\begin{equation} (Y-X_xa)^\top W_x(Y-X_xa)\cdot \end{equation}\]
Minimizando esta expressão fornece-nos o estimador de mínimos quadrados ponderados \[\begin{equation} \widehat{a}(x) \, = \, (X_x^\top W_xX_x)^{-1}X_xW_xY\cdot \end{equation}\] Em particular, \(\widehat{r}_n(x) =\widehat{a}_0(x)\) é o produto interno da primeira linha de \((X_x^\top W_xX_x)^{-1}X_xW_xY\) com \(Y\). Assim nós temos o seguinte teorema.
Teorema 4. A estimativa da regressão polinomial local é \[\begin{equation} \widehat{r}_n(x) \, = \, \sum_{i=1}^n \mathcal{l}_i(x)Y_i, \end{equation}\] onde \(\mathcal{l}(x)^\top = (\mathcal{l}_1(x),\cdots,\mathcal{l}_n(x))\), \[\begin{equation} \mathcal{l}^\top \, = \, e_1^\top (X_x^\top W_xX_x)^{-1}X_x^\top W_x, \end{equation}\] \(e_1=(1,0,\cdots,0)^\top\) e \(X_x\) e \(W_x\) como definidos anteriormente. Este estimador têm por média \[\begin{equation} \mbox{E}\big( \widehat{r}_n(x)\big) \, = \, \sum_{i=1}^n \mathcal{l}_i(x)r(x_i) \end{equation}\] e variância \[\begin{equation} \mbox{Var}\big(\widehat{r}_n(x)\big) \, = \, \sigma^2\sum_{i=1}^n \mathcal{l}_i(x)^2 \, = \, \sigma^2||\mathcal{l}(x)||^2\cdot \end{equation}\]
Demonstração. Ver Scott (1992).
Mais uma vez, nossa estimativa é linear e podemos escolher a largura de banda por validação cruzada.
Exemplo 6.
Vamos utilizar novamente os dados do Exemplo 1 assim como o resultado do modelo de regressão linear paramétrico para mostrar, escolhendo diversos valores da largura de banda \(h\), a curva de regressão obtida como estimativa da regressão polinomial local.
Os dois primeiros gráficos são baseados em pequenas larguras de banda: a acima \(h=0.5\) e abaixo \(h=1.0\), para isto utilizamos a opção bw no comando locpol (Local Polynomial estimation) no pacote de funções homônimo.
library(locpol)
model.npar01 = locpol(logwage ~ age, data = cps71, kernel = gaussK, bw = 0.5)
par(mfrow = c(1,1), mar = c(4, 4, 1, 1))
plot(logwage ~ age, data = cps71, col = "blue", pch=19); grid()
lines(cps71$age, fitted.values(model.par), col="darkblue", lwd = 3)
lines(model.npar01$lpFit[,1], model.npar01$lpFit[,2], col = "red", lwd = 3)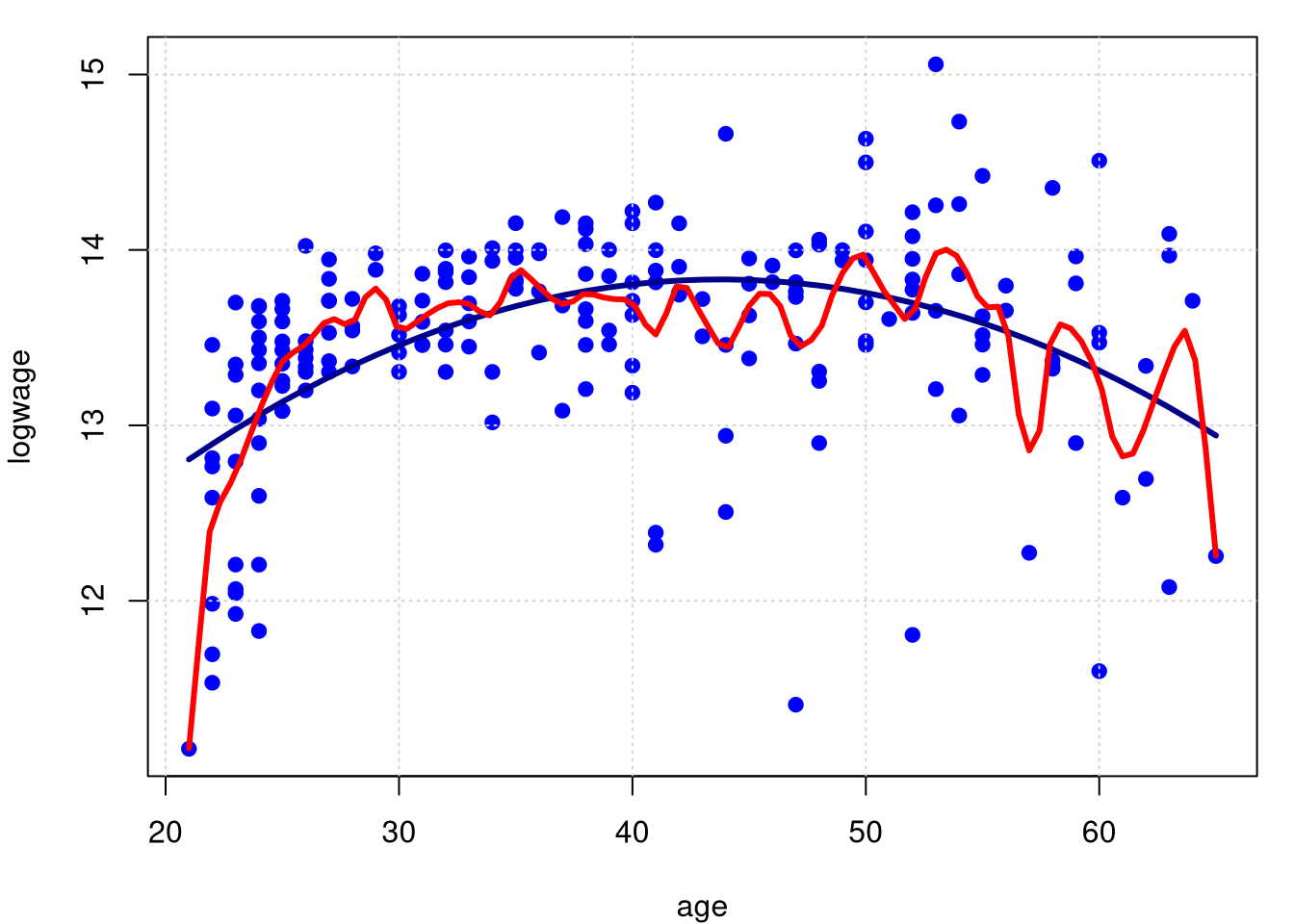
model.npar02 = locpol(logwage ~ age, data = cps71, kernel = gaussK, bw = 1.0)
par(mfrow = c(1,1), mar = c(4, 4, 1, 1))
plot(logwage ~ age, data = cps71, col = "blue", pch=19); grid()
lines(cps71$age, fitted.values(model.par), col="darkblue", lwd = 3)
lines(model.npar02$lpFit[,1], model.npar02$lpFit[,2], col = "red", lwd = 3)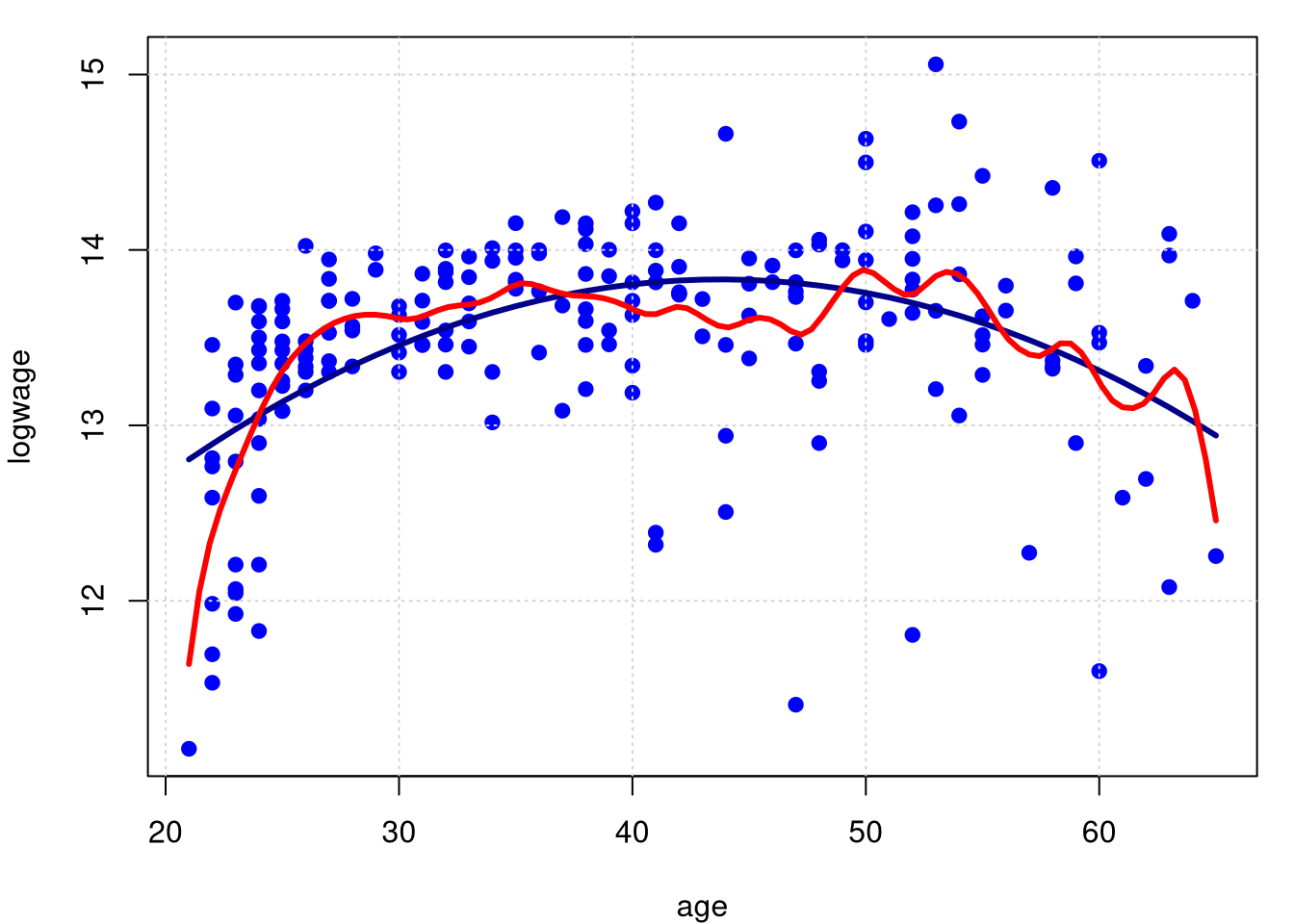
Agora essolhemos as larguras de banda \(h = 5.0\) (acima) e \(\widehat{h} = 1.49995\) (abaixo). Este valor obtido por validação cruzada. Observemos que, a medida que a largura de banda \(h\) aumenta, a função estimada passa de áspera demais para suave demais.
model.npar03 = locpol(logwage ~ age, data = cps71, kernel = gaussK, bw = 5.0)
par(mfrow = c(1,1), mar = c(4, 4, 1, 1))
plot(logwage ~ age, data = cps71, col = "blue", pch=19); grid()
lines(cps71$age, fitted.values(model.par), col="darkblue", lwd = 3)
lines(model.npar03$lpFit[,1], model.npar03$lpFit[,2], col = "red", lwd = 3)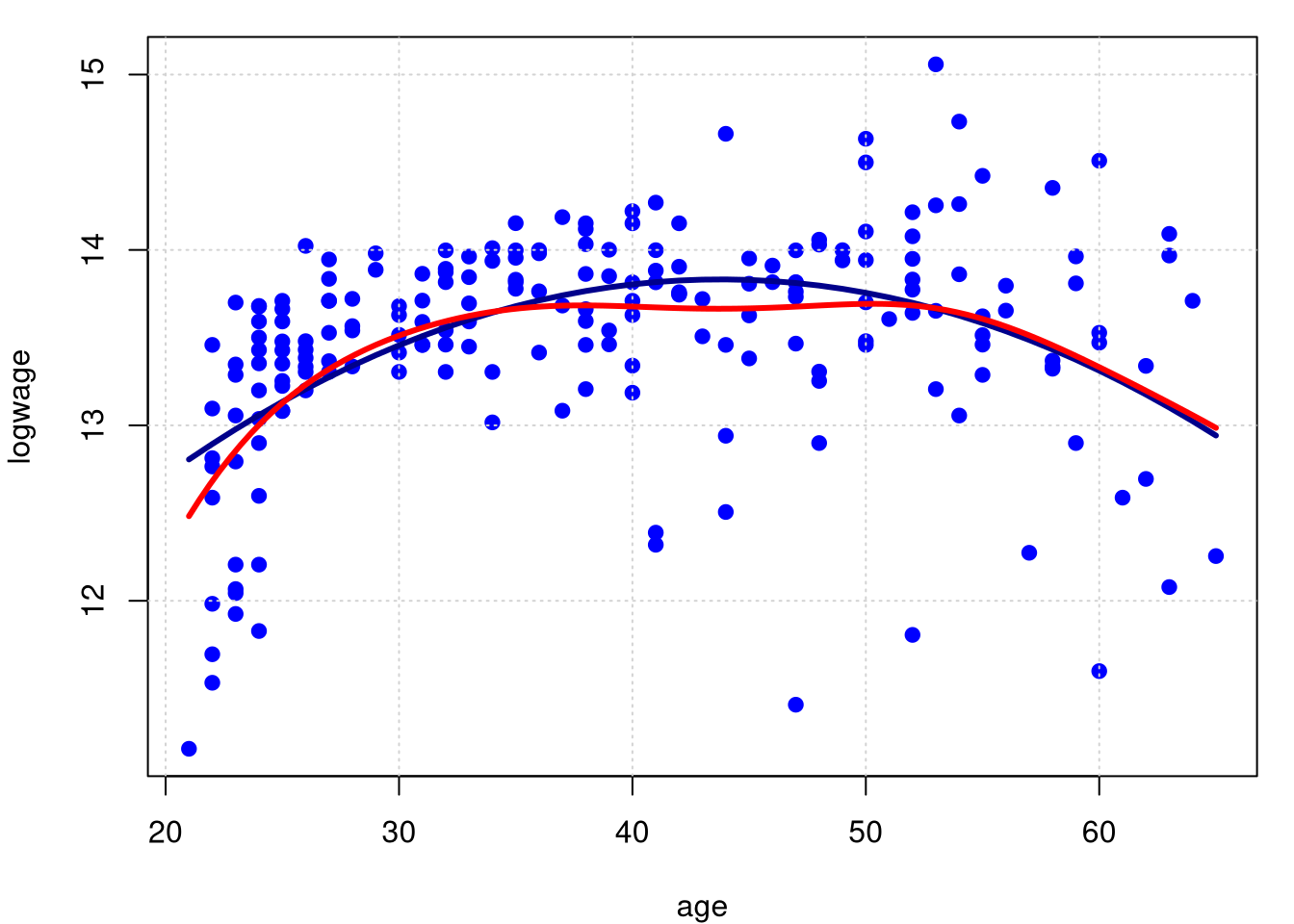
## [1] 1.49995model.npar04 = locpol(logwage ~ age, data = cps71, kernel = gaussK)
par(mfrow = c(1,1), mar = c(4, 4, 1, 1))
plot(logwage ~ age, data = cps71, col = "blue", pch=19); grid()
lines(cps71$age, fitted.values(model.par), col="darkblue", lwd = 3)
lines(model.npar04$lpFit[,1], model.npar04$lpFit[,2], col = "red", lwd = 3)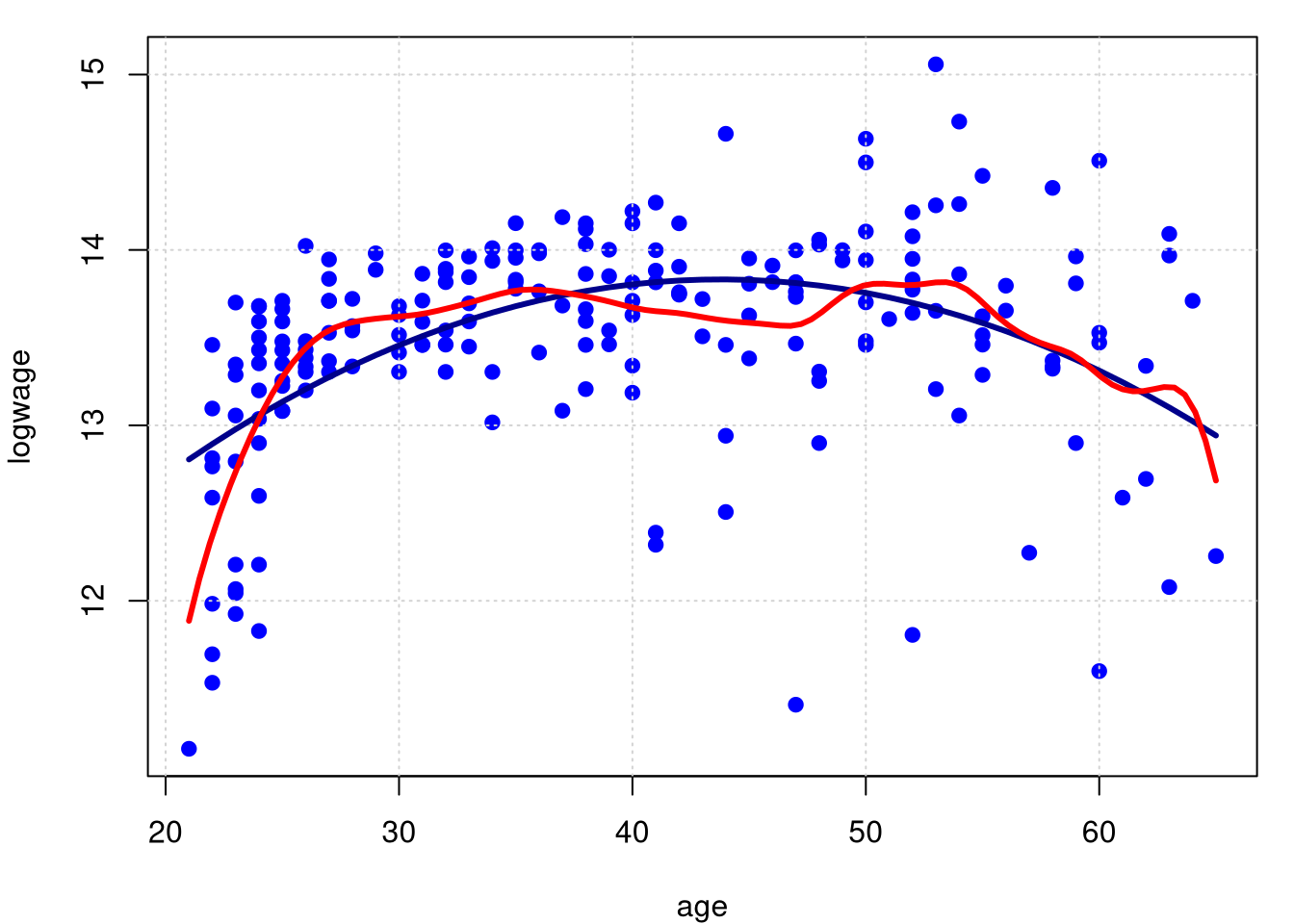
Nos últimos gráficos, o primeiro, baseado numa grande largura de banda \(h=5.0\) o ajuste é muito suave. O segundo gráfico, considerado correto, é àquele onde a largura de banda foi escolhida por validação cruzada. A função para obtermos \(\widehat{h}\) é regCVBwSelC.
Esta função Foi utilizada para mostrar o procedimento mas não é necessário em situações práticas, é o padrão, bastando não dizer nada como em model.npar04.
Este último gráfico também mostra a presença de viés perto dos limites. Como veremos, esta é uma característica geral da regressão kernel.
O teorema a seguir apresenta o comportamento amostral em amostras grandes do risco do estimador linear local e mostra por que a regressão linear local é melhor que a regressão kernel.
- \(f(x)>0\),
- \(f\), \(r''\) e \(\sigma^2\) sejam contínuas numa vizinhança de \(x\),
- \(h_n\to 0\) e \(nh_n\to \infty\).
Seja \(x \, \in \, (a,b)\). Dado \(X_1,\cdots,X_n\) temos o seguinte: o estimador linear local e o estimador kernel ambos têm variância \[\begin{equation} \frac{\sigma^2(x)}{f(x)nh_n}\int K^2(u)\mbox{d}u \, + \, o_P\Big( \frac{1}{nh_n}\Big) \cdot \end{equation}\] O estimador de kernel Nadaraya - Watson tem viés \[\begin{equation} h_n^2\left( \frac{1}{2}r''(x) \, + \, \frac{r'(x)f'(x)}{f(x)} \right)\int u^2K(u)\mbox{d}u \, + \, o_P( h^2) \end{equation}\] enquanto que o estimador linear local tem um viés assintótico \[\begin{equation} h_n^2\frac{1}{2}r''(x) \int u^2K(u)\mbox{d}u \, + \, o_P( h^2)\cdot \end{equation}\]
Demonstração. Fan (1992) e Fan and Gijbels (1996).
Assim, o estimador linear local é livre de viés de projeto, é livre de \(f\). Nos pontos de fronteira \(a\) e \(b\), o estimador kernel de Nadaraya-Watson tem um viés assintótico de ordem \(h_n\), enquanto o estimador linear local tem viés de ordem \(h_n^2\). Nesse sentido, a estimativa linear local elimina o viés nos limites.
Sabemos que o procedimento mais adequado, dentre os vistos até agora, é o de polinômios locais. Dediquemos atenção agora a um detalhe na escolha do parâmetro de alisamento \(h\) por validação cruzada. Dizemos que dentre os gráficos mostrados no Exemplo 5, o último deles é o correto e isso foi afirmado pelo fato de utilizarmos a validação cruzada para encontrarmos a estimativa do parámetro de alisamento. Mas, não é bem assim, a seleção por validação cruzada é um pouco mais complexa, como veremos no próximo exemplo.
Exemplo 7. Queremos entender a dependência entre a estimativa \(\widehat{h}\) da largura de banda e o intervalo onde procurar-la, também queremos obter o valor do \(R^2\) na regressão local. Para isso vamos explorar mais dois detalhes do procedimento escolhido para estimarmos a regressão polinomial local.
O primeiro gráfico dedica-se a pesquisar a relação entre o intervalo onde procurar a largura de banda e o valor da estimativa desta, ou seja, a estimativa de \(h\). Para isso definimos dois vetores do mesmo comprimento, sup e hn. No primeiro deles indicaremos o extremo superior dos intervalos onde procuraremos a estimativa da largura de banda, no segundo guardamos a estimativa da largura de banda obtida.
hn = sup = rep(0, 10)
for(i in 1:10) {sup[i] = 1.5*i}
for(i in 1:10){
hn[i] = regCVBwSelC(cps71$age, cps71$logwage, deg = 1,
kernel = gaussK, interval = c(0,sup[i]))
}
sup## [1] 1.5 3.0 4.5 6.0 7.5 9.0 10.5 12.0 13.5 15.0## [1] 1.499950 2.999952 3.268412 3.268412 3.268417 3.268412 3.268411 3.268422
## [9] 3.268414 3.268419Mostramos os valores escolhidos para os extremos dos intervalos e as estimativas de \(h\) encontradas e os comandos a seguir permitem-nos obter o gráfico conparativo destes.
par(mfrow = c(1,1), mar = c(3, 3, 1, 1))
plot(seq(0,15), seq(0,15), col = "blue", pch=19, type = "n", axes = FALSE); grid()
segments(0,1,sup[1],1)
axis(1)
for(i in 1:10) segments(0,i,sup[i],i)
abline(v=hn[3], col= "red")
text(5,11,expression(paste(hat(h)," = 3.268412")), col="red")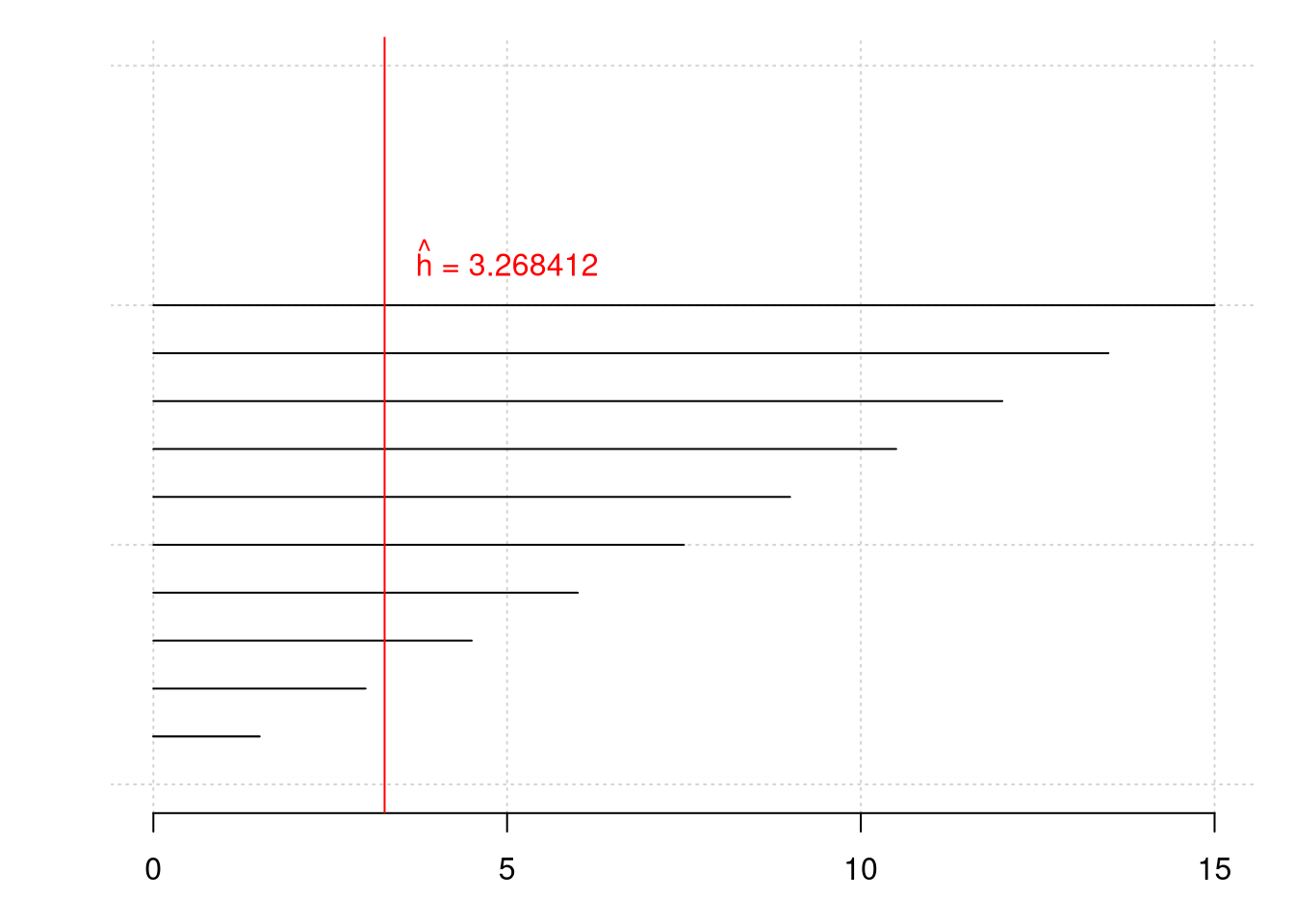
Podemos perceber que, caso não especifiquemos o intervalo de busca, podemos obter uma estimativa de \(h\) que não seja a correta. Conforme ampliamos o intervalo desta busca percebemos que os primeiros dois valores estimados correspondem ao limite superior, sendo isto um indicativo que devemos modificar o extremo superior do intervalo de busca.
Uma vez feito, encontramos o valor de \(\widehat{h}\) correto, ou seja, não importa quão mais amplo seja o intervalo de busca que a estimativa encontrada não muda. Encontramos então o polinômio local mais adequado à nossos dados.
model.npar05 = locpol(logwage ~ age, data = cps71, kernel = gaussK, deg = 1,
bw = hn[3], xevalLen = length(cps71$age))
par(mfrow = c(1,1), mar = c(4, 4, 1, 1))
plot(logwage ~ age, data = cps71, col = "blue", pch=19); grid()
lines(cps71$age, fitted.values(model.par), col="darkblue", lwd = 3)
lines(model.npar05$lpFit[,1], model.npar05$lpFit[,2], col = "red", lwd = 3)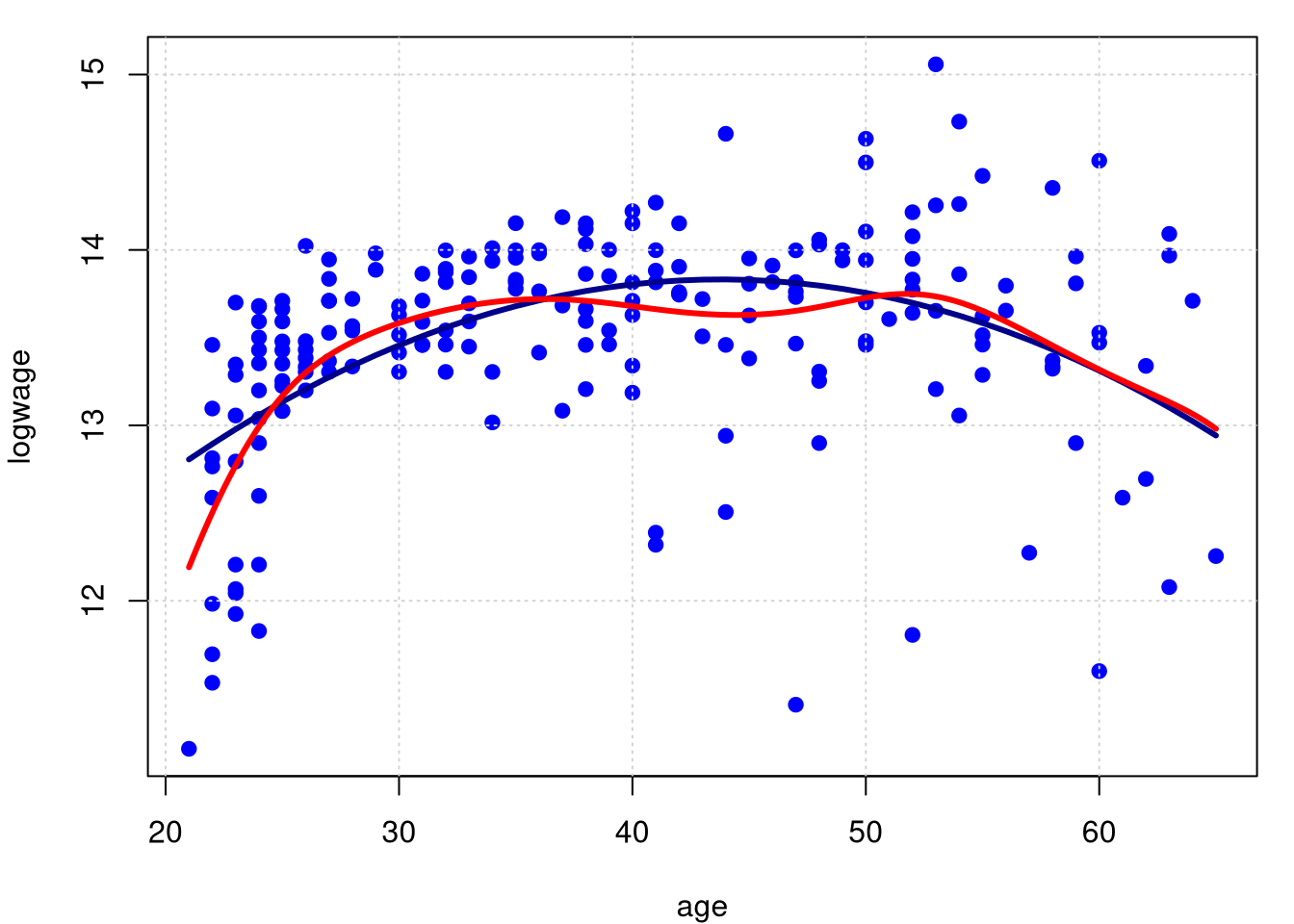
Resolvemos o primeiro problema, como termos certeza de que o valor obtido de \(\widehat{h}\) seja o correto. Queremos agora encontrar o valor do \(R^2\) nesta situação. Aconte que, por padrão, o comando locpol somente avalia em xevalLen = 100, ou seja, avalia em no máximo 100 pontos a curva estimada. Isto caso sejam mais do que 100 os valores da variável regressora.
Em nosso exemplo temos um total de
## [1] 205pontos amostrais.
Isso implica que seja necessário informar ao comando locpol que queremos tantos pontos estimados quantos pontos amostrais. Para isso utilizamos a opção xevalLen = length(cps71$age) e, dessa forma, indicamos que queremos tantas estimativas de \(\widehat{r}\) quantos dados amostrais. Podemos então avaliar na função R2.
R2 = function(Y,r){
media = mean(Y)
soma1 = sum((Y-media)^2)
soma2 = sum((r-media)^2)
soma0 = sum((Y-media)*(r-media))
R2 = (soma0^2)/(soma1*soma2)
return(R2)
}
R2(cps71$logwage,model.npar05$lpFit[,2])## [1] 0.2623865A função R2 foi criada por nós e tem por entrada os valores de resposta \(Y\) e de estimativas \(\widehat{r}\). Por resultado temos que \(R^2=0.26\) é o valor do coeficiente de determinação para a regressão polinomial local, valor ligeiramente superior àquele obtido com o modelo de regressão lineal.