Capítulo 6 Regressão múltipla
Suponhamos agora que a covariável seja de dimensão \(d\), ou seja, \[\begin{equation} x_i \, = \, (x_{i1},x_{i2},\cdots,x_{id} )^\top\cdot \end{equation}\] O modelo de regressão assume a forma \[\begin{equation} Y \, = \, r(x_{1},x_{2},\cdots,x_{d} ) \, + \, \epsilon\cdot \end{equation}\]Em princípio, todos os métodos que discutimos passam facilmente para este caso. Infelizmente, o risco de um estimador de regressão não paramétrico aumenta rapidamente com a dimensão \(d\). Em um problema unidimensional, a taxa ótima de convergência de um estimador não paramétrico é \(n^{−4/5}\) se \(r\) for assumido como tendo uma segunda derivada integrável.
Em \(d\) dimensões, a taxa ótima de convergência é \(n^{−4/(4 + d)}\). Assim, o tamanho da amostra \(m\) requerido para um problema \(d\)-dimensional ter a mesma precisão que um tamanho de amostra \(n\) em um problema unidimensional é \(m \propto n^{cd}\) onde \(c = (4 + d) / (5d)> 0\). Isto implica que para manter um determinado grau de precisão de um estimador, o tamanho da amostra deve aumentar exponencialmente com a dimensão \(d\). Em outras palavras, as bandas de confiança ficam muito grandes à medida que a dimensão \(d\) aumenta. No entanto, vamos continuar e ver como podemos estimar a função de regressão.
6.1 Regressão local
Considere a regressão linear local. A função kernel \(K\) é agora uma função de \(d\) variáveis. Dada uma matriz \(d \times d\) de largura de banda definida positiva não-singular \(H\), definimos \[\begin{equation} K_H \, = \, \frac{1}{|H|^{1/2}}K\big( H^{1/2}x\big)\cdot \end{equation}\] Muitas vezes, a escala de cada covariável tem a mesma média e variância e então usamos o kernel \[\begin{equation} h^{-k}K\big(||x||/h \big), \end{equation}\] onde \(K\) é qualquer kernel unidimensional. Então há um único parâmetro de largura de banda \(h\). Num valor alvo \(x = (x_1,\cdots, x_d)^\top\) a soma dos quadrados local é dada por \[\begin{equation} \sum_{i=1}^n \omega_i(x)\Big( Y_i \, - \, a_0 \sum_{j=1}^d a_j (x_{ij}-x_j)\Big)^2 \end{equation}\] onde \[\begin{equation} \omega_i(x) \, = \, K(||x_i-x||/h)\cdot \end{equation}\]
O estimador é \[\begin{equation} \widehat{r}_n(x) \, = \, \widehat{a}_0, \end{equation}\] onde \(\widehat{a}=(\widehat{a}_0,\cdots,\widehat{a}_d)^\top\) é o valor de \(a=(a_0,\cdots,a_d)^\top\) que minimiza a soma de quadrados ponderada. A solução \(\widehat{a}\) será \[\begin{equation} \widehat{a} \, = \, (X_x^\top W_xX_x)^{-1}X_x^\top W_xY \end{equation}\] onde \[\begin{equation} X_x \, = \, \begin{pmatrix} 1 & x_{11}-x_1 & \cdots & x_{1d}-x_d \\ 1 & x_{21}-x_1 & \cdots & x_{2d}-x_d \\ \vdots & \vdots & \ddots & \vdots \\ 1 & x_{n1}-x_1 & \cdots & x_{nd}-x_d \end{pmatrix} \end{equation}\] e \(W_x\) é a matriz diagonal cujo elemento \((i, i)\) é \(w_i(x)\).
As propriedades teóricas da regressão polinomial local em dimensões maiores são discutidas no seguinte teorema.
Teorema 12. Seja \(\widehat{r}_n(x)\) o estimador linear local multivariado com matriz de largura de banda \(H\) e assumimos satisfeitas as condições de regularidade:
O kernel \(K\) é limitado, de suporte compacto de tal modo que \(\int u u^\top K(u)\mbox{d}u=\mu_2(K)\mbox{I}\), onde \(\mu_2(K)\neq 0\) é escalar e \(\mbox{I}\) é matriz identidade \(d \times d\). Além disso, todos os momentos de ordem ímpar de \(K\) somem, isto é, \(\int u_1^{l_1}\cdots u_d^{l_d}K(u)\mbox{d}u=0\) para todos os inteiros não negativos \(l_1,\cdots,l_d\), de tal forma que sua soma seja ímpar. Esta última condição é satisfeita por kernels esfêricos simétricos e kernels de produtos baseados em kernels univariados simétricos.
O ponto \(x\) está no suporte de \(f\). Em \(x\), \(\nu\) é contínua, \(f\) é continuamente diferenciável e todas as derivadas de segunda ordem de \(r\) são contínuas. Além disso, \(f(x)> 0\) e \(\nu(x)> 0\).
A sequência de matrizes de largura de banda \(H^{1/2}\) é tal que \(n^{-1}|H|\) e cada entrada de \(H\) tende a zero quando \(n\to\infty\) com \(H\) permanecendo simétrica e definida positiva. Além disso, existe uma constante fixa \(L\) tal que a razão de seu maior e menor autovalor é, no máximo, \(L\) para todo \(n\).
O viés de \(\widehat{r}_n(x)\) é \[\begin{equation} \frac{1}{2}\mu_2(K)\mbox{tr}\big( H\mathcal{H}\big)+o_P(\mbox{tr}(H)), \end{equation}\] onde \(\mathcal{H}\) é a matriz das segundas derivadas parciais de \(r\) avaliada em \(x\) e \(\mu_2(K)\) é o escalar definido pelas condições de regularidade no item a.
A variância de \(\widehat{r}_n(x)\) é \[\begin{equation} \frac{\sigma^2(x)\int K(u)^2\mbox{d}u}{n|H|^{1/2}f(x)}\big( 1+o_P(1)\big)\cdot \end{equation}\] Além disso, o viés no limite é da mesma ordem que no interior, ou seja, \(O_P(\mbox{tr}(H))\).
Demonstração. Ruppert and Wand (1994).
Ao estudarmos a regressão local conhecemos duas formas de avaliarmos estes estimadores: o estimador kernel Nadaraya–Watson e os polinômios locais. No Teorema 5 mostramos que a regressão linear local é melhor que a regressão kernel.
Acontece que, pelo menos até o nosso conhecimento, os procedimentos implementados no R somente permitem a estimação do modelo de regressão por polinômios locais até ordem 2, ou seja, somente aceitam no máximo duas covariáveis contínuas.
Nos referimos especificamente ao pacote de funções locpol. Significa que, para aplicarmos modelos de regressão não paramétrica a situações mais complexas devemos recorrer ao estimador kernel, muito bem implementado no pacote de funções np e descrito no artigo de Hayfield and Racine (2008).
O pacote np implementa uma variedade de estimadores não paramétricos e semiparamétricos baseados no estimador kernel Nadaraya–Watson. Existem também procedimentos para testes de significância não paramétricos e testes consistentes de especificação de modelo.
Este pacote foca nos métodos apropriados para a combinação de dados contínuos, discretos e categóricos, frequentemente encontrados em problemas aplicados. Os métodos de seleção de largura de banda orientados por dados são enfatizados, embora tenhamos cautela já que estes procedimentos podem ser exigentes em termos computacionais.
Exemplo 13.
Dados britânicos de corte transversal, consiste de uma amostra aleatória extraída do Inquérito às Famílias da Família Britânica de 1995.
Os agregados familiares consistem em casais casados com um chefe de família empregado entre os 25 e os 55 anos de idade. Existem 1655 observações ao nível do agregado familiar no total.
## food catering alcohol fuel motor fares leisure logexp logwages nkids
## 1 0.14949 0.10460 0.00000 0.07555 0.1940 0.03446 0.16236 4.878 5.533 0
## 2 0.36461 0.03262 0.01533 0.09762 0.1994 0.03679 0.13521 5.094 5.371 0
## 3 0.21042 0.06525 0.07919 0.03367 0.2526 0.01002 0.17814 5.782 6.001 0
## 4 0.07869 0.11527 0.07014 0.05842 0.0000 0.24974 0.02726 5.756 5.861 0
## 5 0.28244 0.05508 0.07505 0.15707 0.2178 0.00000 0.03754 5.280 6.569 0
## 6 0.16636 0.15928 0.17427 0.02050 0.0907 0.00000 0.14901 5.822 5.879 0Esta aqui é uma ilustração simples para ajudá-lo a começar com a regressão do Kernel multivariada e o gráfico através da função gráfica R que chama npplot, no mesmo pacote np.
Engel95 = Engel95[order(Engel95$logexp),]
attach(Engel95)
modelo.iv = npregiv(y=food, z=logexp, w=logwages, method="Landweber-Fridman")## Computing bandwidths and E(y|w) for stopping rule... Computing bandwidths and E(y|z) for iteration 0... Computing bandwidths and E[y-phi(z)|w] for iteration 1... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 1... Computing bandwidths and E[y-phi(z)|w] for iteration 2... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 2... Computing stopping rule for iteration 2... Computing bandwidths and E[y-phi(z)|w] for iteration 3... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 3... Computing stopping rule for iteration 3... Computing bandwidths and E[y-phi(z)|w] for iteration 4... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 4... Computing stopping rule for iteration 4... Computing bandwidths and E[y-phi(z)|w] for iteration 5... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 5... Computing stopping rule for iteration 5... Computing bandwidths and E[y-phi(z)|w] for iteration 6... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 6... Computing stopping rule for iteration 6... Computing bandwidths and E[y-phi(z)|w] for iteration 7... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 7... Computing stopping rule for iteration 7... Computing bandwidths and E[y-phi(z)|w] for iteration 8... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 8... Computing stopping rule for iteration 8... Computing bandwidths and E[y-phi(z)|w] for iteration 9... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 9... Computing stopping rule for iteration 9... Computing bandwidths and E[y-phi(z)|w] for iteration 10... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 10... Computing stopping rule for iteration 10... Computing bandwidths and E[y-phi(z)|w] for iteration 11... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 11... Computing stopping rule for iteration 11... Computing bandwidths and E[y-phi(z)|w] for iteration 12... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 12... Computing stopping rule for iteration 12... Computing bandwidths and E[y-phi(z)|w] for iteration 13... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 13... Computing stopping rule for iteration 13... Computing bandwidths and E[y-phi(z)|w] for iteration 14... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 14... Computing stopping rule for iteration 14... Computing bandwidths and E[y-phi(z)|w] for iteration 15... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 15... Computing stopping rule for iteration 15... Computing bandwidths and E[y-phi(z)|w] for iteration 16... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 16... Computing stopping rule for iteration 16... Computing bandwidths and E[y-phi(z)|w] for iteration 17... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 17... Computing stopping rule for iteration 17... Computing bandwidths and E[y-phi(z)|w] for iteration 18... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 18... Computing stopping rule for iteration 18... Computing bandwidths and E[y-phi(z)|w] for iteration 19... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 19... Computing stopping rule for iteration 19... Computing bandwidths and E[y-phi(z)|w] for iteration 20... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 20... Computing stopping rule for iteration 20... Computing bandwidths and E[y-phi(z)|w] for iteration 21... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 21... Computing stopping rule for iteration 21... Computing bandwidths and E[y-phi(z)|w] for iteration 22... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 22... Computing stopping rule for iteration 22... Computing bandwidths and E[y-phi(z)|w] for iteration 23... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 23... Computing stopping rule for iteration 23... Computing bandwidths and E[y-phi(z)|w] for iteration 24... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 24... Computing stopping rule for iteration 24... Computing bandwidths and E[y-phi(z)|w] for iteration 25... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 25... Computing stopping rule for iteration 25... Computing bandwidths and E[y-phi(z)|w] for iteration 26... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 26... Computing stopping rule for iteration 26... Computing bandwidths and E[y-phi(z)|w] for iteration 27... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 27... Computing stopping rule for iteration 27... Computing bandwidths and E[y-phi(z)|w] for iteration 28... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 28... Computing stopping rule for iteration 28... Computing bandwidths and E[y-phi(z)|w] for iteration 29... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 29... Computing stopping rule for iteration 29... Computing bandwidths and E[y-phi(z)|w] for iteration 30... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 30... Computing stopping rule for iteration 30... Computing bandwidths and E[y-phi(z)|w] for iteration 31... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 31... Computing stopping rule for iteration 31... Computing bandwidths and E[y-phi(z)|w] for iteration 32... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 32... Computing stopping rule for iteration 32... Computing bandwidths and E[y-phi(z)|w] for iteration 33... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 33... Computing stopping rule for iteration 33... Computing bandwidths and E[y-phi(z)|w] for iteration 34... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 34... Computing stopping rule for iteration 34... Computing bandwidths and E[y-phi(z)|w] for iteration 35... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 35... Computing stopping rule for iteration 35... Computing bandwidths and E[y-phi(z)|w] for iteration 36... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 36... Computing stopping rule for iteration 36... Computing bandwidths and E[y-phi(z)|w] for iteration 37... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 37... Computing stopping rule for iteration 37... Computing bandwidths and E[y-phi(z)|w] for iteration 38... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 38... Computing stopping rule for iteration 38... Computing bandwidths and E[y-phi(z)|w] for iteration 39... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 39... Computing stopping rule for iteration 39... Computing bandwidths and E[y-phi(z)|w] for iteration 40... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 40... Computing stopping rule for iteration 40... Computing bandwidths and E[y-phi(z)|w] for iteration 41... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 41... Computing stopping rule for iteration 41... Computing bandwidths and E[y-phi(z)|w] for iteration 42... Computing bandwidths and E[E(y-phi(z)|w)|z] for iteration 42... Computing stopping rule for iteration 42... Calculando a regressão IV, ou seja, regressão de y em z.
## Multistart 1 of 1 |Multistart 1 of 1 |Multistart 1 of 1 |Multistart 1 of 1 /Multistart 1 of 1 |Multistart 1 of 1 | Para os gráficos, restrinjimos a atenção à maior parte dos dados, isto é, para a área de gráfico, cortamos 1/4 de um por cento de cada cauda de \(y\) em \(z\).
trim <- 0.0025
plot(logexp,food,
ylab="Compartilhamento Orçamentário de Alimentos",
xlab="log(Despesa total)",
xlim=quantile(logexp,c(trim,1-trim)),
ylim=quantile(food,c(trim,1-trim)),
main="Regressão Não Paramétrica Kernel Instrumental",
type="p",
cex=.5,
col="lightgrey")
lines(logexp,phihat,col="blue",lwd=2,lty=2)
lines(logexp,fitted(ghat),col="red",lwd=2,lty=4)
legend(quantile(logexp,trim),quantile(food,1-trim),
c(expression(paste("Não Paramétrica IV: ",hat(varphi)(logexp))),
"Regressão Não Paramétrica : E(food | logexp)"),
lty=c(2,4),
col=c("blue","red"),
lwd=c(2,2))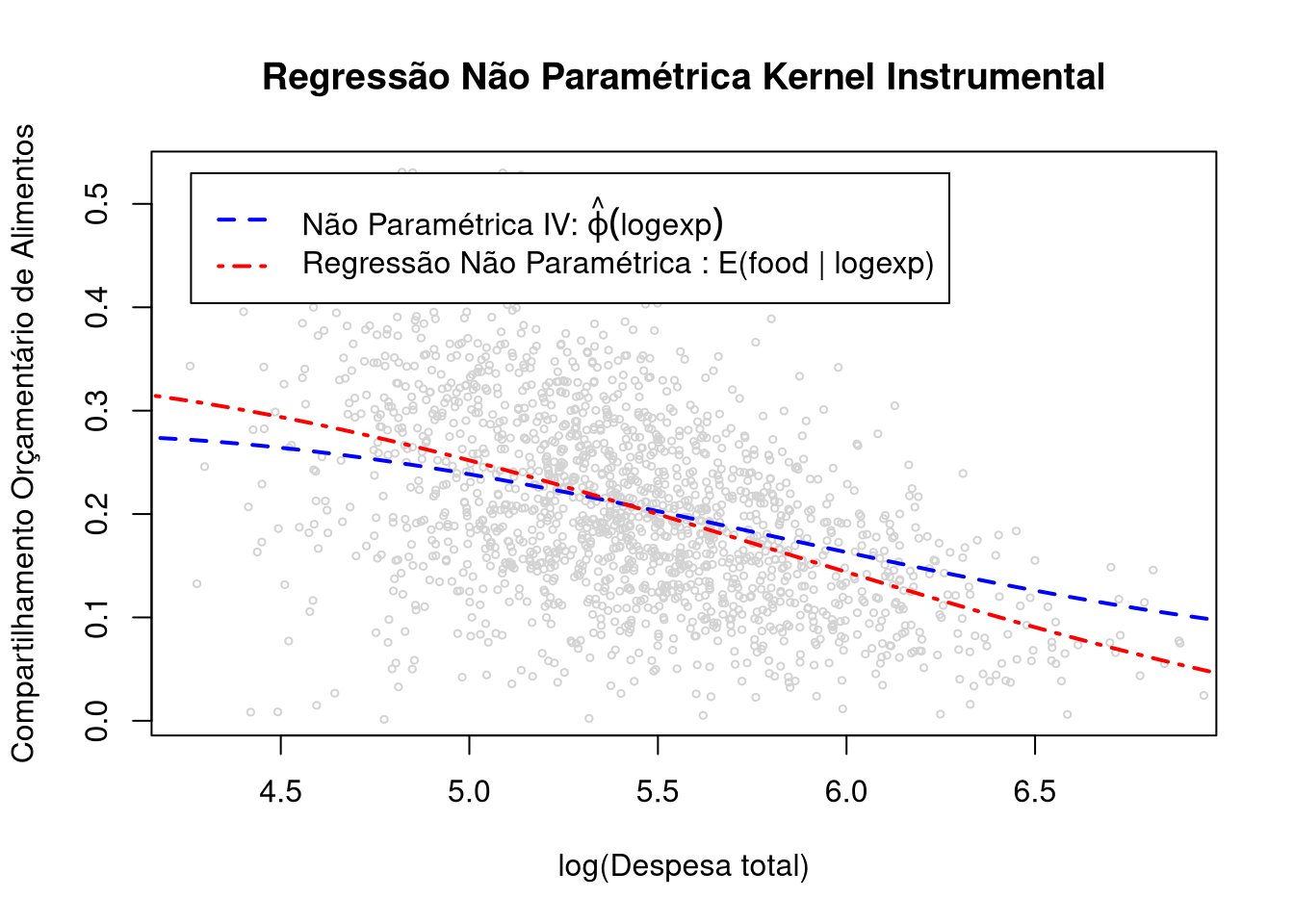
Assim, vemos que em dimensões mais altas, a regressão linear local ainda evita excessivo viés de limite e viéss de design.
6.2 Modelos aditivos
Interpretar e visualizar um ajuste de alta dimensão é difícil. À medida que o número de covariáveis aumenta, a carga computacional torna-se proibitiva. À vezes, uma abordagem mais frutífera é usar um modelo aditivo. Consideramos um modelo como aditivo se assumir a forma \[\begin{equation} Y \, = \, \mu \, + \, \sum_{j=1}^d r_j(x_i) \, + \, \epsilon, \end{equation}\] onde \(r_1,\cdots,r_d\) são funções suaves.
Acontece que este modelo não é identificável, pois podemos adicionar qualquer constante a \(\mu\) e subtrair a mesma constante de um dos \(r_j\) sem alterar a funçção de regressão. Este problema pode ser corrigido de várias maneiras, talvez o mais fácil seja definir \(\widehat{\mu}=\overline{Y}\) e, em seguida, considerar os \(r_j\) como desvios de \(\overline{Y}\). Neste caso, exigimos que \[\begin{equation} \sum_{i=1}^n r_j(x_i) \, = \, 0, \end{equation}\] para cada \(j\).
O modelo aditivo claramente não é tão geral quanto estimar \(r(x_1,\cdots, x_d)\), mas é muito mais simples de ser computado e interpretado e, portanto, é geralmente um bom ponto de partida. Apresentamos um algoritmo simples para transformar qualquer regressão unidimensional mais suave em um método para ajustar modelos aditivos, chamado de backfitting ou adaptação.
ALGORITMO DE ADAPTAÇÃO
- Passo 1: Inicialização: seja \(\widehat{\mu}=\overline{Y}\), e considere valores inciais para \(\widehat{r}_1,\cdots,\widehat{r}_d\).
-
Pssso 2: Iterar até a convergência: para \(j=1,\cdots,d\)
- Calcular \[\begin{equation} \widetilde{Y}_i \, = \, Y_i - \widehat{\mu} \, - \, \sum_{k\neq j} r_k(x_i), \end{equation}\] para \(i=1,\cdots,n\).
- Aplicar o alisamento a \(\widetilde{Y}_i\) no \(x_j\) para obter \(\widehat{r}_j\).
- Atribuir \(\widehat{r}_j\) igual a \[\begin{equation} \widehat{r}_j(x) - \frac{1}{n}\sum_{i=1}^n \widehat{r}_j(x_i)\cdot \end{equation}\]
Uma forma de implementar este algoritmo está disponível para modelos de regressão linear múltipla aplicando splines a somente uma covarável, isso não significa que poderemos aplicar splines a somente uma covariável por vez. Significa que aplicaremos splines undimensionais a uma ou várias covariáveis isoladamente. Veja no seguinte exemplo a forma de utilizarmos o comando ns dentro do pacote de funções splines.
Exemplo 14.
Utilizaremos os dados em mtcars, disponíveis na base do R. Estes dados foram extraídos da revista Motor Trend nos Estados Unidos de 1974 e compreendem o consumo de combustível e 10 aspectos do projeto e desempenho de 32 automóveis modelos de 1973 a 1974.
Interessa-nos como reposta mpg ou milhas/galão em dólares americanos. Como covariáveis temos gear ou a engrenagem contando o número de marchas para a frente, a qual é discreta ou um fator; wt, o peso em 1000 libras e hp, a potência bruta do motor.
library(splines)
mult.spline1 = lm(mpg ~ factor(gear) + ns(wt) + hp, data = mtcars)
summary(mult.spline1)##
## Call:
## lm(formula = mpg ~ factor(gear) + ns(wt) + hp, data = mtcars)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.3025 -1.9307 -0.3722 1.0243 5.9784
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 29.97256 1.84066 16.284 1.74e-15 ***
## factor(gear)4 1.26490 1.34084 0.943 0.3539
## factor(gear)5 1.87356 1.86662 1.004 0.3244
## ns(wt) -15.79711 4.28188 -3.689 0.0010 **
## hp -0.03497 0.01260 -2.775 0.0099 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.619 on 27 degrees of freedom
## Multiple R-squared: 0.8356, Adjusted R-squared: 0.8112
## F-statistic: 34.3 on 4 and 27 DF, p-value: 3.196e-106.3 Splines bidimensionais
Nesta abordagem precisamos definir splines em dimensões maiores. Para \(d = 2\), minimizamos \[\begin{equation} \sum_i \big( Y_i \, - \, \widehat{r}_n(x_{i1},x_{i2})\big)^2 \, + \, \lambda \, J(r), \end{equation}\] onde \[\begin{equation} J(r) \, = \, \int\int\left[ \left(\frac{\partial^2 r(x)}{\partial x_1^2}\right) \, + \, 2\left(\frac{\partial^2 r(x)}{\partial x_1\partial x_2}\right) \, + \, \left(\frac{\partial^2 r(x)}{\partial x_2^2}\right)\right] \mbox{d}x_i\mbox{d}x_2\cdot \end{equation}\]
O minimizador obtido \(\widehat{r}_n\) é chamado spline de placa fina. É difícil descrever e ainda mais difícil, mas certamente não impossível, de ajustar. Veja Green and Silverman (1994) para mais detalhes.
Exemplo 15.(Continuação do Exemplo 14)
Utilizando os mesmos dados, podemos considerar interações entre as variáveis wt e hp, agora do tipo contínuas e aplicados aplines undimensionais a cada uma separadamente.
library(splines)
mult.spline2 = lm(mpg ~ factor(gear) + ns(wt)*ns(hp), data = mtcars)
summary(mult.spline2)##
## Call:
## lm(formula = mpg ~ factor(gear) + ns(wt) * ns(hp), data = mtcars)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.7324 -1.7140 -0.5176 1.4920 4.5603
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 32.103 1.758 18.266 2.36e-16 ***
## factor(gear)4 0.829 1.118 0.741 0.46505
## factor(gear)5 1.817 1.548 1.174 0.25114
## ns(wt) -29.871 5.245 -5.695 5.43e-06 ***
## ns(hp) -28.974 5.867 -4.938 3.95e-05 ***
## ns(wt):ns(hp) 47.236 12.961 3.645 0.00117 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.171 on 26 degrees of freedom
## Multiple R-squared: 0.8912, Adjusted R-squared: 0.8702
## F-statistic: 42.58 on 5 and 26 DF, p-value: 1.035e-11Ou podemos utilizar o comando s no pacote mgcv com o qual podemos ajutar splines bidimensionais.
library(mgcv)
mult.spline2 = gam(mpg ~ factor(gear) + s(wt,hp), data = mtcars)
summary(mult.spline2)##
## Family: gaussian
## Link function: identity
##
## Formula:
## mpg ~ factor(gear) + s(wt, hp)
##
## Parametric coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 19.6247 0.7288 26.926 <2e-16 ***
## factor(gear)4 0.4072 1.2821 0.318 0.754
## factor(gear)5 2.0046 1.6312 1.229 0.231
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Approximate significance of smooth terms:
## edf Ref.df F p-value
## s(wt,hp) 5.059 6.299 16.79 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## R-sq.(adj) = 0.868 Deviance explained = 89.8%
## GCV = 6.4313 Scale est. = 4.8116 n = 32Perceba que, desta última forma, tivemos um ganho real no ajuste. O \(R^2\) ajustado é de 0.898. Lembremos que estamos trabalhando com modelos de regressão, com isso a melhor maneira de descubrirmos se estamos realizaddo um ajuste adequado é analisando os resíduos. Para isso, qualquer seja o modelos escolhido, sempre é possível utilizarmos o comando residuals e mostrar gráficos e testes adequados.
6.4 Prospeção de projeção
Vamos supor que o vetor de variáveis explicativas \(X = (x_1,\cdots, x_p)\) seja de alta dimensão. O modelo aditivo \[\begin{equation} Y = \mu + \sum_{j=1}^p f_j(x_j) + \epsilon \end{equation}\] pode ser muito flexível, pois permite alguns graus de liberdade por cada covariável, mas não cobre o efeito de interações entre as variáveis independentes, explicativas ou covariáveis. A regressão por prospeção de projeção, proposto por Friedman and Stuetzle (1981), aplica um modelo aditivo às variáveis projetadas.
A ideia é aproximar a função de regressão \(r(x_1,x_2,\cdots,x_p)\) com uma função da forma \[\begin{equation} \mu+\sum_{m=1}^M r_m(z_m), \end{equation}\] onde \[\begin{equation} z_m \, = \, \alpha_m^\top X \end{equation}\] e cada \(\alpha_m\) é um vetor unitário, ou seja, de comprimento um, para \(m = 1,\cdots,M\).
Assim, este método usa um modelo aditivo nas variáveis preditoras que são formadas projetando \(X\) em \(M\) direções cuidadosamente escolhidas. Mesmo sendo \(M\) muito grande tais modelos podem aproximar, uniformemente em conjuntos compactos e em muitos outros sentidos, funções contínuas arbitrárias de \(X\). Os termos \(r_m(z_m)\) são chamados de funções de crista, uma vez que são constantes em todas as direções, exceto uma.
Note que cada \(z_m\) é a projeção de \(X\) num subespaço. O vetor de direção \(\alpha\) é escolhido em cada etapa para minimizar a fração de variância inexplicada. Em mais detalhes, denotemos por \(S(\cdot)\) o mapeamento que produz \(n\) valores ajustados de algun método de alisamento, dados os \(Y_i\) e alguns valores das covariáveis unidimensionais \(z_1,\cdots, z_n\).
Seja também \(\widehat{μ} = \overline{Y}\) e substitua \(Y_i\) por \(Y_i - \overline{Y}\). Assim, os \(Y_i\) agora têm média 0. Da mesma forma, modificamos as covariáveis para que cada uma tenham a mesma variância. Então siga os seguintes passos:- Passo 1: Inicialize os resíduos \(\widehat{\epsilon}_i=Y_i\), \(i=1,\cdots,n\) e seja \(m=0\).
- Pssso 2: Encontrar a direção, vetor unitário, \(\alpha\) que maximiza \[\begin{equation} I(\alpha) \, = \, 1-\frac{\displaystyle \sum_{i=1} ^n \big( \widehat{\epsilon}_i-S(\alpha^\top x_i)\big)^2}{\displaystyle\sum_{i=1}^n \widehat{\epsilon}_i^2}, \end{equation}\] e seja \(z_{mi}=\alpha^\top x_i\), \(\widehat{r}_m(z_{mi})=S(z_{mi})\).
- Pssso 3: Defina \(m = m + 1\) e atualize os resíduos: \[\begin{equation} \widehat{\epsilon}_i - \widehat{r}_m(z_{mi}) \, \to \, \widehat{\epsilon}_i\cdot \end{equation}\] Se \(m=M\) paramos, caso contrário, voltamos para o Passo 2.
Exemplo 16.
A função ppr (Projection Pursuit Regression) permite-nos ajustar \[ \mu+\sum_{m=1}^M r_m(\alpha_m^\top X) \] por mínimos quadrados e restringe os vetores \(\alpha_m\) a serem de comprimento unitário.
Primeiramente, ajusta os termos \(M_{max}\) (max.terms) sequencialmente, depois remove de volta a \(M\) (nterms) em cada estágio, eliminando o termo menos eficiente e o reajusta. A função retorna a proporção da variância explicada por todos os ajustes de \(M,\cdots,M_{max}\).
library(np)
data(wage1)
modelo.ppr1 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 2, max.terms = 5)
R2(wage1$lwage, fitted.values(modelo.ppr1))## [1] 0.447875modelo.ppr2 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 2, max.terms = 6)
R2(wage1$lwage, fitted.values(modelo.ppr2))## [1] 0.45218modelo.ppr3 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 2, max.terms = 7)
R2(wage1$lwage, fitted.values(modelo.ppr3))## [1] 0.4691726modelo.ppr4 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 2, max.terms = 8)
R2(wage1$lwage, fitted.values(modelo.ppr4))## [1] 0.4697552modelo.ppr5 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 2, max.terms = 9)
R2(wage1$lwage, fitted.values(modelo.ppr5))## [1] 0.46254O maior valor de \(R^2\) foi encontrado com o modelo no objeto modelo.ppr4 e mostramos o resumo do ajuste.
## Call:
## ppr(formula = lwage ~ female + educ + exper, data = wage1, nterms = 2,
## max.terms = 8)
##
## Goodness of fit:
## 2 terms 3 terms 4 terms 5 terms 6 terms 7 terms 8 terms
## 78.65109 82.16414 71.81014 73.06259 70.76169 70.57399 72.15356
##
## Projection direction vectors ('alpha'):
## term 1 term 2
## femaleFemale -0.96783665 -0.58241848
## femaleMale -0.25074206 -0.80764297
## educ -0.01054856 0.08969389
## exper -0.01758883 0.02136698
##
## Coefficients of ridge terms ('beta'):
## term 1 term 2
## 0.3836857 0.3625912As informações adicionadas são os vetores de direção \(\alpha_m\) e os coeficientes \(\beta_{im}\) em \[\begin{equation} Y_i \, = \, \mu_{i} \, + \, \sum_{m=1}^M \beta_{im}r_m(\alpha_m^\top X) \, + \, \epsilon\cdot \end{equation}\] Note que esta é uma extensão do modelo de regressão por prospeção de projeção para múltiplas respostas e, então, separamos os escalonamentos das funções suaves \(r_m\), que são escalonadas para ter média zero e variância unitária sobre as projeções do conjunto de dados.
O algoritmo primeiro acrescenta os \(M_{max}\) termos (max.terms) um de cada vez; ele usará menos se não conseguir encontrar um termo para adicionar que faça diferença suficiente. Em seguida, ele remove o termo menos importante em cada etapa até que os \(M\) (nterms) termos sejam deixados.
Podemos perceber que o valor de \(M\) é arbitrário, por isso acompanhamos o ajuste de cada modelo com o valor correspondente do valor do \(R^2\). Por isso escolhemos o modelo em modelo.ppr4, mas percebemos que o maior valor de variância explicada acontece quando a quantidade de termos é \(M=3\).
modelo.ppr11 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 3, max.terms = 5)
R2(wage1$lwage, fitted.values(modelo.ppr11))## [1] 0.4752204modelo.ppr21 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 3, max.terms = 6)
R2(wage1$lwage, fitted.values(modelo.ppr21))## [1] 0.4721492modelo.ppr31 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 3, max.terms = 7)
R2(wage1$lwage, fitted.values(modelo.ppr31))## [1] 0.4764152modelo.ppr41 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 3, max.terms = 8)
R2(wage1$lwage, fitted.values(modelo.ppr41))## [1] 0.4460973modelo.ppr51 = ppr(lwage ~ female+educ+exper, data = wage1,
nterms = 3, max.terms = 9)
R2(wage1$lwage, fitted.values(modelo.ppr51))## [1] 0.4861771## Call:
## ppr(formula = lwage ~ female + educ + exper, data = wage1, nterms = 3,
## max.terms = 9)
##
## Goodness of fit:
## 3 terms 4 terms 5 terms 6 terms 7 terms 8 terms 9 terms
## 76.30390 74.10947 73.00246 71.62785 70.10163 70.17658 68.77666
##
## Projection direction vectors ('alpha'):
## term 1 term 2 term 3
## femaleFemale -0.22908816 0.98099440 0.14632600
## femaleMale 0.74008895 0.05253875 0.97618737
## educ 0.63226754 0.08195210 -0.15525979
## exper 0.00497110 0.16784969 -0.03925963
##
## Coefficients of ridge terms ('beta'):
## term 1 term 2 term 3
## 0.3850895 0.3699895 0.2478915Preditos = predict(modelo.ppr51)
par(mfrow=c(2,2), pch = 19, cex = 0.8)
plot(wage1$lwage, Preditos)
plot(modelo.ppr51)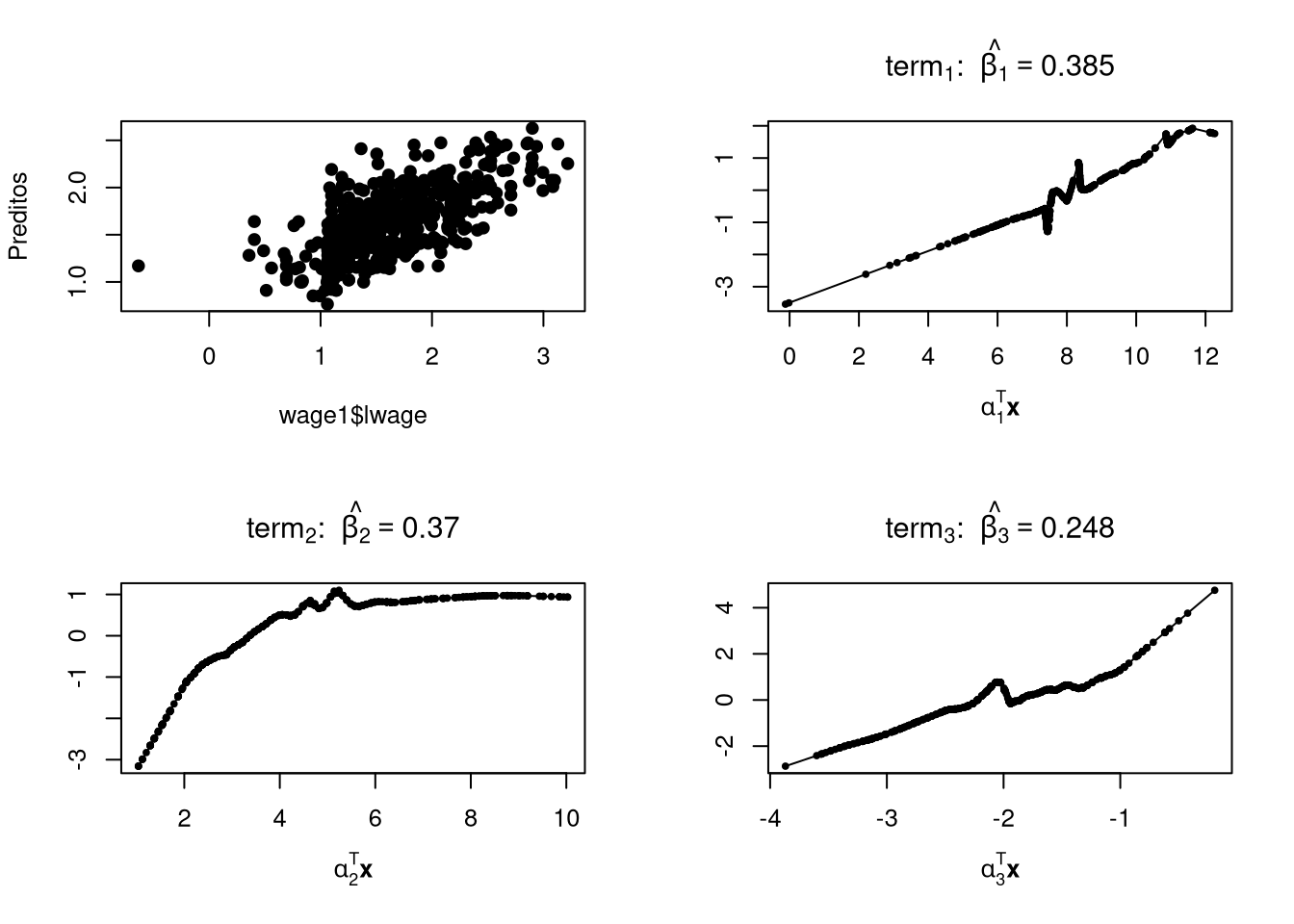
A ideia da prospeção de projeção não é nova. A interpretação de dados de alta dimensão através do uso de projeções de dimensões inferiores bem escolhidas é um procedimento padrão na análise de dados multivariados. A escolha de uma projeção é geralmente guiada por uma figura de mérito apropriada. Se o objetivo é preservar as distncias entre pontos tão bem quanto possível, a figura de mérito apropriada é a variância dos dados projetados, levando à projeção no maior componente principal.
Se o propósito é separar duas amostras gaussianas com matrizes de covariância iguais, a figura de mérito é a taxa de erro de uma regra de classificação unidimensional na projeção, levando à Análise Linear Discriminante. Em ambos os casos, a figura de mérito é especialmente simples e a solução pode ser encontrada pela álgebra linear.
A regressãoo por prospeção de projeção segue uma receita semelhante. Ela constrói um modelo da superfície de regressão com base em projeções dos dados nos planos abrangidos pela resposta \(Y\) e uma combinação linear \(\alpha X\) das preditoras. Aqui, a figura de mérito para uma projeção é a fração de variância explicada por um alisamento de \(Y\) versus \(\alpha X\). A estrutura é removida formando os resíduos do alisamento e substituindo-os pela resposta. O modelo em cada iteração é a soma das suavizações que foram subtraídas anteriormente e, portanto, incorpora a estrutura até agora encontrada.
6.5 Regressão spline adaptativa
Regressão spline adaptativa multivariada (MARS, Friedman, 1991) é uma metodologia para aproximar a função de muitas variáveis de entrada dado o valor da função - talvez contaminada com ruído - em uma coleção de pontos no espaço de entrada. Embora os tempos de treinamento para esse método tendam a ser muito mais rápidos do que as redes neurais, ele ainda pode ser bastante lento para problemas grandes que exigem aproximações complexas (muitas unidades). Surgiu então uma modificação, no algoritmo MARS original, que podem aumentar drasticamente sua velocidade nessas situações.
O problema da supervisão de aprendizagem é facilmente formulado. Um deles tem um conjunto de variáveis \(x=(x_1,\cdots,x_p)\) que são consideradas entradas simultâneas de um sistema e outro conjunto \(y = (y_1,\cdots,y_q)\) que são consideradas saídas. Uma relação da forma \[\begin{equation} \tag{1} y_j = f_j(x_1,\cdots,x_p)+\epsilon_j, \quad j=1,\cdots,q, \end{equation}\] a qual presume-se que exista entre as entradas e saídas, onde \(\{f_j\}_{j=1}^q\) são funções determinísticas (alvo) de valor único de \(p\) variáveis e \(\{e_j\}_{j=1}^q\) são quantidades que variam aleatoriamente, refletindo o fato de que um conjunto simultâneo de valores de entrada pode não ter valores específicos exclusivos para as saídas.
O sistema em estudo pode estar respondendo a entradas adicionais (ocultas) que não são medidas nem controladas. Por convenção, os valores esperados (médios) de cada componente aleatório \(\epsilon_j\) são considerados zero \(\mbox{E}(\epsilon_j)=0\).
O objetivo da aprendizagem supervisionada é obter uma aproximação útil \(\widehat{f}_j\) para cada função (determinística) \(f_j\) em (1), usando um conjunto de exemplos (dados de treinamento) \[\begin{equation} \tag{2} \{y_{i1},\cdots,y_{iq};x_{i1},\cdots,x_{ip}\}_{i=1}^N, \end{equation}\] obtido pela observação do sustem em estudo.
Entre as características distintivas da abordagem MARS estão tempos de treinamento geralmente rápidos, a capacidade de lidar com variáveis de entrada não numéricas (categóricas) de múltiplos valores e valores de entrada ausentes, de maneira natural e de produzir funções de aproximação que tenham valor interpretativo. Ou seja, eles percebem a natureza da dependência (multivariável) dos resultados em relação aos insumos.
6.5.1 Algoritmo MARS
MARS produz aproximações que assumem a forma de uma expansão em um conjunto de funções bases \(\{B_m(x_1,\cdots,x_p)\}_{m=0}^M\) \[\begin{equation} \tag{3} \widehat{f}(x_1,\cdots,x_p) = \sum_{m=0}^M a_m B_m(x_1,\cdots,x_p)\cdot \end{equation}\]
Para um determinado conjunto de funções bases, os coeficientes são determinados por um ajuste de mínimos quadrados de (3) aos dados de treinamento \[\begin{equation} \tag{4} \{a_m\}_{m=0}^M = \operatorname*{arg\,min}_{\{\alpha_m\}_{m=0}^M} \sum_{n=1}^N \left(y_n-\sum_{m=0}^M \alpha_m B_m(x_{n1},\cdots,x_{np})\right)^2\cdot \end{equation}\]
Na abordagem MARS as funções base também são adaptadas aos dados de treinamento. Elas asumem a forma \[\begin{equation} \tag{5} B_m(x_{1},\cdots,x_{p})=\prod_{k=1}^{K_m} b_{km} (x_{\nu(k,m)} \, | \, P_{km}), \end{equation}\] que é um produto de \(K_m\) funções elementares \(b_{km}(\cdot)\), cada uma de uma única variável de entrada \(x_{\nu(k,m)}\) e caracterizada por um conjunto de parâmetros \(P_{km}\). Se a variável de entrada \(x_{\nu(k,m)}\) assumir valores numéricos (reais) ordenáveis, então \[\begin{equation} \tag{6} b_{km} (x \, | \, s,t) =[s(x-t)]_+, \end{equation}\] sendo que o sobescrito indica a parte positiva do argumento \[\begin{equation} [z]_+ = \left\{ \begin{array}{ll} z, & \mbox{ se } z>0 \\ 0, & \mbox{caso contrário} \end{array}\right.\cdot \end{equation}\]
Os parâmetros associados a (6) são a localização dos “nós” \(-\infty \leq t\leq \infty\) e o sentido de truncamento \(s=\pm 1\). Ou seja, \(P_{km}=(s_{km} ,t_{km})\) no fator correspondente (5). Se \(x_{\nu(k,m)}\) assume um conjunto de valores categóricos \[\begin{equation} \tag{7} x_{\nu(k,m)}\in \{c_1,\cdots,c_K\}, \end{equation}\] onde não há relação de ordem ou distância entre os valores, então \[\begin{equation} \tag{8} b_{km}(x \, | \, A) = H(x \in A), \end{equation}\] onde \(A\) é um subconjunto dos valores assumíveis para \(x\), \(A\subset \{c_1,\cdots,c_K\}\) e \(H(\eta)\) é uma função com valor 0/1 indicando a verdade de seu argumento (lógico) \[\begin{equation} \tag{9} H(\eta) = \left\{ \begin{array}{rl} 1, & \mbox{ se } \eta \mbox{ for verdadeiro} \\ 0, & \mbox{ caso contrário} \end{array} \right.\cdot \end{equation}\] Nesta situação \(P_{km}=A_{km}\), um subconjunto enumerável de valores para \(x_{\nu(k,m)}\) (5).
O objetivo do algoritmo MARS é produzir um bom conjunto de funções base \(\{B_m\}_{m=0}^M\) (3), (5), (6), (8) para aproximar cada saída da função alvo \(f_j\) (1), com cálculo viável. Isto é conseguido através de uma abordagem iterativa forward/backward. A parte forward tenta sintetizar um super conjunto dessas funções base. Ou seja, um número maior que o ideal é gerado em uma tentativa deliberada de superajustar a amostra de treinamento (2). O procedimento iterativo backward então exclui seletivamente as funções base com o objetivo de produzir a aproximação mais generalizável, onde todas as funções base são elegíveis para exclusão. A motivação para esta abordagem (forward/backware) é dada em Friedman (1991).
A síntese da função básica na parte forward do algoritmo prossegue da seguinte forma. O conjunto de funções de base inicial, na iteração \(I=0\), consiste na única função constante \[\begin{equation} \tag{10} B_0(x) = 1\cdot \end{equation}\]
Cada iteração sucessiva \(I\geq 1\) sintetiza duas novas funções básicas da forma \[\begin{equation} \tag{11a} B_{2 I-1}(x) = B_\ell(x)b(x_\nu \, | \, P), \end{equation}\] \[\begin{equation} \tag{11b} B_{2 I}(x) = B_\ell(x)b(x_\nu \, | \, \overline{P}), \end{equation}\] onde \(B_\ell(x)\) é uma das funções base produzidas em uma iteração anterior \((0\leq \ell\leq 2I-2)\), \(x_\nu\) é uma das variáveis de entrada \((1\leq \nu\leq p)\) e \(b(\cdot \, | \, P )\) assume a forma de (6) ou (8), dependendo da natureza de \(x_\nu\), real ou categórico.
Os parâmetros \(\overline{P}\) associados a (11b) estão relacionados a \(P\), aqueles associados a (11a). Se \(x_\nu\) tem valor real (6), então \[\begin{equation} \tag{12a} P=(s,t) \qquad \mbox{e} \qquad \overline{P} = (-s,t) \end{equation}\] enquanto que, se \(x_\nu\) for categórico (8) \[\begin{equation} \tag{12b} P=A \qquad \mbox{e} \qquad \overline{P}=\overline{A}, \end{equation}\] onde \(\overline{A}\) é o subconjunto complementar de valores de \(x_\nu\) aos de \(A\); significa que \[\begin{equation} \tag{13} H(x_\nu \in \overline{A}) = 1-H(x_\nu\in A)\cdot \end{equation}\]
As duas funções base (11) produzidas em cada iteração são caracterizadas pelos parâmetros \[\begin{equation} \tag{14} \{\ell,\nu,P\}, \end{equation}\] essa é a função base anterior específica \(B_\ell(x)\), a variável de entrada específica \(x_\nu\), que serve como argumento para a função de variável única \(b(\cdot \, | \, P)\) e os parâmetros correspondentes \(P\) da função.
Os valores para esses parâmetros são ajustados para fornecer o melhor ajuste de mínimos quadrados da aproximação resultante aos dados de treinamento, \[\begin{equation} \tag{15} \begin{array}{rcl} (\ell^*,\nu^*,P^*) & = & \displaystyle \operatorname*{arg\,min}_{\substack{ \{\alpha_i\}_{i=0}^{2I} \\ (\ell,\nu,P)}} \sum_{n=1}^N \Big( y_i -\sum_{i=1}^{2I-2} a_i B_i(x_n) \\ & & \displaystyle \qquad \qquad -a_{2I-1}B_\ell(x_n)b(x_{\nu_n}\, | \, P) -a_{2I}B_\ell (x_n) b(x_{\nu_n}\, | \, \overline{P})\Big)^2\cdot \end{array} \end{equation}\]
Esses valores de parâmetros ideais \((\ell^*,\nu^*,P^*)\) são então usados com (11) para criar as duas novas funções básicas na \(I\)-ésima iteração. Estas são então incluídas no conjunto de funções base previamente sintetizadas para servir como potenciais “pais” em iterações futuras.
Essas iterações continuam até que um número máximo especificado de funções básicas usadas, \(M_\mbox{max}\) seja sintetizado. Regras heurísticas para escolher \(M_\mbox{max}\) são fornecidas em Friedman (1991).
O procedimento stepwise de backware é então aplicado a este conjunto de funções de base final para excluir seletivamente funções de base individuais cuja inclusão é julgada, por um critério de seleção de modelo, para reduzir a generalização.
O algoritmo forward desenvolve funções básicas na forma dada por (5) de maneira hierárquica. Novas funções básicas são criadas multiplicando uma das funções básicas “pai” existentes por uma função simples de uma única variável. As funções de base finais são, portanto, cada produto tensorial de fatores, onde cada fator é função de uma única variável. A aproximação resultante é obtida por um ajuste de mínimos quadrados da saída nas funções básicas que sobrevivem à eliminação retroativa.
Para ilustrar vários conceitos de modelagem MARS, usaremos os dados da Boston Housing.
library(earth) # fit MARS models
Boston.dataset = read.table("http://leg.ufpr.br/~lucambio/MSM/Bostondataset.txt",
header = TRUE, sep = "")
print(Boston.dataset[1:6,], digits = 3)## CRIM ZN INDUS CHAS NOX RM AGE DIS RAD TAX PTRATIO B LSTAT MEDV
## 1 0.00632 18 2.31 0 0.538 6.58 65.2 4.09 1 296 15.3 397 4.98 24.0
## 2 0.02731 0 7.07 0 0.469 6.42 78.9 4.97 2 242 17.8 397 9.14 21.6
## 3 0.02729 0 7.07 0 0.469 7.18 61.1 4.97 2 242 17.8 393 4.03 34.7
## 4 0.03237 0 2.18 0 0.458 7.00 45.8 6.06 3 222 18.7 395 2.94 33.4
## 5 0.06905 0 2.18 0 0.458 7.15 54.2 6.06 3 222 18.7 397 5.33 36.2
## 6 0.02985 0 2.18 0 0.458 6.43 58.7 6.06 3 222 18.7 394 5.21 28.7O conjunto de dados da Boston Housing foi analisado por Harrison and Rubinfeld (1978), eles queriam descobrir se o ar limpo tinha influência nos preços das casas. Compreende 506 observações para cada distrito censitário da área metropolitana de Boston. O conjunto de dados foi analisado em Belsley, Kuh and Welsch (1980).
Para esse estudo foram coletadas as seguintes informações:
\(X_1\): CRIM Taxa de criminalidade per capita,
\(X_2\): ZN Proporção de terrenos residenciais zoneados para grandes lotes,
\(X_3\): INDUS Proporção de acres não negociáveis,
\(X_4\): CHAS Rio Charles (1 se o trato limita o rio, 0 caso contrário),
\(X_5\): NOX Concentração de óxidos nítricos,
\(X_6\): RM Número médio de cômodos por domicílio,
\(X_7\): AGE Proporção de unidades ocupadas pelos proprietários construídas antes de 1940,
\(X_8\): DIS Distâncias ponderadas para cinco centros de emprego de Boston,
\(X_9\): RAD Índice de acessibilidade às rodovias radiais,
\(X_{10}\): TAX Taxa de imposto predial de valor total por US$ 10.000,
\(X_{11}\): PTRATIO Relação aluno/professor,
\(X_{12}\): B \(1000 (B-0.63)^2\mbox{I}\big(B<0.63 \big)\) onde \(B\) é a proporção de afro-americanos,
\(X_{13}\): LSTAT % estrato inferior da população,
\(X_{14}\): MEDV Valor médio das casas ocupadas pelos proprietários em US$ 1.000.
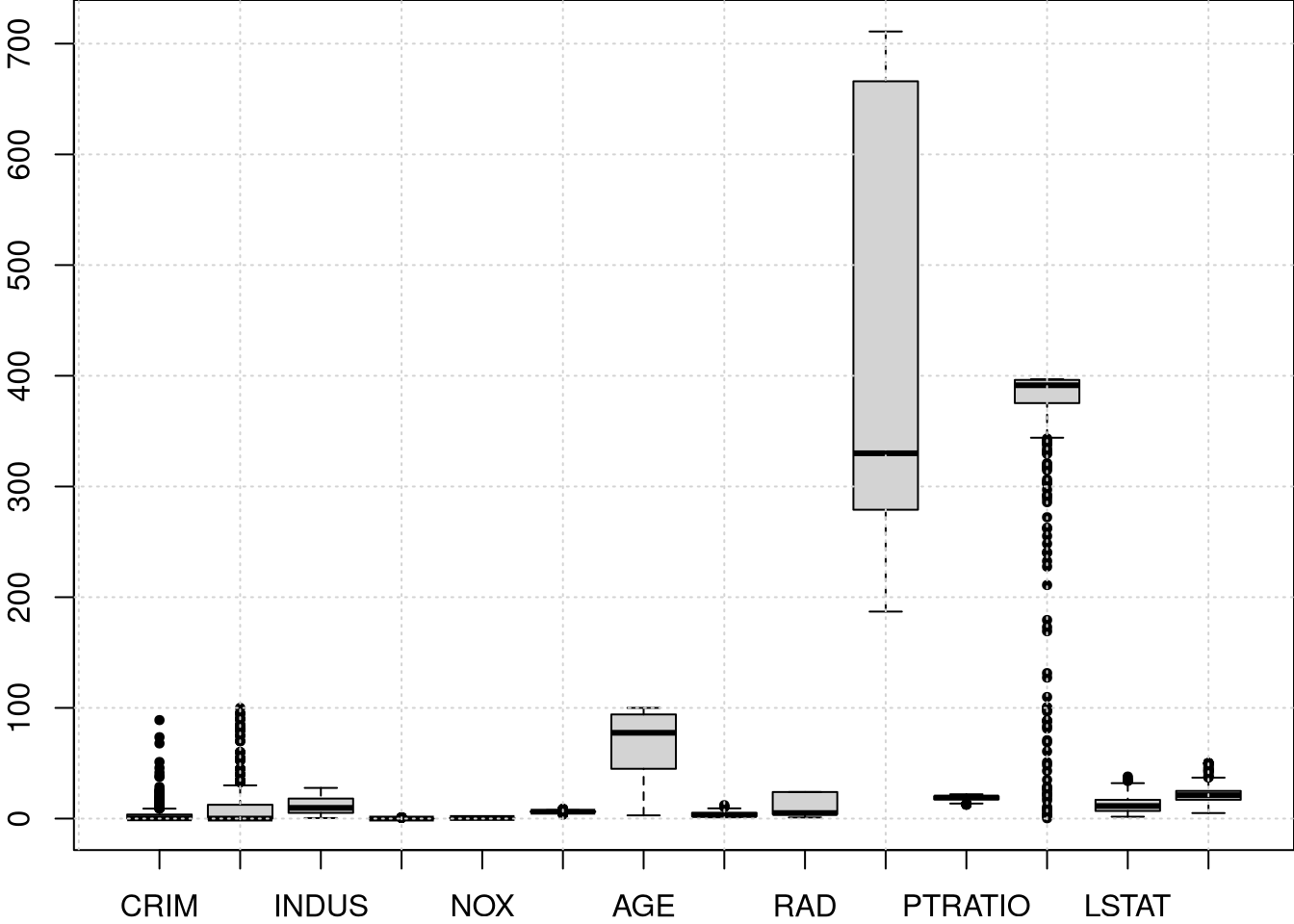
Tomar o logaritmo ou elevar as variáveis à potência de algo menor que um ajuda a reduzir a assimetria. Isso se deve ao fato de que os valores mais baixos se afastam mais um do outro, enquanto a distância entre valores maiores é reduzida por essas transformações. A figura acima exibe os boxplots para as variáveis na escala original e abaixo as transformadas.
Como a maioria das variáveis exibe uma assimetria com maior densidade no lado esquerdo, as seguintes transformações são propostas: \(\widetilde{X}_1 = \log(X_1)\), \(\widetilde{X}_2=X_2/10\), \(\widetilde{X}_3=\log(X_3)\), \(\widetilde{X}_4\) nenhuma transformação, dado que \(X_4\) é binária, \(\widetilde{X}_5=\log(X_5)\), \(\widetilde{X}_6=\log(X_6)\), \(\widetilde{X}_7=X_7^{2.5}/10000\), \(\widetilde{X}_8=\log(X_8)\), \(\widetilde{X}_9=\log(X_9)\), \(\widetilde{X}_{10}=\log(X_{10})\), \(\widetilde{X}_{11}=\exp(0.4\times X_{11})/1000\), \(\widetilde{X}_{12}=X_{12}/100\), \(\widetilde{X}_{13}=\sqrt{X_{13}}\) e \(\widetilde{X}_{14}=\log(X_{14})\).
Boston.trans = Boston.dataset
Boston.trans[,1] = log(Boston.dataset[,1])
Boston.trans[,2] = Boston.dataset[,2]/10
Boston.trans[,3] = log(Boston.dataset[,3])
Boston.trans[,4] = Boston.dataset[,4]
Boston.trans[,5] = log(Boston.dataset[,5])
Boston.trans[,6] = log(Boston.dataset[,6])
Boston.trans[,7] = Boston.dataset[,7]^2.5/10000
Boston.trans[,8] = log(Boston.dataset[,8])
Boston.trans[,9] = log(Boston.dataset[,9])
Boston.trans[,10] = log(Boston.dataset[,10])
Boston.trans[,11] = exp(0.4*Boston.dataset[,11])/1000
Boston.trans[,12] = Boston.dataset[,12]/100
Boston.trans[,13] = sqrt(Boston.dataset[,13])
Boston.trans[,14] = log(Boston.dataset[,14])
par(mar=c(2,2,0,0))
boxplot(Boston.trans[,1:14], cex = 0.6, pch = 19)
grid()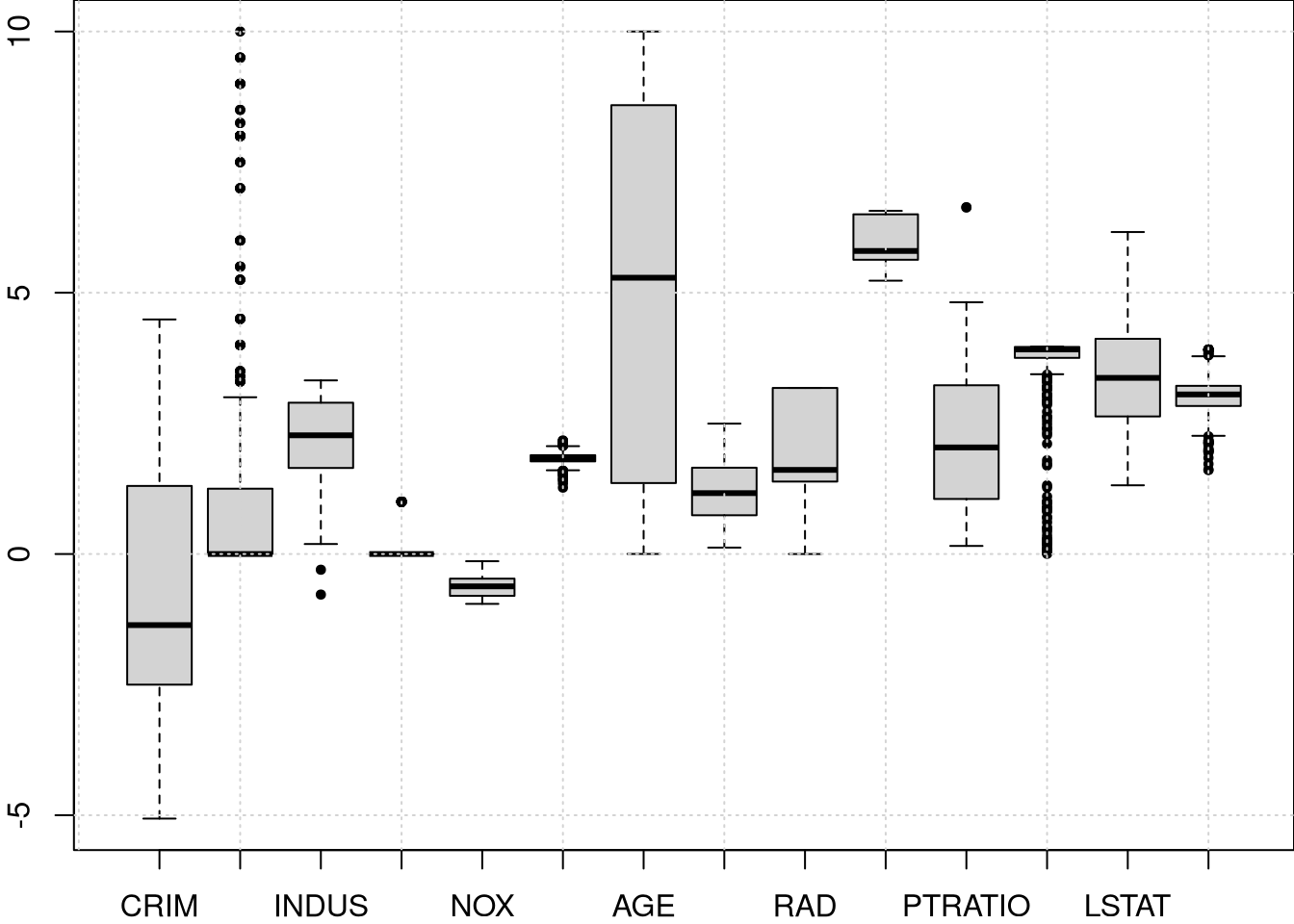
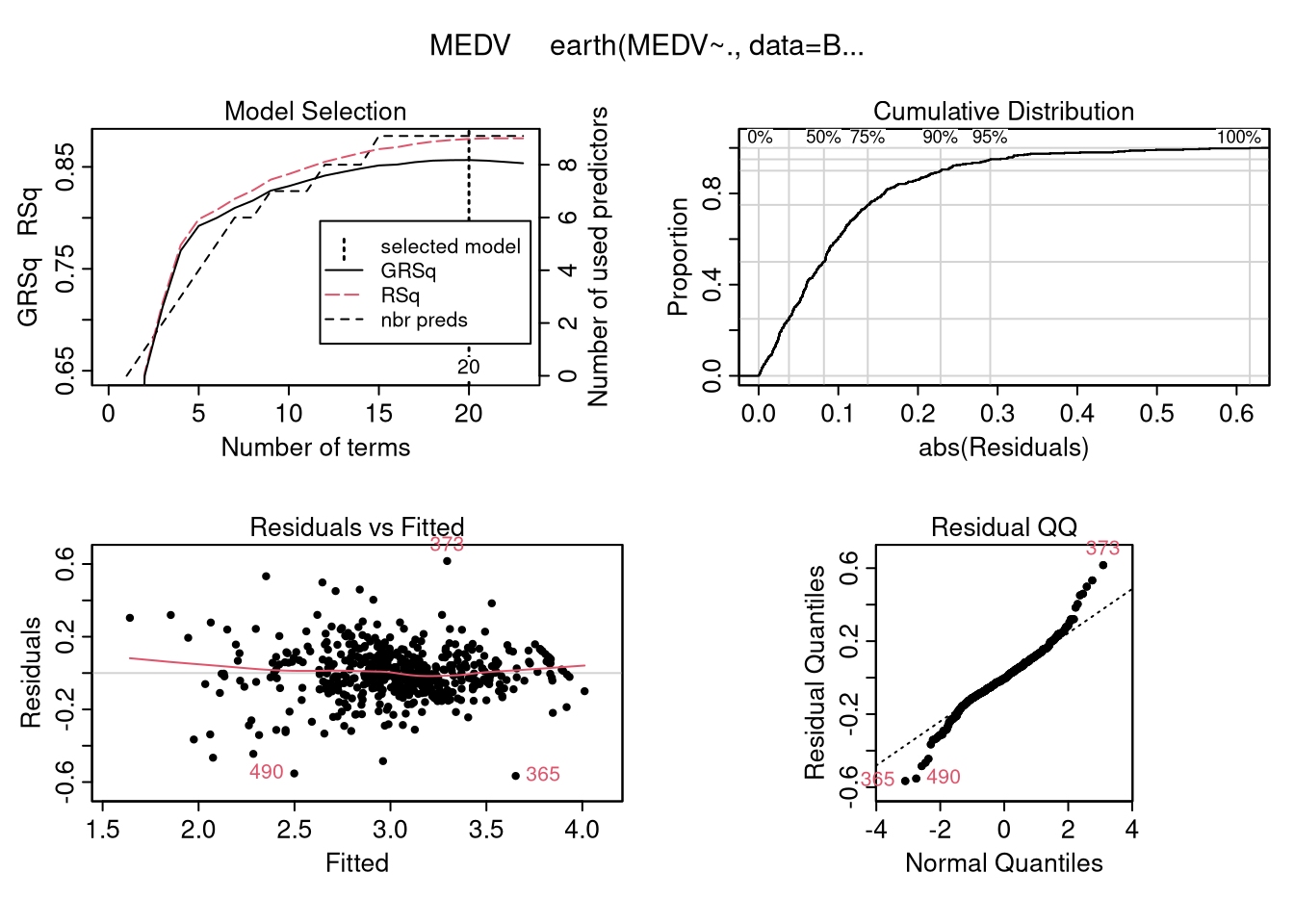
## Call: earth(formula=MEDV~., data=Boston.trans)
##
## coefficients
## (Intercept) 3.07
## h(CRIM-1.48666) -0.21
## h(NOX- -0.537854) 3.25
## h(-0.471605-NOX) 0.27
## h(NOX- -0.471605) -8.63
## h(NOX- -0.366725) 12.07
## h(NOX- -0.331286) -6.58
## h(RM-1.84609) 1.53
## h(0.563722-DIS) 1.10
## h(DIS-0.563722) 0.51
## h(DIS-0.787275) -0.65
## h(2.07944-RAD) -0.06
## h(RAD-2.07944) 0.30
## h(5.71703-TAX) 0.26
## h(TAX-5.71703) -0.30
## h(4.44707-PTRATIO) 0.05
## h(3.9472-B) -0.04
## h(B-3.9472) -1.91
## h(2.34521-LSTAT) 0.22
## h(LSTAT-2.34521) -0.18
##
## Selected 20 of 23 terms, and 9 of 13 predictors
## Termination condition: Reached nk 27
## Importance: LSTAT, RM, CRIM, DIS, NOX, PTRATIO, RAD, TAX, B, ZN-unused, ...
## Number of terms at each degree of interaction: 1 19 (additive model)
## GCV 0.024 RSS 10 GRSq 0.86 RSq 0.88## nsubsets gcv rss
## LSTAT 19 100.0 100.0
## RM 18 49.7 51.2
## CRIM 17 41.0 42.7
## DIS 16 32.2 34.5
## NOX 15 27.5 30.0
## PTRATIO 14 25.7 28.2
## RAD 12 21.6 24.0
## TAX 9 15.4 18.0
## B 6 9.9 12.6Esta última função retorna uma matriz que mostra as importâncias relativas das variáveis no modelo. Existe uma linha para cada variável. O nome da linha é o nome da variável, mas com -unused anexado se a variável não aparecer no modelo final.
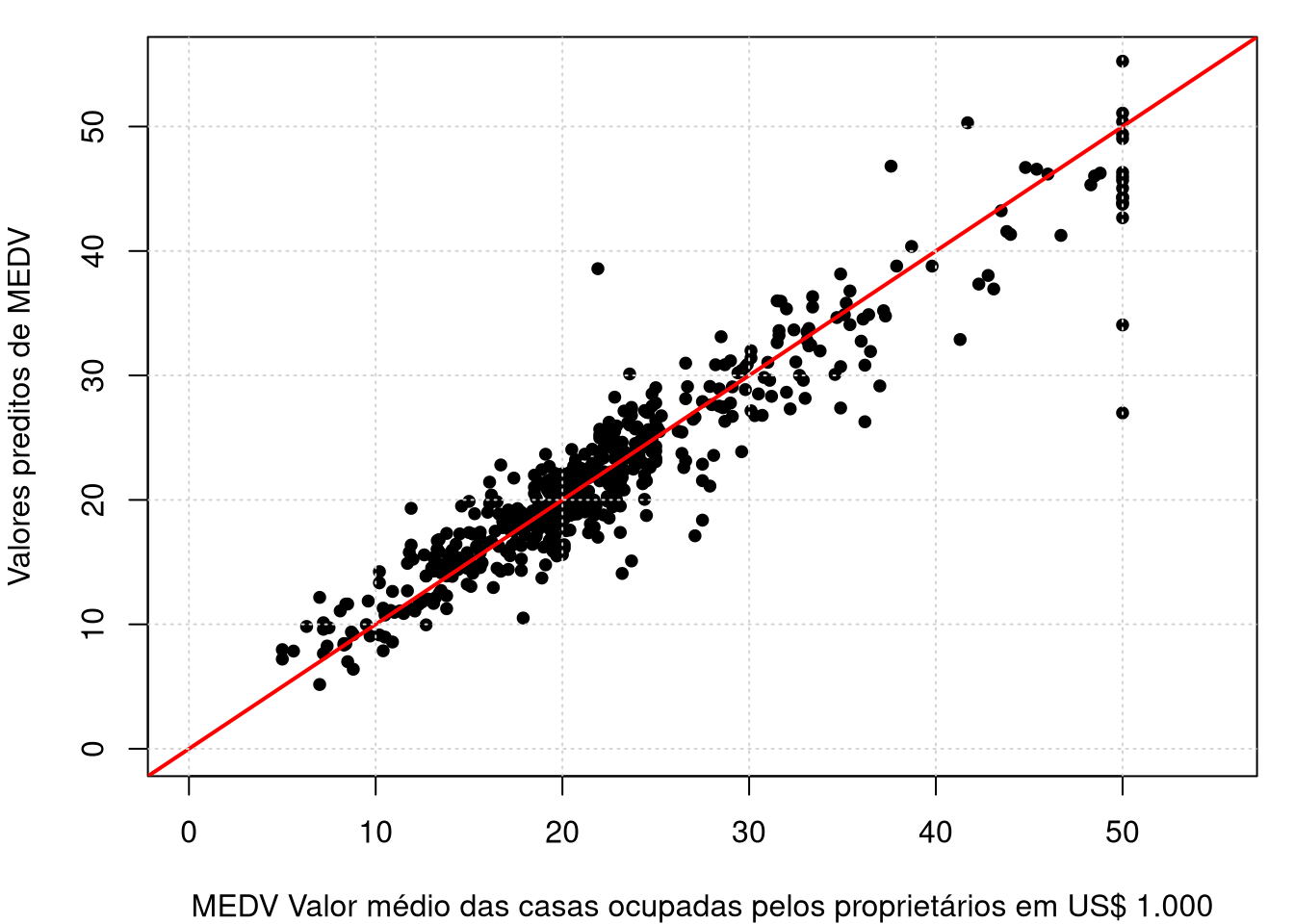
par(mar=c(4,4,1,1))
plot(Boston.dataset[,14],exp(ajuste.mars$fitted.values), cex = 0.8, pch =19,
xlim = c(0,55), ylim = c(0,55),
xlab = "MEDV Valor médio das casas ocupadas pelos proprietários em US$ 1.000",
ylab = "Valores preditos de MEDV")
abline(a=0,b=1,lwd=2,col="red");grid()