Capítulo 7 Árvores de regressão
Uma árvore de regressão é um modelo da forma \[\begin{equation} r(x) \, = \, \sum_{m=1}^M c_m I(x\in R_m), \end{equation}\] onde \(c_1,\cdots,c_M\) são constantes e \(R_1,\cdots,R_M\) são retângulos disjuntos que dividem o espaço das covariáveis. Os modelos de árvores de regressão foram introduzidos por Morgan and Sonquist (1963) e Breiman et al. (1984). O modelo é ajustado de uma maneira recursiva que pode ser representada como uma árvore; daí o nome. Nossa descrição segue Hastie et al. (2001).
Denote um valor genérico das covariáveis por \(x = (x_1,\cdots, x_j, \cdots , x_d)\). A covariável para a \(i\)-ésima observação é \(x_i = (x_{i1}, \cdots, x_{ij},\cdots ,x_{id})\). Dada a covariável \(j\) e um ponto de divisão \(s\) definimos os retângulos \[\begin{equation} R_1 = R_1(j,s) = \{x: x_j\leq s\} \qquad e \qquad R_2 = R_2 (j, s) = \{x: x_j> s\} \end{equation}\] onde, nesta expressão, \(x_j\) se refere à \(j\)-ésima covariável, não a observação \(j\).
Escolhemos então \(c_1\) como a média de todos o \(Y_i\) tais que \(x_i\in R_1\) e \(c_2\) como a média de todos os \(Y_i\) de tal forma que \(x_i\in R_2\). Observe que \(c_1\) e \(c_2\) minimizam as somas de quadrados \(\sum_{x_i\in R_1}(Y_i-c_1)^2\) e \(\sum_{x_i\in R_2}(Y_i-c_2)^2\). A escolha de qual covariável \(x_j\) dividir e qual o ponto de divisão \(s\) para usar é baseado na minimização das somas de quadrados residuais. O processo de divisão é repetido em cada retângulo \(R_1\) e \(R_2\).
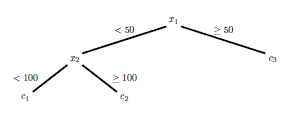
A figura mostra um exemplo simples de uma árvore de regressão; também são mostrados os retângulos correspondentes. A estimativa da função é constante sobre os retângulos.
|

|
|---|
Exemplo de uma árvore de regressão para duas covariáveis \(x_1\) e \(x_2\). A estimativa da função é \[\begin{equation} r(x) = c_1I(x\in R_1) + c_2I(x \in R_2) + c_3I(x \in R_3) \end{equation}\] onde \(R_1\), \(R_2\) e \(R_3\) são os retângulos mostrados ao lado.
Geralmente, uma árvore cresce muito e, em seguida, a árvore é podada para formar uma sub-árvore, colapsando as regiões juntas. O tamanho da árvore é um parámetro de ajuste escolhido da seguinte maneira. Seja \(N_m\) o némero de pontos em um retângulo \(R_m\) de uma sub-árvore \(T\) e defina \[\begin{equation} c_m \, = \, \frac{1}{N_m}\sum_{x_i\in R_m} Y_i \qquad e \qquad Q_m(T) \, = \, \frac{1}{N_m}\sum_{x_i\in R_m} (Y_i-c_m)^2\cdot \end{equation}\]
Definamos a complexidade de \(T\) por \[\begin{equation} C_{\alpha}(T) \, = \, \sum_{m=1}^{|T|} N_m Q_m(T)+\alpha|T|, \end{equation}\] onde \(\alpha> 0\) e \(|T|\) é o número de nós terminais da árvore. Seja \(T_\alpha\) a menor sub-árvore que minimize o \(C_\alpha\). O valor \(\widehat{\alpha}\) de \(\alpha\) pode ser escolhido por validação cruzada. A estimativa final é baseada na árvore \(T_{\widehat{\alpha}}\).
Exemplo 17.
Este exemplo, de Venables and Ripley (2002), envolve três covariáveis e uma variável de resposta. Os dados são 48 amostras de rochas de um reservatório de petróleo.
A resposta é a perm, a permeabilidade da rocha em milli-Darcies. As covariáveis são: area, a área dos poros em pixels de 256 por 256, peri, o perímetro em pixels e shape, a forma do poro medida em perímetro/\(\sqrt{\mbox{área}}\).
O objetivo é prever a permeabilidade utilizando as covariáveis. Um modelo não paramétrico é \[\begin{equation} \mbox{permeabilidade} \, = \, r(\mbox{área}, \mbox{perímetro}, \mbox{forma}) \, + \, \epsilon\cdot \end{equation}\]
## area peri shape perm
## 1 4990 2791.90 0.0903296 6.3
## 2 7002 3892.60 0.1486220 6.3
## 3 7558 3930.66 0.1833120 6.3
## 4 7352 3869.32 0.1170630 6.3
## 5 7943 3948.54 0.1224170 17.1
## 6 7979 4010.15 0.1670450 17.1library(tree)
rock.model1 = tree(perm ~ area + peri + shape, data = rock)
# opção xpd = NA, caso contrário, em alguns dispositivos, o texto é recortado
par(mfrow = c(1,1), xpd = NA, mar=c(4,4,1,1))
plot(rock.model1); grid()
text(rock.model1, cex=.75)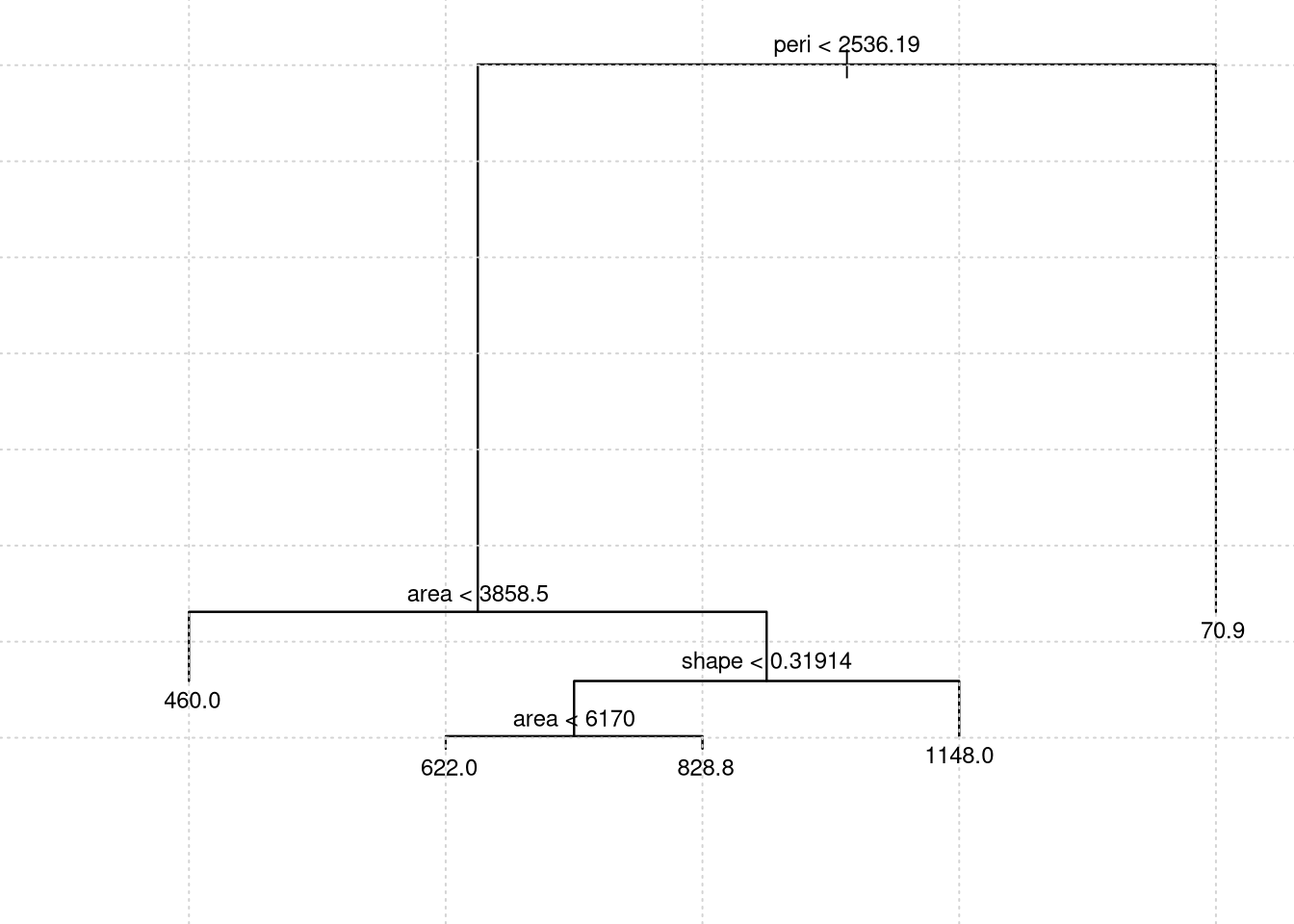
Podemos comparar as previsões com o conjunto de dados.
## [1] 0.790755## [1] 860## [1] 460## [1] 70.9O qual indica um bom ajuste aos dados. Podemos comparar as previsões com o conjunto de dados sendo que, quanto mais escuro maior permeabilidade. Na figura abaixo observe os valores médios de permeabilidade segundo as diferentes classificações.
## node), split, n, deviance, yval
## * denotes terminal node
##
## 1) root 48 9009000 415.4
## 2) peri < 2536.19 24 3252000 760.0
## 4) area < 3858.5 6 1123000 460.0 *
## 5) area > 3858.5 18 1409000 860.0
## 10) shape < 0.31914 13 659500 749.2
## 20) area < 6170 5 409700 622.0 *
## 21) area > 6170 8 118300 828.8 *
## 11) shape > 0.31914 5 175100 1148.0 *
## 3) peri > 2536.19 24 58880 70.9 *rock.model11 = snip.tree(rock.model1, nodes = 5)
perm.deciles = quantile(rock$perm, 0:10/10)
cut.perm = cut(rock$perm, perm.deciles, include.lowest=TRUE)
par(mar=c(4,4,1,1))
plot(rock$peri, rock$area, col=grey(10:2/11)[cut.perm], pch=20,
xlab="Perímetro",ylab="Área"); grid()
partition.tree(rock.model11, ordvars = c("peri", "area"), add = TRUE)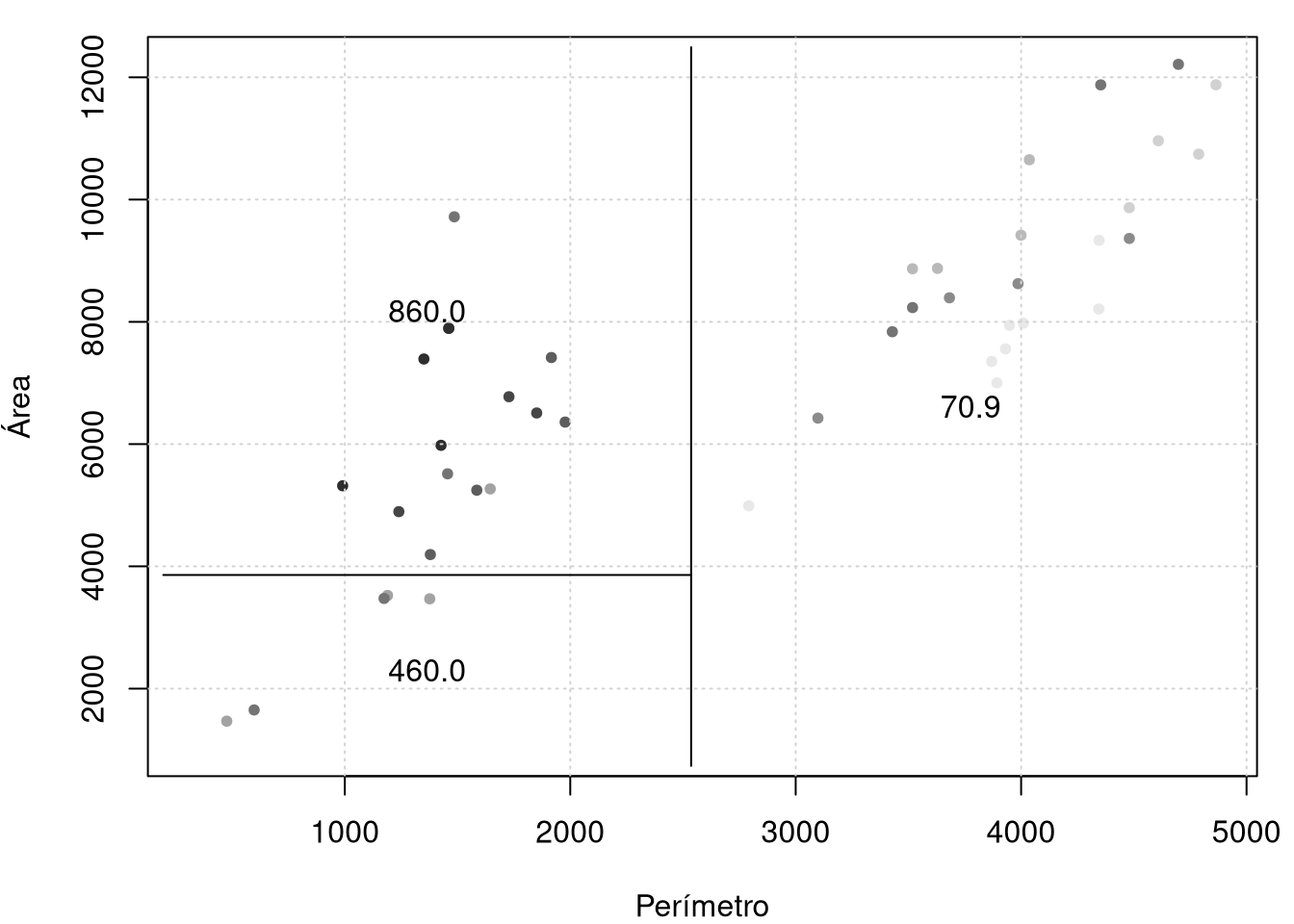
Os gráficos são em duas dimensões, por isso, devemos escolher uma sub-árvore em snip.tree que contenha somente duas variáveis e com esse objetivo eliminamos, por exemplo, o nodo 5 obténdo-se o gráfico a direita acima.
##
## Regression tree:
## tree(formula = perm ~ area + peri + shape, data = rock)
## Number of terminal nodes: 5
## Residual mean deviance: 43840 = 1885000 / 43
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -522.0 -64.6 11.5 0.0 71.1 840.0No resumo do modelo Residual mean deviance significa o erro quadrático médio residual. A flexibilidade da árvore de regressão é basicamente controlada por quantas folhas às árvores têm, isto devido a que são quantas células elas particionam.
A função de ajuste da árvore tem um número de configurações de controles que limitam o quanto crescerá, cada nó deve conter um certo número de pontos e adicionar um nó deve reduzir o erro em pelo menos uma certa quantidade. O padrão para min.dev, é 0.01; vamos desligá-lo e ver o que acontece:
rock.model2 = tree(perm ~ area + peri + shape, data = rock, mindev = 0.001)
par(mar=c(4,4,1,1))
plot(rock.model2); grid()
text(rock.model2, cex=.75)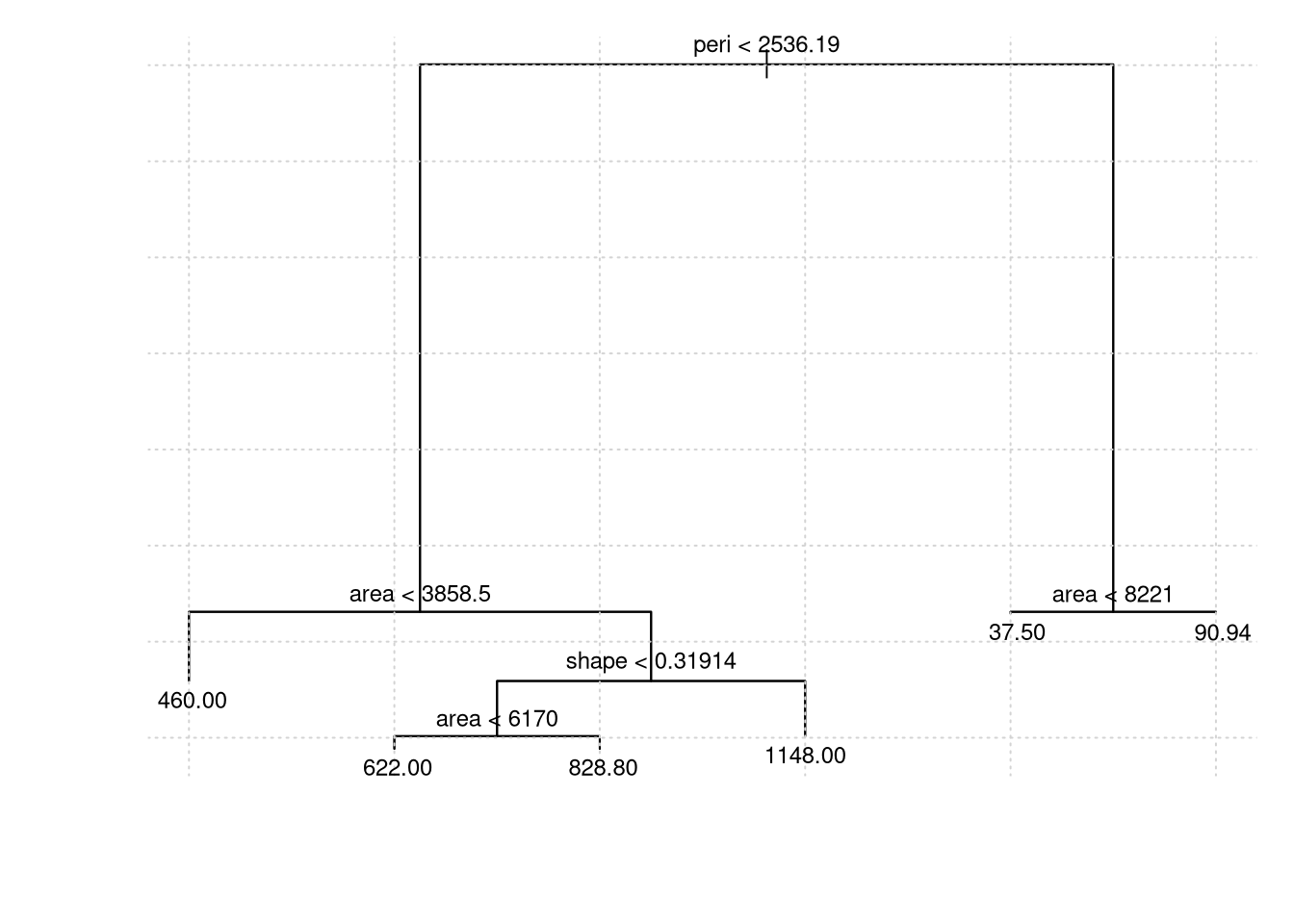
##
## Regression tree:
## tree(formula = perm ~ area + peri + shape, data = rock, mindev = 0.001)
## Number of terminal nodes: 6
## Residual mean deviance: 44500 = 1869000 / 42
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -522.00 -32.34 -8.54 0.00 87.25 840.00As árvores de classificação geram a classe prevista para uma determinada amostra. Vamos usar aqui o conjunto de dados rock.dat dividido em dois: de treinamento e teste. O conjunto de treinamento será de aproximadamente 70% do conjunto original.
set.seed(101)
alpha = 0.7 # percentagem do conjunto de treino
inTrain = sample(1:nrow(rock), alpha * nrow(rock))
train.set = rock[inTrain,]
test.set = rock[-inTrain,]Existem duas opções para a saída: a previsão pontual, na qual simplesmente se fornece a predição da classe e a previsão da distribuição, neste fornece-se uma probabilidade para cada classe. Nosso caso, a resposta é contínua, então temos somente a previsão pontual.
# Ajuste do modelo para a base de treinamento
rock.model3 = tree(perm ~ area + peri + shape, data = train.set)
rock.model3## node), split, n, deviance, yval
## * denotes terminal node
##
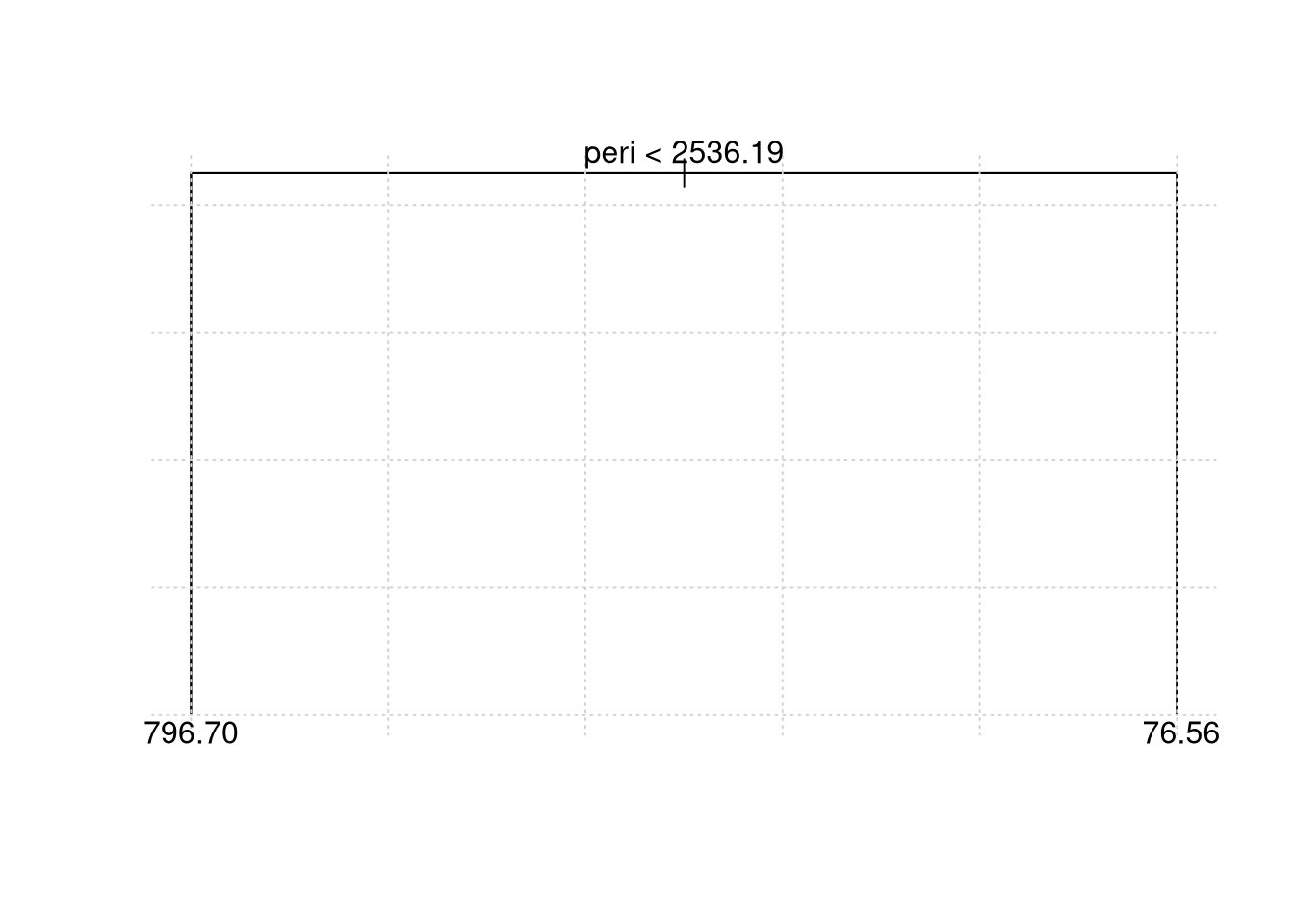
## 1) root 33 6252000 403.90
## 2) peri < 2536.19 15 1966000 796.70
## 4) area < 5183 5 1004000 564.00 *
## 5) area > 5183 10 556400 913.00
## 10) shape < 0.278451 5 149400 790.00 *
## 11) shape > 0.278451 5 255700 1036.00 *
## 3) peri > 2536.19 18 43000 76.56 *##
## Regression tree:
## tree(formula = perm ~ area + peri + shape, data = train.set)
## Number of terminal nodes: 4
## Residual mean deviance: 50060 = 1452000 / 29
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -464.000 -70.260 5.839 0.000 65.440 736.000# Previsão pontual
my.prediction = predict(rock.model3, test.set) # gives the probability for each class
my.prediction## 2 5 7 15 19 23 27 28
## 76.56111 76.56111 76.56111 76.56111 76.56111 76.56111 790.00000 790.00000
## 29 30 36 39 40 42 45
## 790.00000 564.00000 790.00000 564.00000 790.00000 564.00000 564.00000Observe que a variável area não aparece na árvore. Isso significa que essa variável nunca foi a covariável ideal para dividir o algoritmo nessa base de dados de treinamento. O resultado é que a árvore depende apenas da area de poros e do perímetro.
Isso ilustra uma característica importante da árvore de regressão: ela executa automaticamente a seleção de variáveis no sentido de que uma covariável \(x_j\) não aparecerá; na árvore se o algoritmo achar que a variável não é importante.
par(mar=c(4,4,1,1))
plot(rock$peri, rock$shape, pch=19, col=as.numeric(rock$perm),
xlab = "Perímetro", ylab = "Área"); grid()
partition.tree(tree(perm ~ peri + shape, data = train.set),
label="Perímetro", add=TRUE)
legend("topright",legend=unique(rock$perm), col = unique(as.numeric(rock$perm)),
pch=19, cex = 0.6, horiz = FALSE, ncol = 3)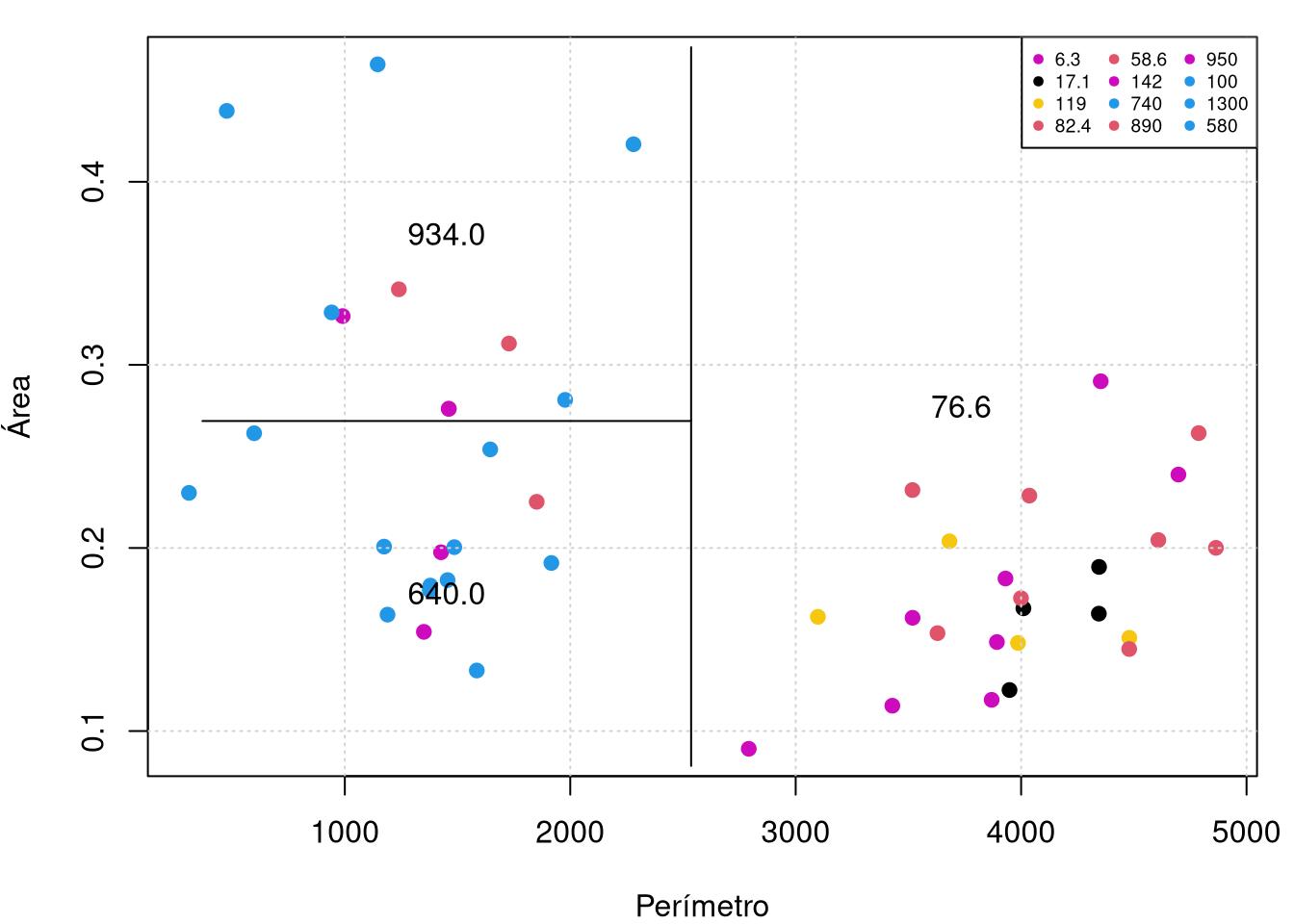
Podemos podar a árvore para evitar overfitting. A próxima função prune.tree() nos permite escolher quantas folhas queremos que a árvore tenha e ela retorna a melhor árvore com esse tamanho. O argumento newdata aceita novas entradas para tomar a decisão de podar. Se novos dados não forem fornecidos, o método usará o conjunto de dados original a partir do qual o modelo de árvore foi criado.
Para árvores de classificação, ou seja, de resposta multinomial também podemos usar o método method = misclass, de modo que a medida de poda seja o número de erros de classificação.
Neste pacote também podemos utilizar validação cruzada para encontrar a melhor árvore, usando cv.tree(). Aqui, vamos usar todas as variáveis e todas as amostras. Na figura acima mostramos dois gráficos, a árvore com somente duas folha (best = 2) e o resultado da árvore obtida por validação cruzada.
##
## Regression tree:
## tree(formula = perm ~ ., data = rock)
## Number of terminal nodes: 5
## Residual mean deviance: 43840 = 1885000 / 43
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -522.0 -64.6 11.5 0.0 71.1 840.0Mostramos o desvio para cada árove segundo o número de folha, quanto menor melhor.
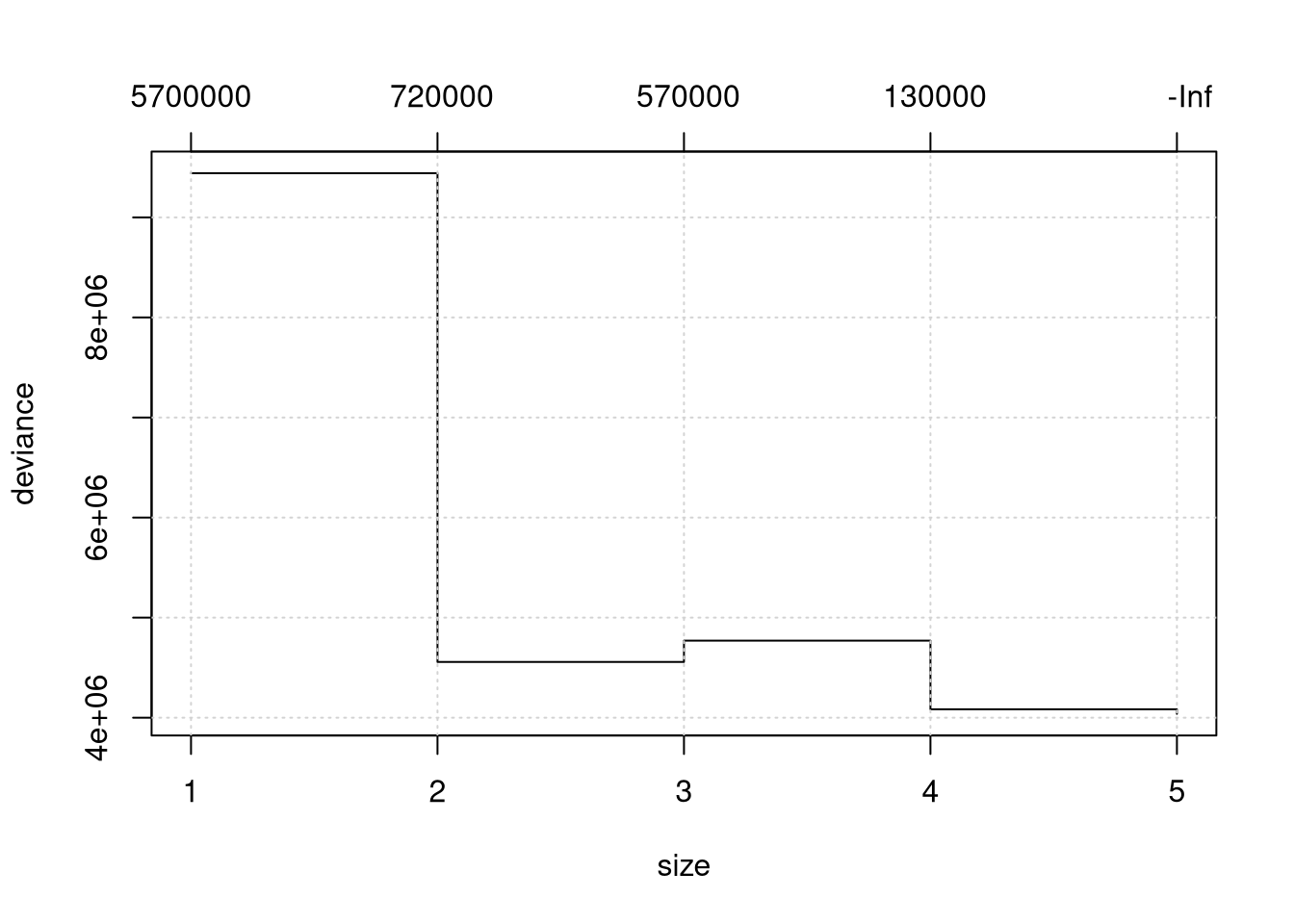
## [1] 4037964 4083612 4769690 4556738 9442144Como é muito difícil decidir assim, perguntamos então qual tamanho é melhor?
## [1] 5e temos por resposta a árvore com 5 folhas. Vamos refazer o modelo da árvore com o número de folhas não sendo maior que o melhor tamanho.
##
## Regression tree:
## tree(formula = perm ~ ., data = rock)
## Number of terminal nodes: 5
## Residual mean deviance: 43840 = 1885000 / 43
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -522.0 -64.6 11.5 0.0 71.1 840.0Podemos fazer mais bonito ainda. O desenvolvimento teórico permanece o mesmo porêm o pacote rpart é mais rápido do que o tree e a qualidade de plotagem e funções de texto são melhores utilizando o pacote partykit.
Exemplo 18. (Continuação do Exemplo 17)
library(rpart)
rock.rpart = rpart(perm ~ ., data = train.set)
plot(rock.rpart, uniform=TRUE, branch=0.6, margin=0.05); grid()
text(rock.rpart, all=TRUE, use.n=TRUE)
title("Árvore no Conjunto de Treinamento")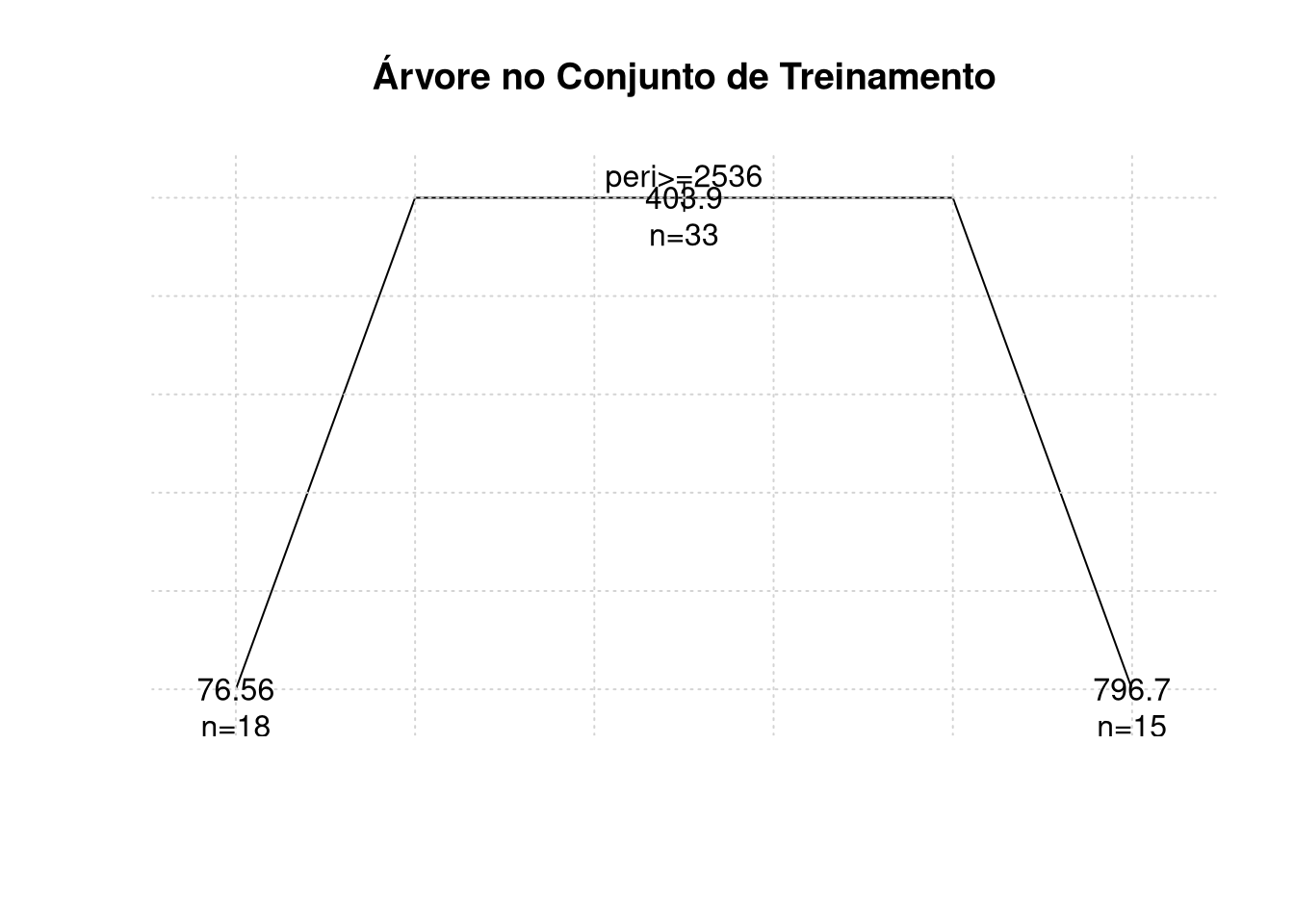
## predictions
## 76.5611111111111 796.666666666667
## 6.3 1 0
## 17.1 2 0
## 58.6 1 0
## 82.4 1 0
## 100 0 2
## 142 1 0
## 580 0 1
## 740 0 2
## 890 0 2
## 950 0 1
## 1300 0 1prune.rpart = prune(rock.rpart, cp=0.02) # podando a árvore
plot(prune.rpart, uniform=TRUE, branch=0.6); grid()
text(prune.rpart, all=TRUE, use.n=TRUE)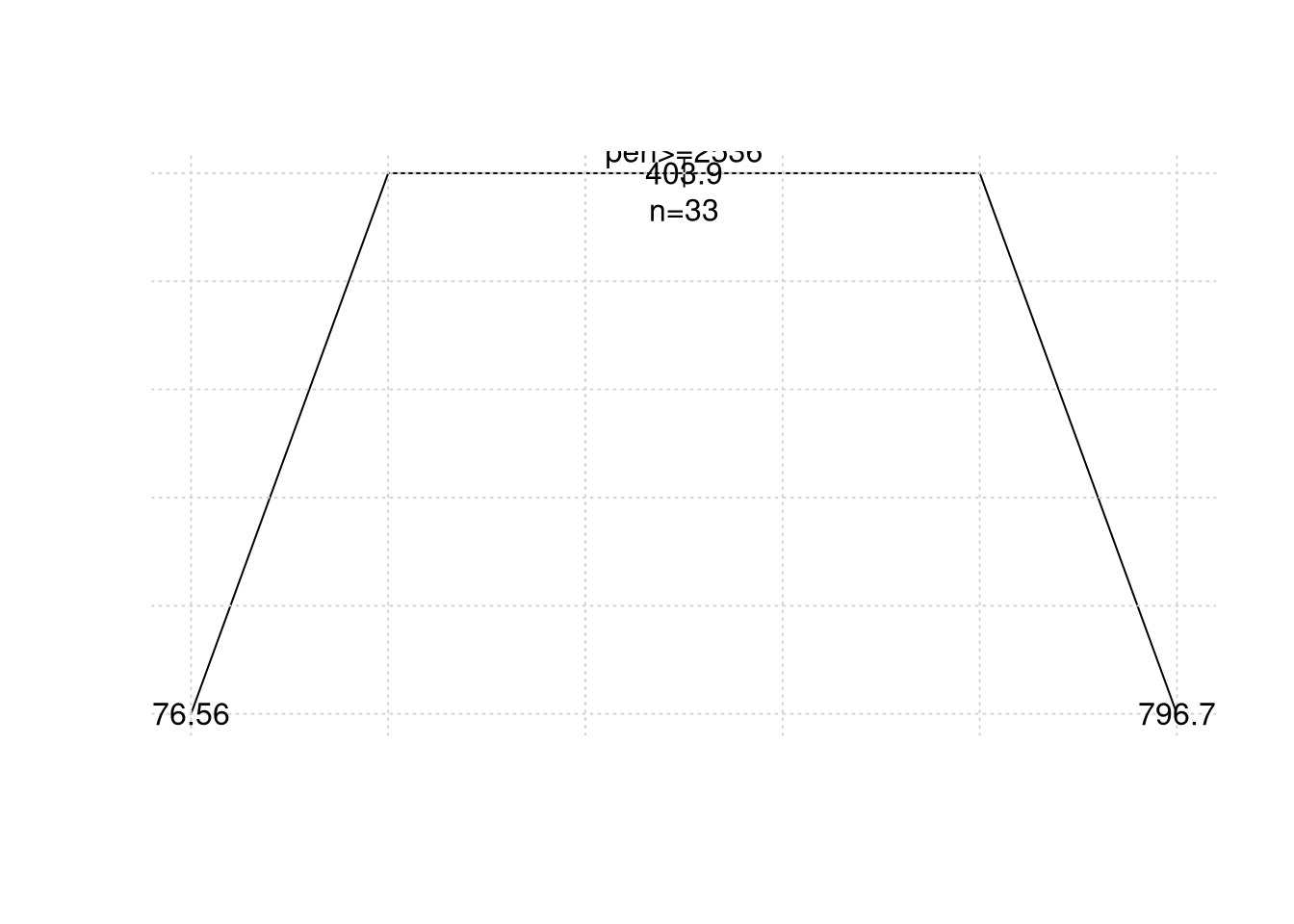
## Call:
## rpart(formula = perm ~ ., data = train.set)
## n= 33
##
## CP nsplit rel error xerror xstd
## 1 0.6786543 0 1.0000000 1.0582701 0.2013903
## 2 0.0100000 1 0.3213457 0.6896235 0.2824689
##
## Variable importance
## peri area shape
## 48 29 23
##
## Node number 1: 33 observations, complexity param=0.6786543
## mean=403.8818, MSE=189443.4
## left son=2 (18 obs) right son=3 (15 obs)
## Primary splits:
## peri < 2536.195 to the right, improve=0.6786543, (0 missing)
## shape < 0.2693715 to the left, improve=0.3865376, (0 missing)
## area < 7475 to the right, improve=0.2069304, (0 missing)
## Surrogate splits:
## area < 6392.5 to the right, agree=0.818, adj=0.600, (0 split)
## shape < 0.172007 to the left, agree=0.758, adj=0.467, (0 split)
##
## Node number 2: 18 observations
## mean=76.56111, MSE=2389.01
##
## Node number 3: 15 observations
## mean=796.6667, MSE=131062.2Apresentamos o resultado do modelo quando podamos a ávore mas não o gráfico porque é o mesmo modelo.
# criando gráficos adicionais
par(mfrow = c(1,2))
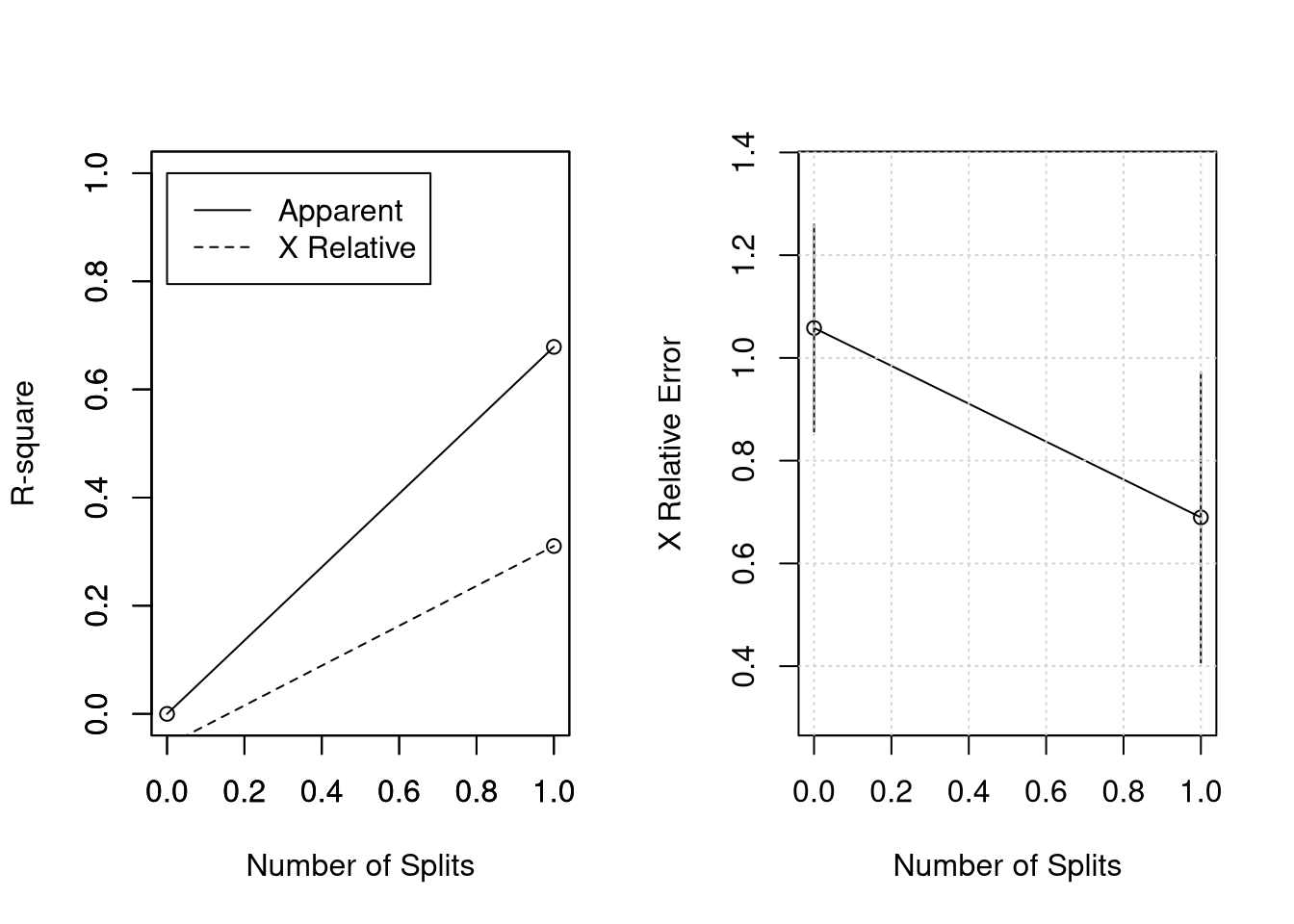
rsq.rpart(rock.rpart);grid() # visualiza resultados de validação cruzada##
## Regression tree:
## rpart(formula = perm ~ ., data = train.set)
##
## Variables actually used in tree construction:
## [1] peri
##
## Root node error: 6251634/33 = 189443
##
## n= 33
##
## CP nsplit rel error xerror xstd
## 1 0.67865 0 1.00000 1.05827 0.20139
## 2 0.01000 1 0.32135 0.68962 0.28247