Experimento fatorial duplo com um controle negativo
Fábio B. Ono & Walmes M. Zeviani
11 de julho de 2019
Source:vignettes/fat2_adic.Rmd
fat2_adic.RmdDescrição e análise exploratória
#-----------------------------------------------------------------------
# Carrega os pacotes necessários.
rm(list = ls())
library(lattice)
library(latticeExtra)
library(gridExtra)
library(doBy)
library(multcomp)
library(EACS)Os dados que serão analisados são de um experimento fatorial duplo 4 \(\times\) 2 com um controle negativo, comumente representado por 4 \(\times\) 2 \(+\) 1. Os dados estão no objeto fat2_adic.
A primeira dica importante para análise de experimentos fatoriais com tratamentos adicionais é: deixe os tratamentos adicionais serem os últimos níveis em todos os fatores. Nessa tabela foi usado controle como rótulo do controle negativo (sem adubação) tanto na coluna do fator fonte quanto na coluna do fator modo. Não é obrigatório que os nomes sejam os mesmo nas colunas. No entanto, não se deve colocar NA porque isso leva a remoção das linhas pelas funções de ajuste que precisam de observações completas.
# Estrutura dos dados.
str(fat2_adic)## 'data.frame': 36 obs. of 4 variables:
## $ fonte: Factor w/ 5 levels "A","B","C","D",..: 5 5 5 5 1 1 1 1 2 2 ...
## $ modo : Factor w/ 3 levels "lanco","sulco",..: 3 3 3 3 1 1 1 1 1 1 ...
## $ bloc : Factor w/ 4 levels "1","2","3","4": 1 2 3 4 1 2 3 4 1 2 ...
## $ prod : num 51.6 48.7 46.1 44.1 61.6 ...fat2_adic## fonte modo bloc prod
## 1 controle controle 1 51.568
## 2 controle controle 2 48.713
## 3 controle controle 3 46.085
## 4 controle controle 4 44.061
## 5 A lanco 1 61.614
## 6 A lanco 2 59.760
## 7 A lanco 3 67.227
## 8 A lanco 4 63.886
## 9 B lanco 1 55.000
## 10 B lanco 2 56.311
## 11 B lanco 3 59.037
## 12 B lanco 4 56.685
## 13 C lanco 1 55.650
## 14 C lanco 2 66.488
## 15 C lanco 3 64.330
## 16 C lanco 4 61.576
## 17 D lanco 1 61.719
## 18 D lanco 2 57.272
## 19 D lanco 3 60.148
## 20 D lanco 4 56.355
## 21 A sulco 1 60.927
## 22 A sulco 2 68.679
## 23 A sulco 3 63.321
## 24 A sulco 4 67.593
## 25 B sulco 1 55.461
## 26 B sulco 2 59.483
## 27 B sulco 3 53.930
## 28 B sulco 4 55.735
## 29 C sulco 1 61.696
## 30 C sulco 2 63.479
## 31 C sulco 3 61.202
## 32 C sulco 4 62.893
## 33 D sulco 1 65.255
## 34 D sulco 2 61.439
## 35 D sulco 3 56.969
## 36 D sulco 4 63.056# Nomes curtos são mais fáceis de manusear.
da <- fat2_adic
# Tabela de frequência dos níveis.
xtabs(~ fonte + modo, data = da)## modo
## fonte lanco sulco controle
## A 4 4 0
## B 4 4 0
## C 4 4 0
## D 4 4 0
## controle 0 0 4A tabela de frequência mostra o que já sabemos: temos um experimento fatorial 4 \(\times\) 2 \(+\) 1 com quatro repetições. Além disso, o nível que corresponde ao controle é o último em cada fator.
xy1 <- resizePanels(
xyplot(prod ~ fonte | modo,
data = da,
xlab = "Fonte de adubo",
ylab = "Produção",
type = c("p", "a"),
layout = c(NA, 1),
scales = list(x = list(relation = "free"))),
w = c(4, 4, 1))
xy2 <- resizePanels(
xyplot(prod ~ modo | fonte,
data = da,
xlab = "Forma de aplicação",
ylab = "Producação",
type = c("p", "a"),
layout = c(NA, 1),
scales = list(x = list(relation = "free"))),
w = c(2, 2, 2, 2, 1))
grid.arrange(xy1, xy2, ncol = 1)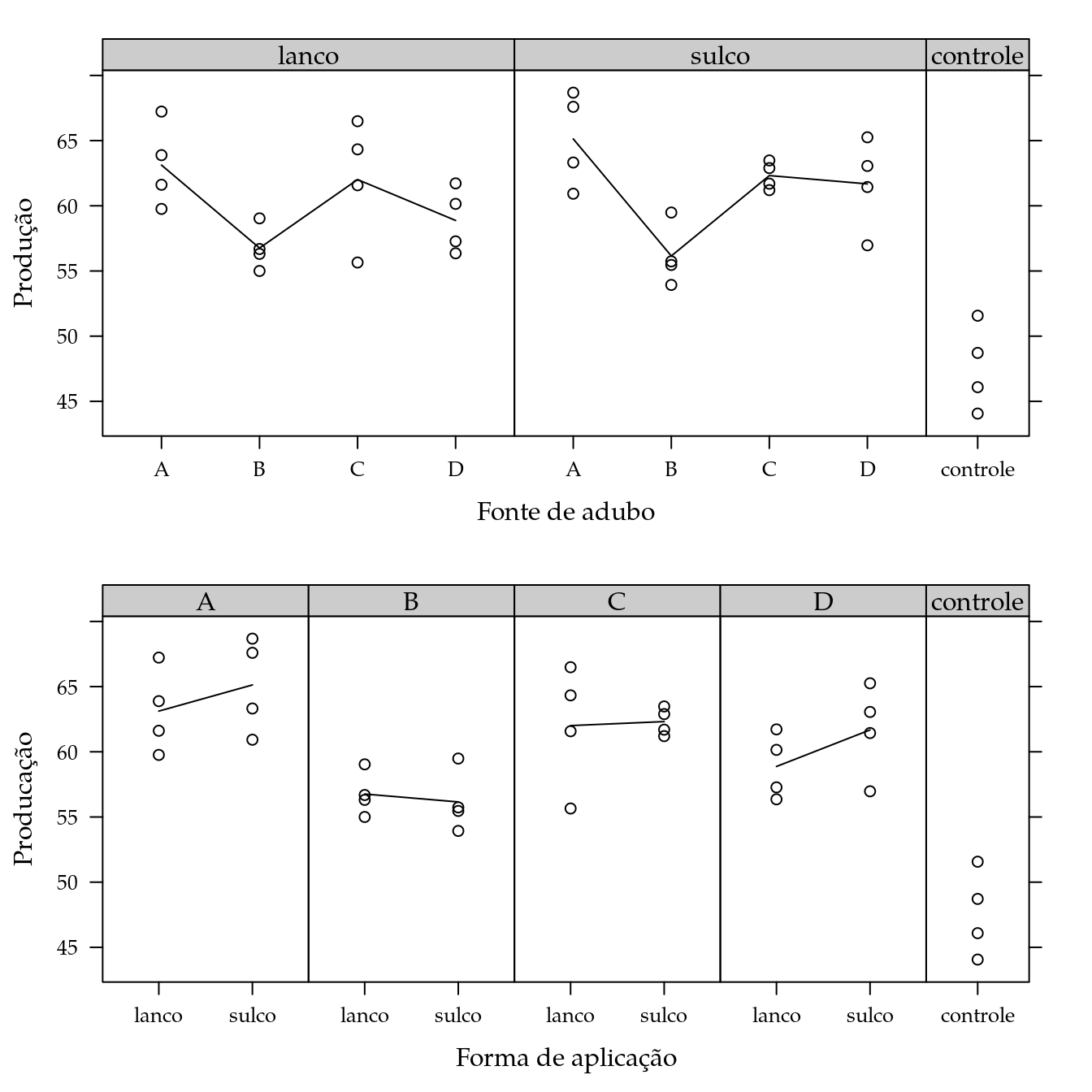
O gráfico de cima mostra que existe uma leve diferença do desempenho da fonte B em relação as outras fontes de adubação. Verifica-se também que o desempenho do controle está abaixo das celas do fatorial. O gráfico de baixo indica haver pouca diferença entre as formas de aplicação. Além disso, não fornece evidências que marcam interação entre os fatores.
Ajuste do modelo e pressupostos
A especificação do modelo via fórmula é a mesma, seja o experimento fatorial completo ou não. No entanto, devido o fatorial com tratamento adicional na realidade ser um fatorial completo com celas perdidas, alguns efeitos não serão estimados, justamente por causa das celas perdidas. Na realidade, as celas não foram perdidas. Quero dizer, não foi um acidente mas algo planejado fazer o experimento da forma como feito.
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.16 3.388 0.3355 0.7998
## fonte 4 871.24 217.811 21.5688 1.173e-07 ***
## modo 1 10.19 10.193 1.0093 0.3251
## fonte:modo 3 14.55 4.849 0.4801 0.6991
## Residuals 24 242.36 10.098
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## [,1]
## (Intercept) 62.5927778
## bloc2 1.4148889
## bloc3 0.3732222
## bloc4 0.3277778
## fonteB -6.3635000
## fonteC -1.1107500
## fonteD -4.2482500
## fontecontrole -15.5150000
## modosulco 2.0082500
## modocontrole NA
## fonteB:modosulco -2.6142500
## fonteC:modosulco -1.7017500
## fonteD:modosulco 0.7980000
## fontecontrole:modosulco NA
## fonteB:modocontrole NA
## fonteC:modocontrole NA
## fonteD:modocontrole NA
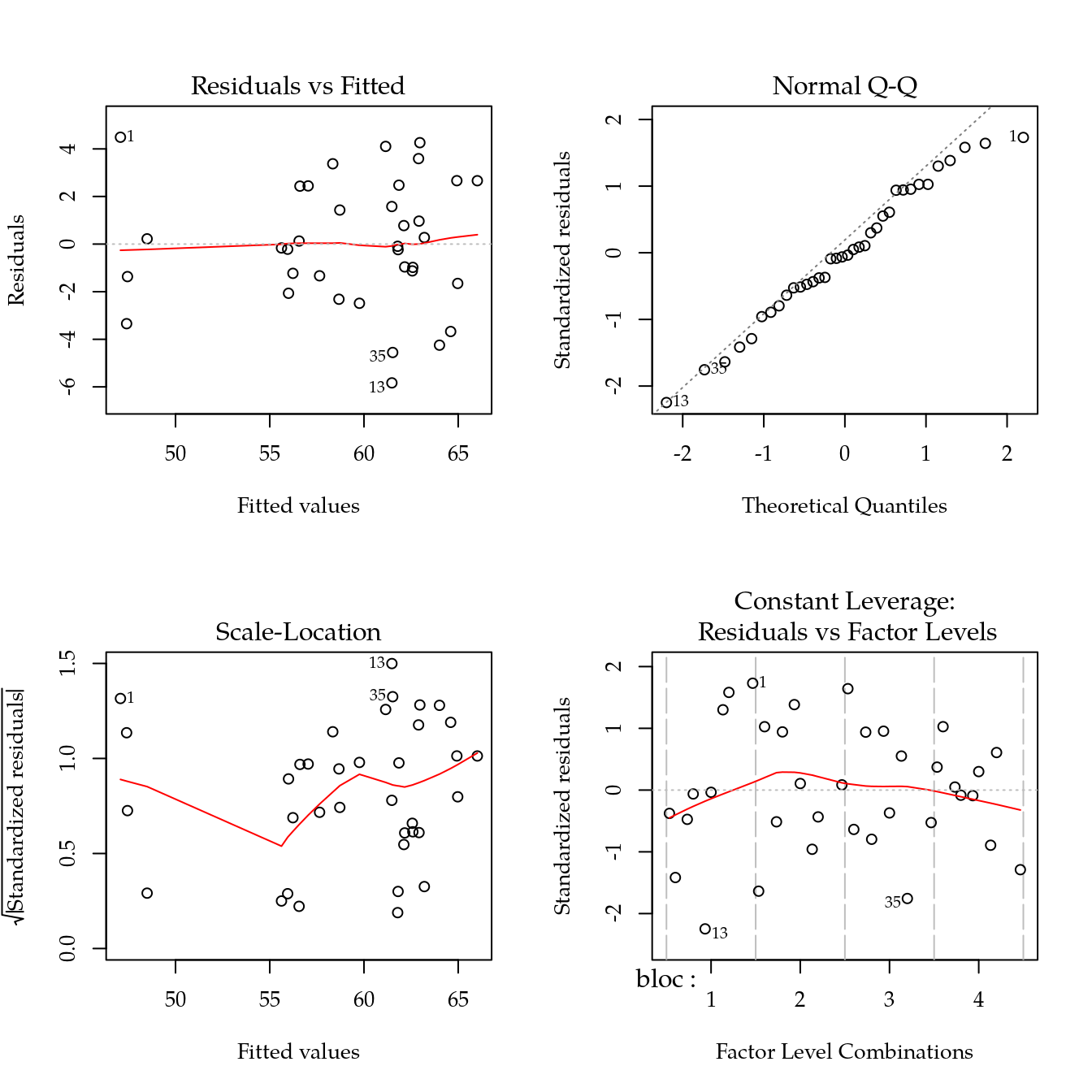
## fontecontrole:modocontrole NAOs gráficos sobre os resíduos não apontam nenhuma evidência contra o atendimentos dos pressupostos.
No quadro de análise de variância mostra não existência de interação. Também não foi significativo o efeito de modo de aplicação. Restou, portanto, apenas o efeito de fonte de adubo.
Note que fonte tem 4 graus de liberdade (GL) enquanto que modo tem apenas 1 e a interação fonte:modo tem 3. Os 4 GL de fonte decorrem do fator ter 5 níveis (4 níveis do fatorial e o controle). Porém, o modo que deveria ter 2, porque têm 3 níveis (2 modos e o controle), fica com apenas 1 porque o efeito de controle já foi acomodado no termo fonte. E a interação resulta o número de combinações de celas que se cruzam nos dois fatores, ou seja, \((4 - 1) \times (2 - 1) = 3\).
As combinações que não existem possuem valor igual a NA no vetor de estimativas. Isso indica que esses efeitos não foram estimados. De fato, só seriam estimados se estas celas experimentais existissem.
Desdobramento das somas de quadrados
Por uma questão didática, será ilustrado como desdobrar a soma de quadrados dentro do quadro de ANOVA. Os 4 GL associados ao termo fonte serão particionados em duas hipóteses: (a) \(H_0\): a diferença entre as fontes A - D é nula (\(\mu_A = \mu_B = \mu_C = \mu_D\)) e (b) \(H_0\): não existe diferença da testemunha para as fontes de adubação (\((\mu_A + \mu_B + \mu_C + \mu_D)/4 = \mu_{\text{controle}}\)).
Para facilmente conseguir esse desdobramento, serão utilizados os contrastes de Helmert. Nestes contrastes, a última coluna representa a nossa hipótese (b).
# Contrastes de Helmert. A última coluna corresponde à "testemunha vs
# demais" nos dois fatores.
contrasts(C(da$modo,
contr = "contr.helmert"))## [,1] [,2]
## lanco -1 -1
## sulco 1 -1
## controle 0 2## [,1] [,2] [,3] [,4]
## A -1 -1 -1 -1
## B 1 -1 -1 -1
## C 0 2 -1 -1
## D 0 0 3 -1
## controle 0 0 0 4A função aov() será usada porque ela permite desdobrar as somas de quadrados dentro do quadro. Para isso é utilizado o argumento split = da função summary() quando operando sobre um objeto de classe aov. Os contrastes de Helmert são passados no argumento contrasts = para a função aov().
# Fonte é o primeiro termo -> Testemunha vs Fonte.
m0 <- aov(prod ~ bloc + fonte * modo,
data = da,
contrasts = list(fonte = "contr.helmert",
modo = "contr.helmert"))
# Desdobramento das somas de quadrados.
summary(m0,
split = list(
"fonte" = list("Entre Fontes" = 1:3,
"Test vs Fontes" = 4)
), expand.split = FALSE)## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.2 3.4 0.336 0.799767
## fonte 4 871.2 217.8 21.569 1.17e-07 ***
## fonte: Entre Fontes 3 256.5 85.5 8.467 0.000517 ***
## fonte: Test vs Fontes 1 614.7 614.7 60.873 4.90e-08 ***
## modo 1 10.2 10.2 1.009 0.325086
## fonte:modo 3 14.5 4.8 0.480 0.699135
## Residuals 24 242.4 10.1
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Agora modo é o primeiro termo -> Testemunha vs Modos (não faz muito
# sentido para esses dados).
m1 <- update(m0, . ~ bloc + modo * fonte)
# Desdobramento das somas de quadrados.
summary(m1,
split = list(
"modo" = list("Entre Modos" = 1,
"Test vs Modos" = 2)
), expand.split = FALSE)## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.2 3.4 0.336 0.799767
## modo 2 624.9 312.5 30.941 2.27e-07 ***
## modo: Entre Modos 1 10.2 10.2 1.009 0.325086
## modo: Test vs Modos 1 614.7 614.7 60.873 4.90e-08 ***
## fonte 3 256.5 85.5 8.467 0.000517 ***
## modo:fonte 3 14.5 4.8 0.480 0.699135
## Residuals 24 242.4 10.1
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Independente da ordem dos termos na fórmula (fonte * modo ou modo * fonte), a soma de quadrado para hipótese da testemunha versus os demais níveis foi o mesmo. Para os dados e o modelo que temos, isso era mesmo para acontecer. A soma de quadrados de modo, assim como de fonte, mudam de um modelo para outro. Isso acontece porque ora quem tem o nível controle é fonte, que entra primeiro no modelo m0, ora é modo que é o primeiro termo no modelo m1. A soma de quadrados da hipótese (a) é o que é exclusiva de fonte e modo. Em ambos os casos, não muda o resultado para a interação, que foi não significativa.
Obtenção das médias ajustadas
As médias ajustadas são médias obtidas por meio de funções lineares dos efeitos estimados. Em experimentos balanceados e com efeito ortogonais, as médias ajustadas coincidem as com as médias amostrais. No entanto, em situações mais gerais, as médias amostrais não são os estimadores corretos das médias populacionais. Exceto para situações muito específicas, trabalhar com as médias ajustadas é sempre o adequado.
# Novamente o mesmo modelo, classe lm.
m0 <- lm(prod ~ bloc + fonte * modo,
data = da)
# Quadro de anova.
anova(m0)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.16 3.388 0.3355 0.7998
## fonte 4 871.24 217.811 21.5688 1.173e-07 ***
## modo 1 10.19 10.193 1.0093 0.3251
## fonte:modo 3 14.55 4.849 0.4801 0.6991
## Residuals 24 242.36 10.098
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Por causa dos efeitos não estimados, essa função retorna as médias
# ajustadas, apenas NAs.
LSmeans(m0, effect = "fonte")## Coefficients:
## estimate se df t.stat p.value
## [1,] NA NA NA NA NA
## [2,] NA NA NA NA NA
## [3,] NA NA NA NA NA
## [4,] NA NA NA NA NA
## [5,] NA NA NA NA NAA função doBy::LSmeans() obtém as médias ajustadas para modelos onde todos os efeitos são estimados. No caso em questão, tem-se NA no vetor de estimativas, o que indica efeitos não estimados. Com isso a obtenção das ls-means deverá ser feita explicitamente.
# Matriz do modelo descartadas as linhas repetidas, caso hajam.
U <- unique(model.matrix(m0))
# Fatores do modelo, removidas as linhas repetidas também.
db <- unique(subset(da, select = all.vars(formula(m0)[-2])))
# Os tamanhos são iguais? Devem ser.
nrow(db) == nrow(U)## [1] TRUE# Criando a matriz de LSmeans de forma alternativa.
agr <- aggregate(U ~ fonte + modo,
data = db,
FUN = mean)
# Índices para separar a parte tabela da parte matriz.
i <- 1:2
pred <- agr[, i]
X <- as.matrix(agr[, -i])
dim(X)## [1] 9 18# write.table(format(t(X), justify = "right"),
# row.names = FALSE,
# col.names = FALSE,
# quote = FALSE)
# Matriz para obter as médias ajustadas ao multiplicas pelas estimativas
# dos efeitos.
print.table(local({X[X == 0] <- "."; colnames(X) <- NULL; t(X)}),
justify = "centre")## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 1 1 1 1 1 1 1 1 1
## [2,] 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## [3,] 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## [4,] 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## [5,] . 1 . . . 1 . . .
## [6,] . . 1 . . . 1 . .
## [7,] . . . 1 . . . 1 .
## [8,] . . . . . . . . 1
## [9,] . . . . 1 1 1 1 .
## [10,] . . . . . . . . 1
## [11,] . . . . . 1 . . .
## [12,] . . . . . . 1 . .
## [13,] . . . . . . . 1 .
## [14,] . . . . . . . . .
## [15,] . . . . . . . . .
## [16,] . . . . . . . . .
## [17,] . . . . . . . . .
## [18,] . . . . . . . . 1Para poder obter as médias ajustadas tem-se que resolver um problema de inconsistência de dimensão. O vetor de efeitos estimados, coef(m0), com entradas para os efeitos não estimados que estão como NA. A matriz de covariância das estimativas, contém dimensão igual ao número de efeitos estimados. Os dois devem ter dimensão compatível. Para tornar compatível, serão removidos os NA do vetor. Essa modificação será feita nas entranhas do objeto m0.
# ATTENTION: Dentro do objeto que guarda o ajuste está sendo removido os
# NA de dentro do vetor de parâmetros estimados. Isso pode produzir
# efeitos colaterais. Não é aconselhável alterar o conteúdo de objetos
# assim.
m0$coefficients <- coef(m0)[!is.na(coef(m0))]
# As dimensões agora são compatíveis.
dim(vcov(m0))## [1] 12 12## [1] 12## [1] 9 12# Estima as médias de cada cela experimental, com intervalo de
# confiança.
ci <- confint(glht(m0, linfct = X), calpha = univariate_calpha())
cbind(pred, ci$confint)## fonte modo Estimate lwr upr
## 1 A lanco 63.12175 59.84242 66.40108
## 2 B lanco 56.75825 53.47892 60.03758
## 3 C lanco 62.01100 58.73167 65.29033
## 4 D lanco 58.87350 55.59417 62.15283
## 5 A sulco 65.13000 61.85067 68.40933
## 6 B sulco 56.15225 52.87292 59.43158
## 7 C sulco 62.31750 59.03817 65.59683
## 8 D sulco 61.67975 58.40042 64.95908
## 9 controle controle 47.60675 44.32742 50.88608# Médias amostrais das celas experimentais (apenas para conferir os
# resultados).
aggregate(prod ~ fonte + modo, data = da, mean)## fonte modo prod
## 1 A lanco 63.12175
## 2 B lanco 56.75825
## 3 C lanco 62.01100
## 4 D lanco 58.87350
## 5 A sulco 65.13000
## 6 B sulco 56.15225
## 7 C sulco 62.31750
## 8 D sulco 61.67975
## 9 controle controle 47.60675Análise do modelo reduzido
Como não houve interação e nem diferença entre modos de aplicação, o modelo pode ser simplificado pelo abandono destes termos de efeito nulo. Com isso, desaparecem as dificuldades causadas pelo fatorial incompleto pois entramos em uma situação com um único fator. Tem o detalhe o número de repetições não é igual, mas isso não é um complicador diante dos recursos que temos.
m1 <- update(m0, prod ~ bloc + fonte)
# Testa se o conjunto de termos abandados tem efeito nulo.
anova(m1, m0)## Analysis of Variance Table
##
## Model 1: prod ~ bloc + fonte
## Model 2: prod ~ bloc + fonte * modo
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 28 267.10
## 2 24 242.36 4 24.739 0.6124 0.6577# Quadro de anova.
anova(m1)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.16 3.388 0.3552 0.7857
## fonte 4 871.24 217.811 22.8330 1.796e-08 ***
## Residuals 28 267.10 9.539
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativa dos parâmetros e medidas de ajuste.
summary(m1)##
## Call:
## lm(formula = prod ~ bloc + fonte, data = da)
##
## Residuals:
## Min 1Q Median 3Q Max
## -5.9853 -1.5202 -0.0058 2.1867 5.5073
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 63.5969 1.4097 45.113 < 2e-16 ***
## bloc2 1.4149 1.4560 0.972 0.3395
## bloc3 0.3732 1.4560 0.256 0.7996
## bloc4 0.3278 1.4560 0.225 0.8235
## fonteB -7.6706 1.5443 -4.967 3.03e-05 ***
## fonteC -1.9616 1.5443 -1.270 0.2145
## fonteD -3.8492 1.5443 -2.493 0.0189 *
## fontecontrole -16.5191 1.8914 -8.734 1.75e-09 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.089 on 28 degrees of freedom
## Multiple R-squared: 0.7674, Adjusted R-squared: 0.7093
## F-statistic: 13.2 on 7 and 28 DF, p-value: 2.172e-07# Média ajustadas. Desse objeto é extraído a matriz para obter médias
# ajustadas.
lsm <- LSmeans(m1, effect = "fonte")
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 64.1259 1.0920 28.0000 58.7246 0
## [2,] 56.4552 1.0920 28.0000 51.7000 0
## [3,] 62.1643 1.0920 28.0000 56.9282 0
## [4,] 60.2766 1.0920 28.0000 55.1995 0
## [5,] 47.6067 1.5443 28.0000 30.8276 0# Matriz para obter médias ajustdas.
X <- lsm$L
# Tabela com os níveis dos fatores.
pred <- lsm$grid
# Comparações múltiplas por contrantes de Tukey. ATTENTION: são
# contrastes de Tukey (comparações aos pares) e não o teste de Tukey. O
# Teste de Tukey para essas médias iria envolver aproximações do tipo
# média geométrica do número de repetições. A aproximação não
# funcionaria bem.
comp <- summary(glht(m1, linfct = mcp(fonte = "Tukey")))
# Representação com letras (compact letter display).
cld <- cld(comp, decreasing = TRUE)
cld## A B C D controle
## "a" "b" "a" "ab" "c"# Intervalos de confiança para as médias.
ci <- confint(glht(m1, linfct = X), calpha = univariate_calpha())
ci <- as.data.frame(ci$confint)
# Junta todas as colunas em uma tabela só.
pred <- cbind(pred, ci, cld = cld$mcletters$Letters)
pred$fonte <- factor(pred$fonte, levels(da$fonte))
pred## fonte Estimate lwr upr cld
## 1 A 64.12587 61.88906 66.36269 a
## 2 B 56.45525 54.21844 58.69206 b
## 3 C 62.16425 59.92744 64.40106 a
## 4 D 60.27662 58.03981 62.51344 ab
## 5 controle 47.60675 44.44342 50.77008 c# Gráfico de segmentos.
segplot(reorder(fonte, Estimate) ~ lwr + upr,
centers = Estimate,
data = pred,
xlab = "Produção",
ylab = "Fontes de abubo",
cld = pred$cld,
draw = FALSE) +
layer(panel.text(x = centers,
y = z,
pos = 3,
labels = sprintf("%0.2f %s",
centers,
cld)))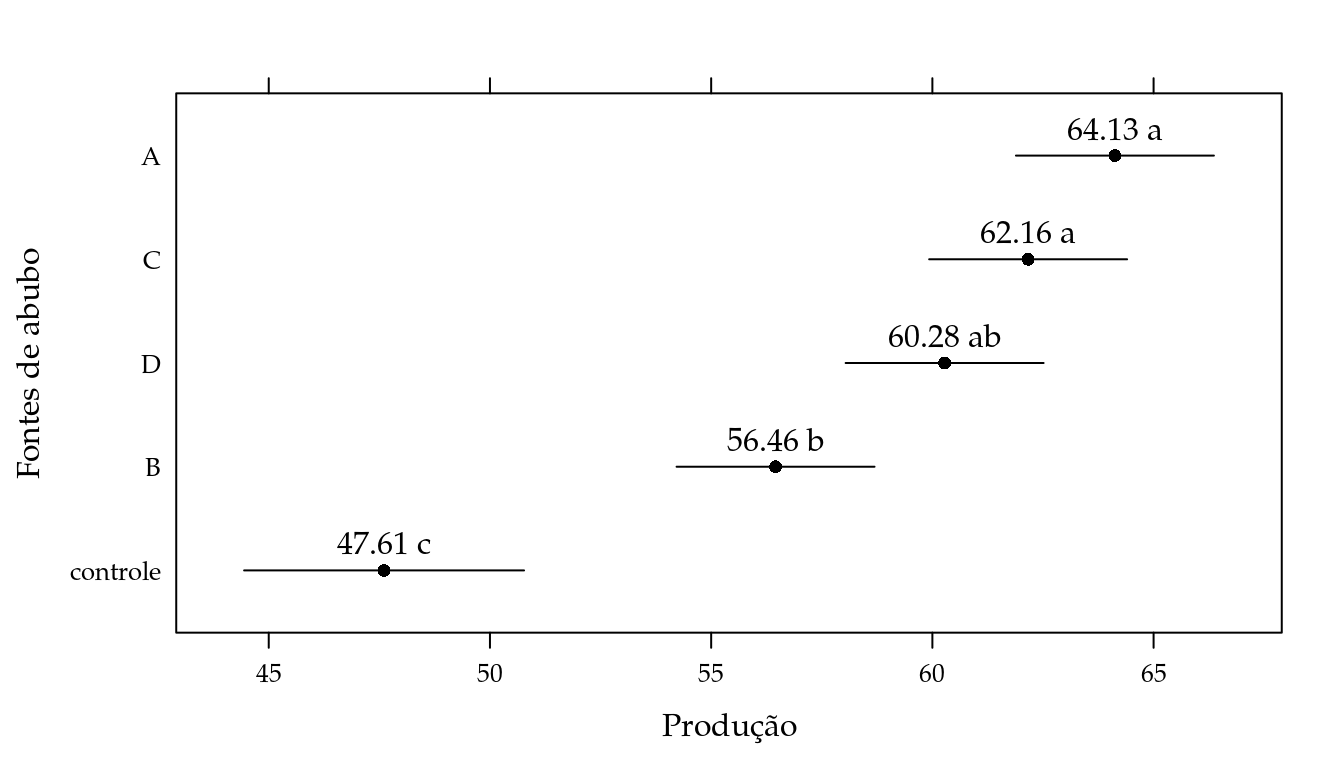
O gráfico de segmentos resume os resultados do experimento. Verifica-se por ele que existem diferenças entre as fontes de adubação.
Informações da sessão
## Atualizado em 11 de julho de 2019.
##
## R version 3.6.1 (2019-07-05)
## Platform: x86_64-pc-linux-gnu (64-bit)
## Running under: Ubuntu 18.04.2 LTS
##
## Matrix products: default
## BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.7.1
## LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.7.1
##
## locale:
## [1] LC_CTYPE=pt_BR.UTF-8 LC_NUMERIC=C
## [3] LC_TIME=pt_BR.UTF-8 LC_COLLATE=en_US.UTF-8
## [5] LC_MONETARY=pt_BR.UTF-8 LC_MESSAGES=en_US.UTF-8
## [7] LC_PAPER=pt_BR.UTF-8 LC_NAME=C
## [9] LC_ADDRESS=C LC_TELEPHONE=C
## [11] LC_MEASUREMENT=pt_BR.UTF-8 LC_IDENTIFICATION=C
##
## attached base packages:
## [1] stats graphics grDevices utils datasets methods
## [7] base
##
## other attached packages:
## [1] multcomp_1.4-10 TH.data_1.0-10 MASS_7.3-51.4
## [4] survival_2.44-1.1 mvtnorm_1.0-11 doBy_4.6-2
## [7] gridExtra_2.3 EACS_0.0-7 wzRfun_0.91
## [10] captioner_2.2.3 latticeExtra_0.6-28 RColorBrewer_1.1-2
## [13] lattice_0.20-38 knitr_1.23
##
## loaded via a namespace (and not attached):
## [1] zoo_1.8-6 tidyselect_0.2.5 xfun_0.8
## [4] remotes_2.1.0 purrr_0.3.2 splines_3.6.1
## [7] testthat_2.1.1 htmltools_0.3.6 usethis_1.5.1
## [10] yaml_2.2.0 rlang_0.4.0 pkgbuild_1.0.3
## [13] pkgdown_1.3.0 pillar_1.4.2 glue_1.3.1
## [16] withr_2.1.2 sessioninfo_1.1.1 plyr_1.8.4
## [19] stringr_1.4.0 commonmark_1.7 gtable_0.3.0
## [22] devtools_2.1.0 codetools_0.2-16 memoise_1.1.0
## [25] evaluate_0.14 callr_3.3.0 ps_1.3.0
## [28] Rcpp_1.0.1 backports_1.1.4 desc_1.2.0
## [31] pkgload_1.0.2 fs_1.3.1 digest_0.6.20
## [34] stringi_1.4.3 processx_3.4.0 dplyr_0.8.3
## [37] grid_3.6.1 rprojroot_1.3-2 cli_1.1.0
## [40] tools_3.6.1 sandwich_2.5-1 magrittr_1.5
## [43] tibble_2.1.3 crayon_1.3.4 pkgconfig_2.0.2
## [46] Matrix_1.2-17 xml2_1.2.0 prettyunits_1.0.2
## [49] assertthat_0.2.1 rmarkdown_1.13 roxygen2_6.1.1
## [52] rstudioapi_0.10 R6_2.4.0 compiler_3.6.1