Ajuste da Curva de Retenção de Água do Solo
Walmes M. Zeviani & Milson E. Serafim
Source:vignettes/teca_cra.Rmd
teca_cra.RmdAnálise Exploratória
#-----------------------------------------------------------------------
# Carrega os pacotes necessários.
library(lattice)
library(latticeExtra)
library(nlme)
library(plyr)
library(wzRfun)
library(EACS)
library(captioner)
figs <- captioner(prefix = "Figura")
tbls <- captioner(prefix = "Tabela")
#-----------------------------------------------------------------------
# Análise exploratório dos dados.
# Pra facilitar o manuseio, vamos usar um nome curto.
cra <- teca_cra
str(cra)
cra$tens[cra$tens == 0] <- 0.1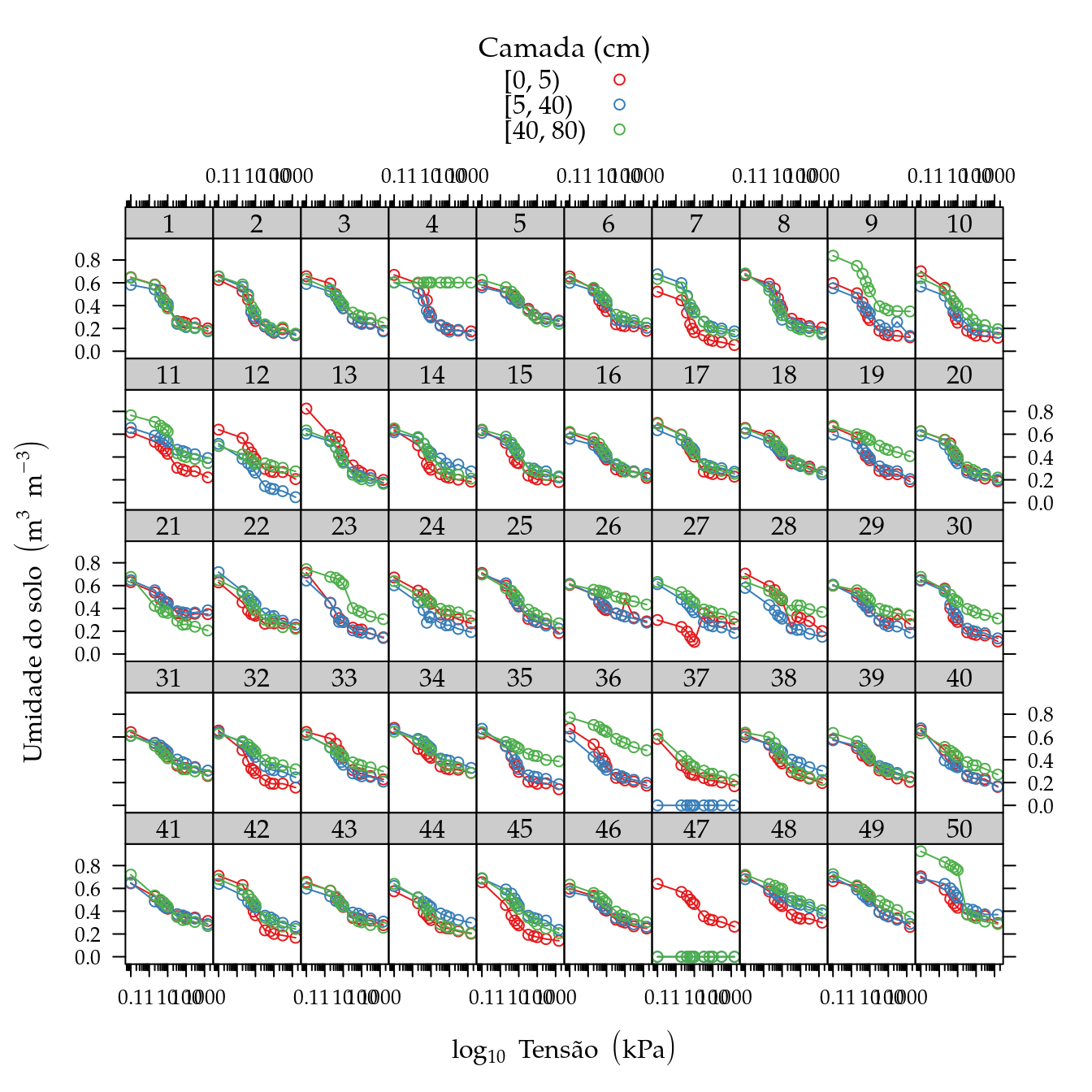
Figura 1: Umidade do solo em função da tensão matricial para 50 sítios cultivados com teca em 3 camadas do solo.
#-----------------------------------------------------------------------
# Remove curvas atípicas.
del <- with(cra, {
(loc == 4 & cam == levels(cam)[3]) |
(loc == 27 & cam == levels(cam)[1]) |
(loc == 37 & cam == levels(cam)[2]) |
(loc == 47 & cam == levels(cam)[2]) |
(loc == 47 & cam == levels(cam)[3])
})
cra <- droplevels(cra[!del, ])Ajuste da Curva de Retenção de Água do Solo
O ajuste foi feito considerando a seguinte parametrização do modelo van Genuchten
\[ U(x) = U_r + \frac{U_s - U_r}{(1 + \exp\{n(\alpha + x)\})^{1 - 1/n}} \]
em que \(U\) é umidade (m3 m-3) do solo, \(x\) é o log na base 10 da tensão matricial aplicada (kPa), \(U_r\) é a umidade residual (assíntota inferior), \(U_s\) é a umidade de satuação (assíntota superior), \(\alpha\) e \(n\) são parâmetros empíricos de forma da curva de retenção de água. Uma vez conhecido valores para as quatidades mencionadas, são obtidos
\[ \begin{align*} S &= -n\cdot \frac{U_s - U_r}{(1 + 1/m)^{m + 1}} \newline I &= -\alpha - \log(m)/n \newline U_I &= U(x = I) \end{align*} \]
em que \(S\) é a taxa no ponto de inflexão, parâmetro que é tido como central para avaliação da qualidade física do solo, bem como \(I\) que corresponde ao log da tensão no ponto de inflexão da curva de retenção de água do solo. A umidade correspondente a tensão no ponto de inflexão é representada por \(U_I\).
O modelo foi ajustado aos dados usando o método de mínimos quadrados com algoritmo de Newton-Raphson para encontrar estimativas para os parâmetros.
#-----------------------------------------------------------------------
# Ajuste da CRA.
xyplot(umid ~ tens,
# data = subset(cra, loc == 40 & cam == "[40, 80)"),
data = cra,
scales = list(x = list(log = 10)),
xscale.components = xscale.components.log10ticks,
ylab = expression("Umidade do solo" ~ (m^{3} ~ m^{-3})),
xlab = expression(log[10] ~ "Tensão" ~ (kPa)))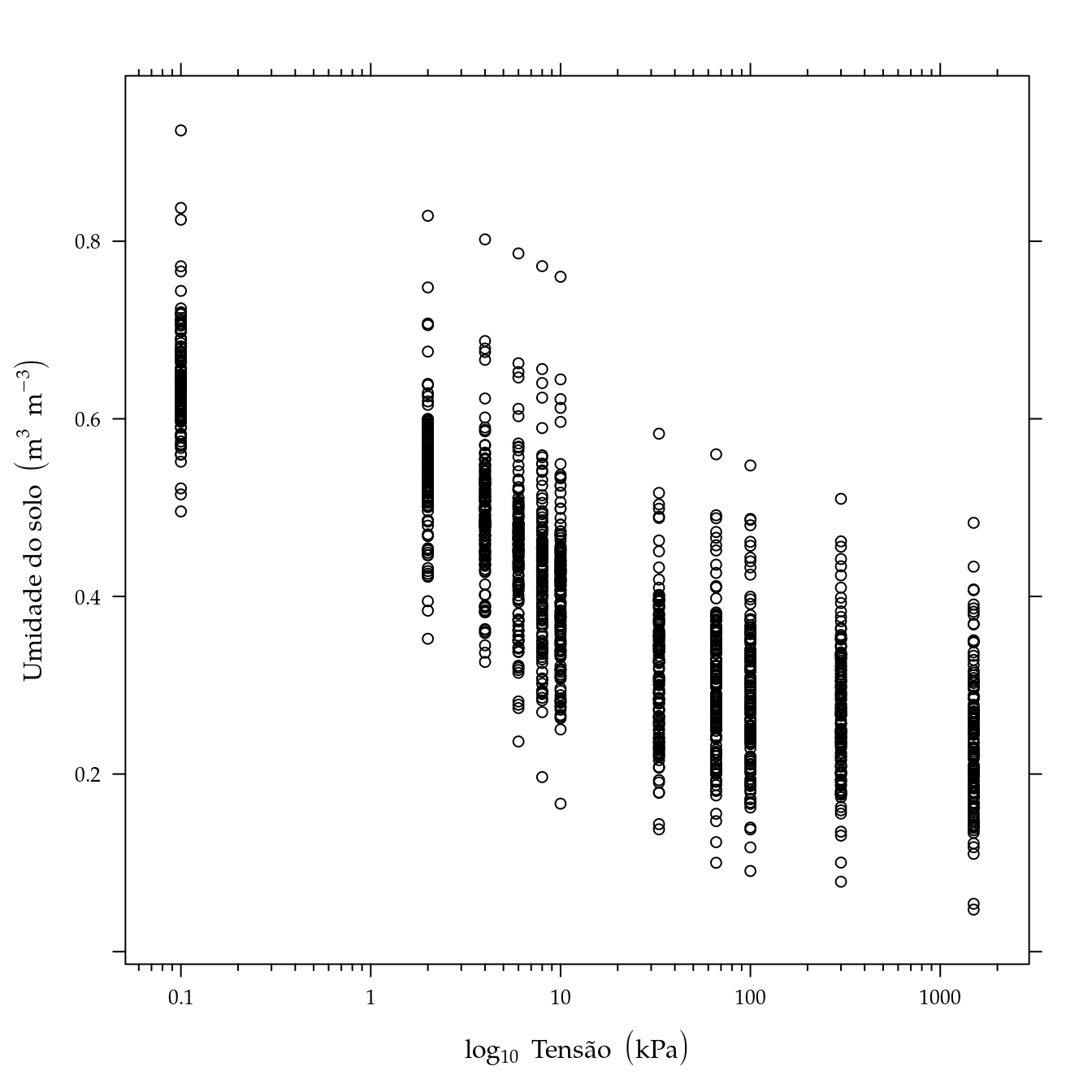
# Logaritmo na base 10 da tensão matricial.
cra$ltens <- log10(cra$tens)
# Expressão do modelo van Genuchten.
model <- umid ~ Ur + (Us - Ur)/(1 + exp(n * (alp + ltens)))^(1 - 1/n)
# Valores iniciais para os parâmetros da curva.
start <- list(Ur = 0.3, Us = 0.6, alp = -0.5, n = 4)
n00 <- nls(model, data = cra, start = start)
coef(n00)## Ur Us alp n
## 0.2390291 0.6519457 -0.4708593 2.4380958#-----------------------------------------------------------------------
# Ajustar para cada unidade experimental, 50 loc x 3 cam = 150 ue, se
# não tivesse sido removido algumas curvas.
cra$ue <- with(cra, interaction(loc, cam, drop = TRUE))
nlevels(cra$ue)## [1] 145db <- groupedData(umid ~ ltens | ue,
data = cra, order.groups = FALSE)
n0 <- nlsList(model = model, data = db,
start = as.list(coef(n00)))
c0 <- coef(n0)
pairs(c0)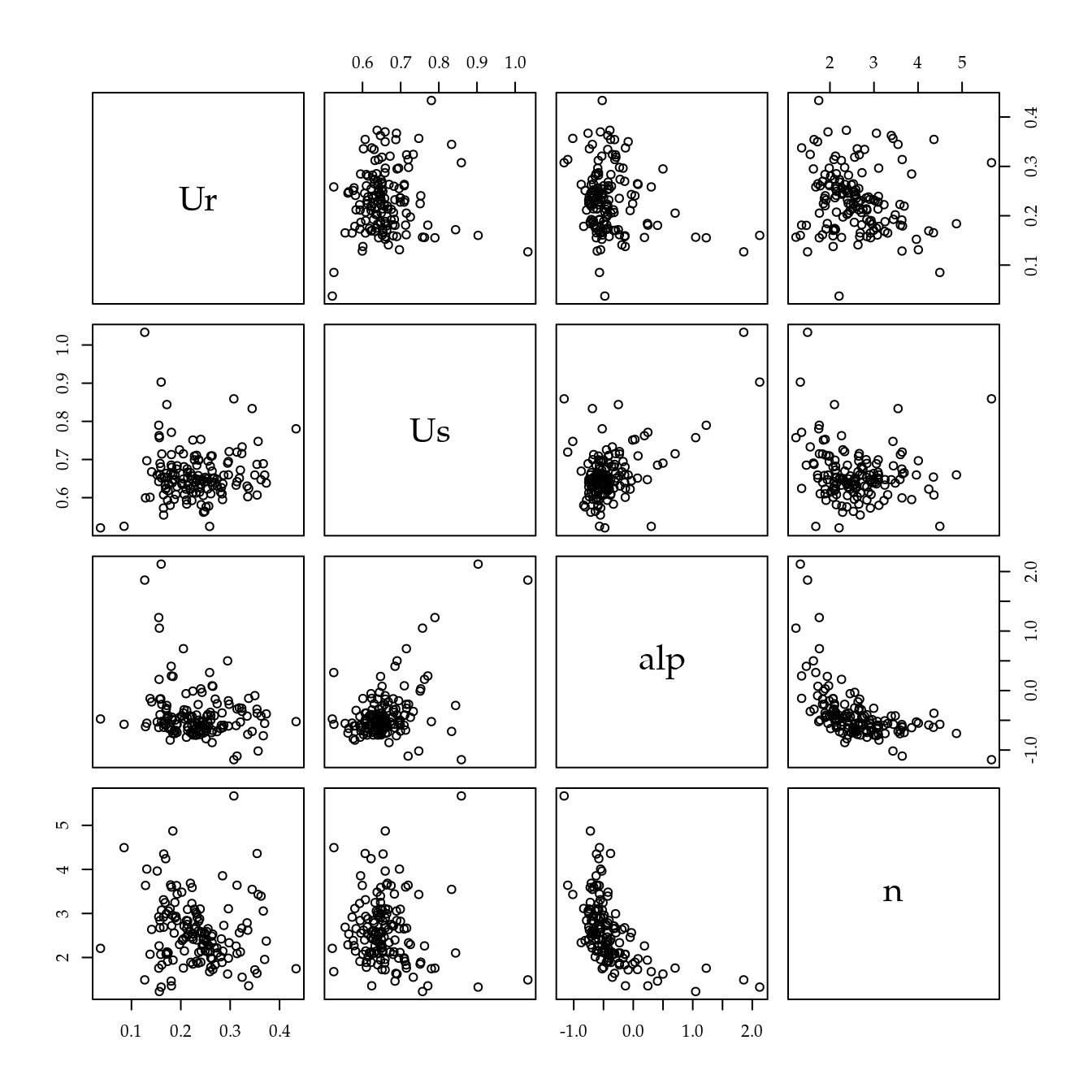
# Alguma curva sem ajustar?
sum(!complete.cases(c0))## [1] 1plot(augPred(n0),
strip = FALSE,
as.table = TRUE,
ylab = expression("Umidade do solo" ~ (m^{3} ~ m^{-3})),
xlab = expression(log[10] ~ "Tensão" ~ (kPa)))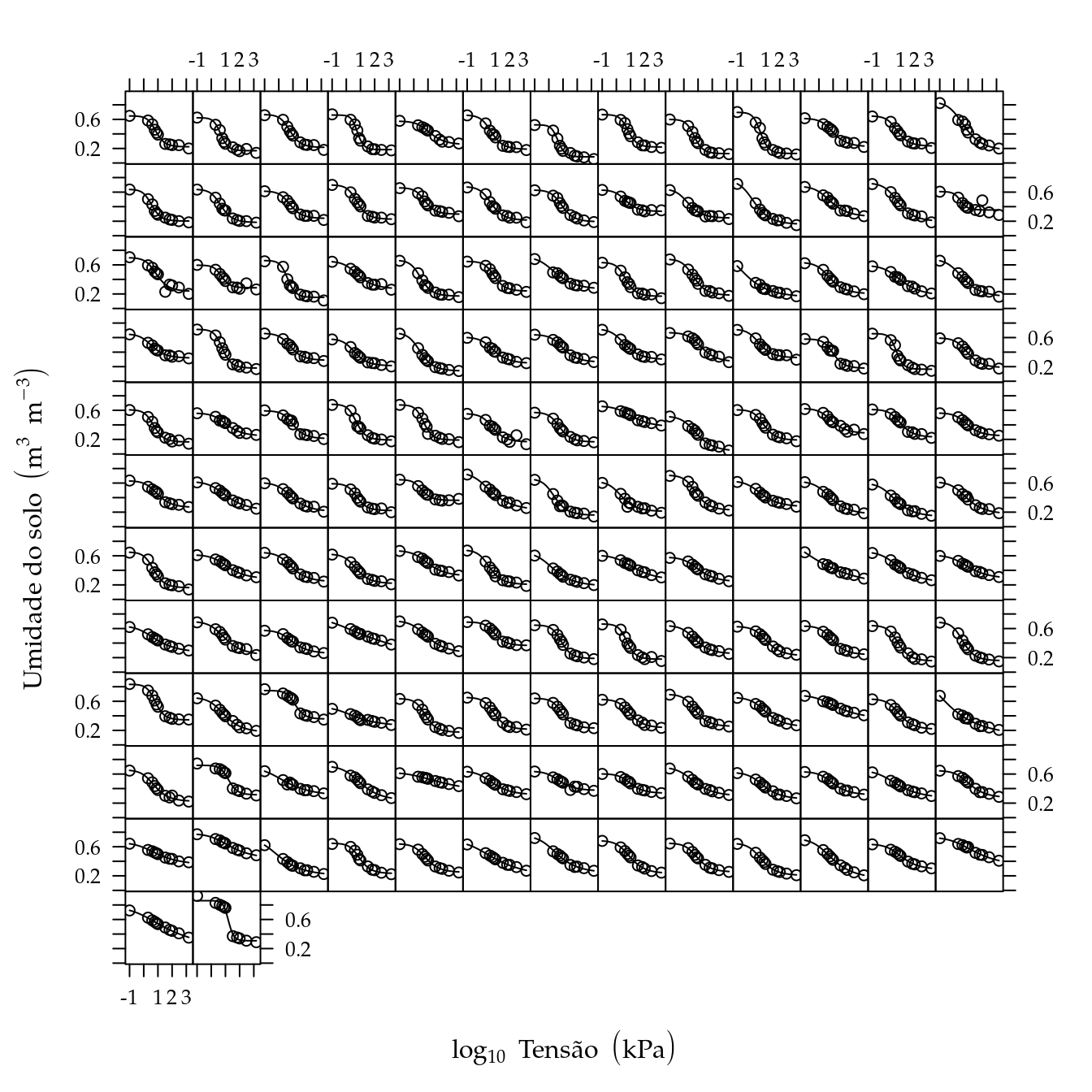
#-----------------------------------------------------------------------
# Determinar os demais parâmetros da curva de água do solo.
params <- as.data.frame(
do.call(rbind, strsplit(rownames(c0),
split = "\\.")))
names(params) <- c("loc", "cam")
params <- transform(params,
loc = as.integer(loc),
cam = factor(cam, levels = levels(cra$cam)))
params <- na.omit(cbind(params, c0))
params <- within(params, {
m <- 1 - 1/n
d <- Us - Ur
S <- -d * n * (1 + 1/m)^(-m - 1)
I <- -alp - log(m)/n
Ui <- Ur + (Us - Ur)/(1 + exp(n * (alp + I)))^(1 - 1/n)
cad <- Ui - Ur
rm(d, m)
})
str(params)## 'data.frame': 144 obs. of 10 variables:
## $ loc: int 1 12 23 34 45 47 48 49 50 2 ...
## $ cam: Factor w/ 3 levels "[0, 5)","[5, 40)",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ Ur : num 0.223 0.169 0.214 0.184 0.251 ...
## $ Us : num 0.648 0.622 0.667 0.66 0.576 ...
## $ alp: num -0.724 -0.578 -0.608 -0.72 -0.809 ...
## $ n : num 3.59 4.24 3.09 4.87 2.38 ...
## $ cad: num 0.227 0.239 0.245 0.249 0.182 ...
## $ Ui : num 0.45 0.408 0.459 0.433 0.433 ...
## $ I : num 0.815 0.642 0.734 0.767 1.037 ...
## $ S : num -0.341 -0.439 -0.306 -0.537 -0.159 ...
## - attr(*, "na.action")= 'omit' Named int 88
## ..- attr(*, "names")= chr "40.[5, 40)"# addmargins(xtabs(~loc + cam, data = params))
params <- arrange(params, loc, cam)
splom(params[, -(1:2)], type = c("p", "r"))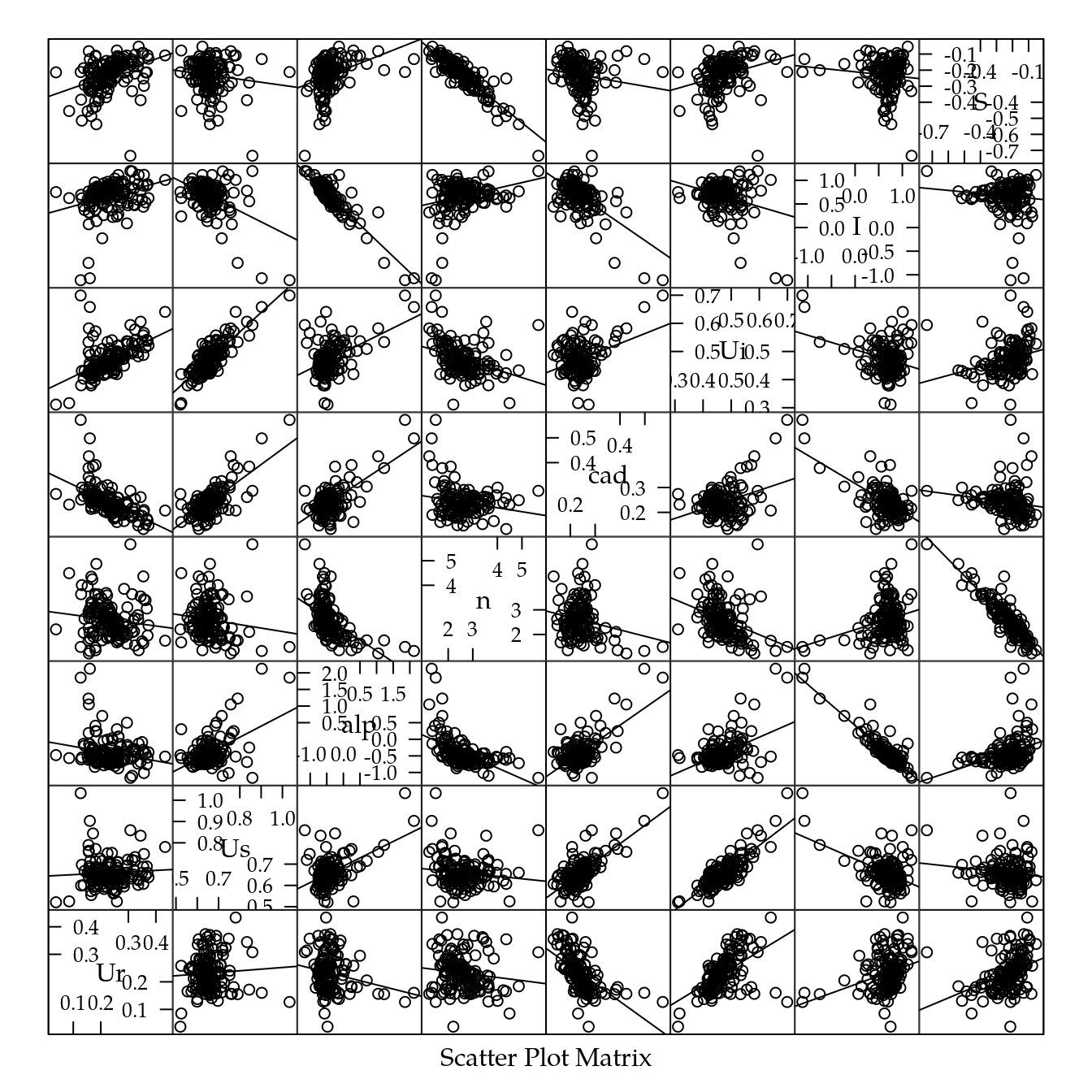
#-----------------------------------------------------------------------
# Salvando em um objeto no pacote.
teca_crapar <- params
str(teca_crapar)## 'data.frame': 144 obs. of 10 variables:
## $ loc: int 1 1 1 2 2 2 3 3 3 4 ...
## $ cam: Factor w/ 3 levels "[0, 5)","[5, 40)",..: 1 2 3 1 2 3 1 2 3 1 ...
## $ Ur : num 0.223 0.178 0.188 0.131 0.165 ...
## $ Us : num 0.648 0.58 0.647 0.697 0.573 ...
## $ alp: num -0.724 -0.833 -0.722 -0.548 -0.547 ...
## $ n : num 3.59 3.11 3.25 4.01 2.92 ...
## $ cad: num 0.227 0.217 0.247 0.3 0.222 ...
## $ Ui : num 0.45 0.396 0.435 0.431 0.387 ...
## $ I : num 0.815 0.957 0.835 0.62 0.691 ...
## $ S : num -0.341 -0.273 -0.329 -0.515 -0.257 ...
## - attr(*, "na.action")= 'omit' Named int 88
## ..- attr(*, "names")= chr "40.[5, 40)"Informações da sessão
## Atualizado em 11 de julho de 2019.
##
## R version 3.6.1 (2019-07-05)
## Platform: x86_64-pc-linux-gnu (64-bit)
## Running under: Ubuntu 18.04.2 LTS
##
## Matrix products: default
## BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.7.1
## LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.7.1
##
## locale:
## [1] LC_CTYPE=pt_BR.UTF-8 LC_NUMERIC=C
## [3] LC_TIME=pt_BR.UTF-8 LC_COLLATE=en_US.UTF-8
## [5] LC_MONETARY=pt_BR.UTF-8 LC_MESSAGES=en_US.UTF-8
## [7] LC_PAPER=pt_BR.UTF-8 LC_NAME=C
## [9] LC_ADDRESS=C LC_TELEPHONE=C
## [11] LC_MEASUREMENT=pt_BR.UTF-8 LC_IDENTIFICATION=C
##
## attached base packages:
## [1] stats graphics grDevices utils datasets methods
## [7] base
##
## other attached packages:
## [1] plyr_1.8.4 nlme_3.1-140 EACS_0.0-7
## [4] wzRfun_0.91 captioner_2.2.3 latticeExtra_0.6-28
## [7] RColorBrewer_1.1-2 lattice_0.20-38 knitr_1.23
##
## loaded via a namespace (and not attached):
## [1] Rcpp_1.0.1 highr_0.8 compiler_3.6.1
## [4] prettyunits_1.0.2 remotes_2.1.0 tools_3.6.1
## [7] testthat_2.1.1 digest_0.6.20 pkgbuild_1.0.3
## [10] pkgload_1.0.2 evaluate_0.14 memoise_1.1.0
## [13] rlang_0.4.0 rstudioapi_0.10 cli_1.1.0
## [16] commonmark_1.7 yaml_2.2.0 pkgdown_1.3.0
## [19] xfun_0.8 withr_2.1.2 stringr_1.4.0
## [22] roxygen2_6.1.1 xml2_1.2.0 desc_1.2.0
## [25] fs_1.3.1 devtools_2.1.0 rprojroot_1.3-2
## [28] grid_3.6.1 glue_1.3.1 R6_2.4.0
## [31] processx_3.4.0 rmarkdown_1.13 sessioninfo_1.1.1
## [34] callr_3.3.0 magrittr_1.5 usethis_1.5.1
## [37] backports_1.1.4 ps_1.3.0 htmltools_0.3.6
## [40] MASS_7.3-51.4 assertthat_0.2.1 stringi_1.4.3
## [43] crayon_1.3.4