Desenvolvimento de mudas de teca em resposta à doses de Cálcio
Milson E. Serafim & Walmes M. Zeviani
11 de julho de 2019
Source:vignettes/gen_teca.Rmd
gen_teca.RmdDescrição e Análise Exploratória
#-----------------------------------------------------------------------
# Carrega os pacotes necessários.
rm(list = ls())
library(lattice)
library(latticeExtra)
library(grid)
library(gridExtra)
library(plyr)
library(doBy)
library(multcomp)
library(wzRfun)
library(EACS)
# Obtém o intervalo em que a muda morreu.
deathint <- function(data, timevar, respvar) {
o <- order(data[, timevar])
x <- data[o, timevar]
y <- data[o, respvar]
nna <- !is.na(y)
if (sum(nna)) {
i <- which(nna)
imax <- max(i)
st <- 3 # Censura intervalar.
int <- x[imax + 0:1]
} else {
st <- 2 # Censura a esquerda.
int <- rep(min(x), 2)
}
if (is.na(int[2])) {
int[2] <- int[1]
st <- 0 # Censura a direita.
}
r <- c(int, st)
names(r) <- c("left", "right", "status")
return(r)
}Os gráficos abaixo exibem a curva de crescimento das mudas em altura e diâmetro à altura do colo para cada um dos genótipos (paineis) e cada dose de cálcio (cores das linhas). Verifica-se que existem mudas com interrupção das medidas o que é resultado da morte das mudas.
#-----------------------------------------------------------------------
# Análise gráfica exploratória para altura e diâmetro.
# Estrutura dos dados.
str(gen_teca)## 'data.frame': 5800 obs. of 7 variables:
## $ gen : Factor w/ 29 levels "1","2","3","4",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ dose: num 0 0.0184 0.0368 0.0736 0.1472 ...
## $ data: Date, format: "2016-06-15" ...
## $ dac : num 38.6 40.2 43 40 39.8 23.8 33.9 37 40 41.8 ...
## $ alt : num 101 61.7 130 128 128 126 132 121 129 140 ...
## $ mspa: num NA NA NA NA NA NA NA NA NA NA ...
## $ msr : num NA NA NA NA NA NA NA NA NA NA ...# Nomes curtos são mais fáceis de manipular.
da <- gen_teca
# Supondo que a primeira medida foi no dia 2.
da$dias <- as.numeric(da$data - min(da$data) + 2)
xyplot(alt ~ dias | gen,
data = da,
groups = dose,
type = "l",
xlab = "Tempo após a primeira avaliação (dias)",
ylab = "Altura das mudas de teca (mm)",
as.table = TRUE,
scales = list(y = list(log = FALSE)))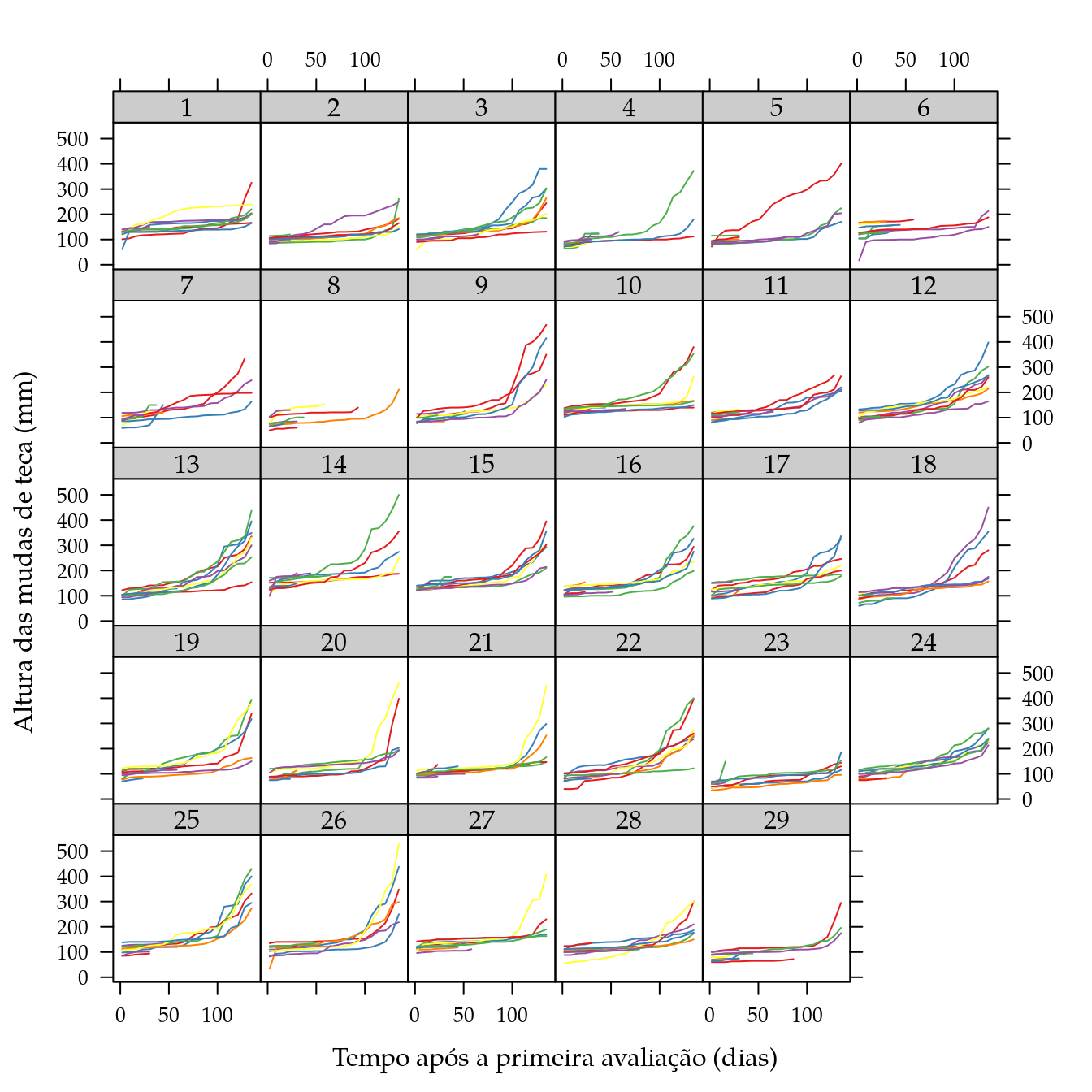
xyplot(dac ~ dias | gen,
data = da,
groups = dose,
type = "l",
xlab = "Tempo após a primeira avaliação (dias)",
ylab = "Diâmetro à altura do colo das mudas de teca (mm)",
as.table = TRUE)
Para utilizar o fator métrico dose de cálcio nas análises (dose), é recomendável que se utilize uma transformação na escala das doses de forma a obter um espaçamento mais equidistante entre níveis, o que evita problemas de alancagem para ajustes de modelos de regressão. Em vista disso, definiu-se como critério minimizar a variância entre as diferenças de doses consecutivas na escala unitária por meio de uma função potência das doses transformadas. Na escala unitária, a menor é 0 e a maior é 1. O valor obtido para transformar a dose foi de 0.3.
#-----------------------------------------------------------------------
# Trabalhando com a dose.
# Criando dose categórico.
x <- unique(sort(da$dose))
da$dos <- factor(da$dose,
levels = x,
labels = seq_along(x) - 1)
# Variância das distâncias entre níveis em escala unitária.
esp <- function(p) {
u <- x^p
u <- (u - min(u))
u <- u/max(u)
var(diff(u))
}
# Otimiza para obter o valor de potência para maior uniformidade.
op <- optimize(f = esp, interval = c(0, 1))
op$minimum## [1] 0.3064936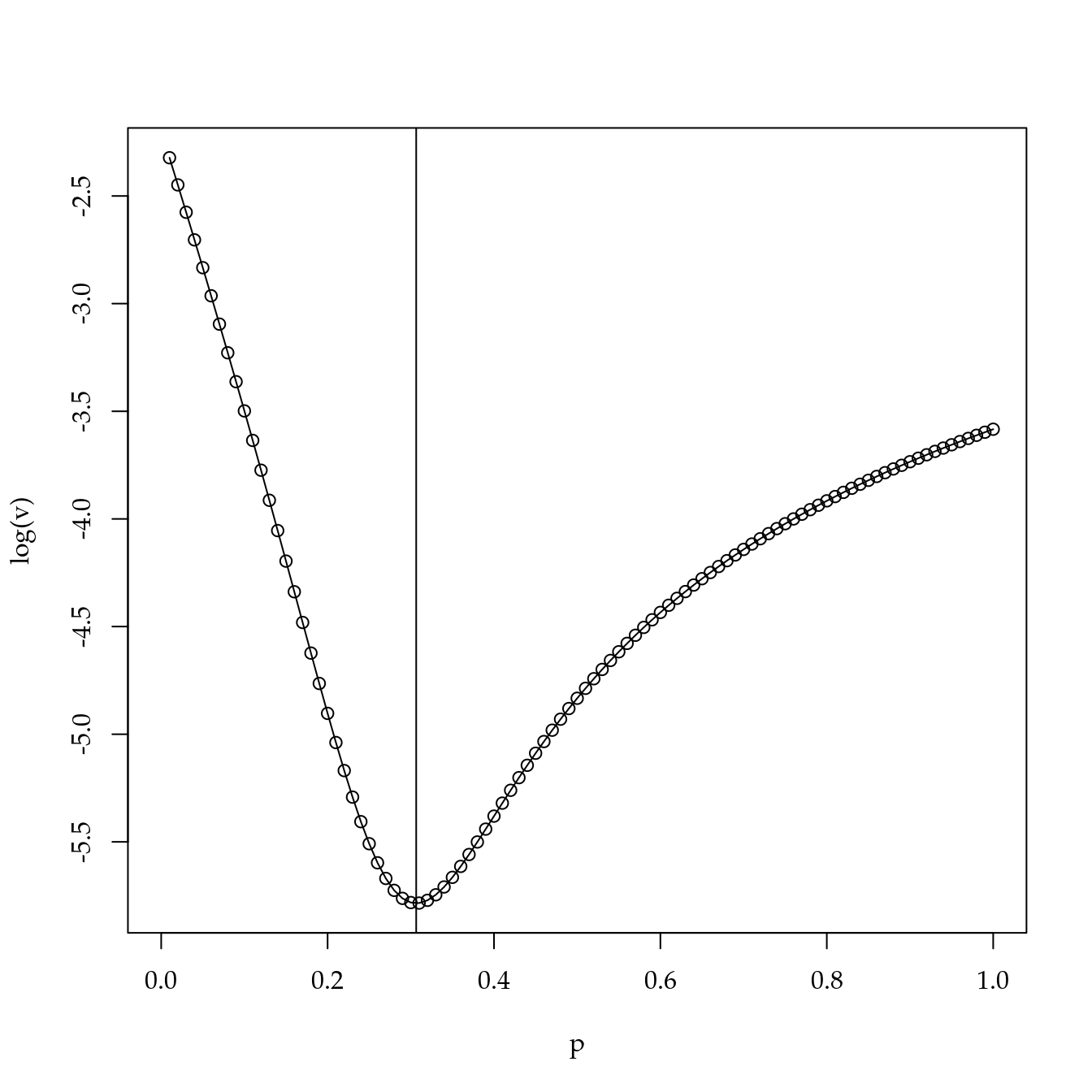
da$dosep <- da$dose^0.3O gráfico de segmentos a seguir exibe os intervalos de tempo em que as mudas morreram no experimento. A morte das mudas, até onde se sabe, pode não ter relação com os fatores estudados pois depende muito de fatores agronômicos que representam o estado inicial da muda. No entanto, embora o emperimento não tenha sido realizado para estudar fatores relacionados ao “pegamento” das mudas, pode-se verificar se existe influência dos fatores estudados na sobreviênvia das mudas.
#-----------------------------------------------------------------------
# Determinar o intervalo em que a muda morreu.
# Cria a variável unidade experimental.
da$ue <- with(da, interaction(gen, dos, drop = TRUE))
# Número de níveis dos fatores.
with(da, c(gen = nlevels(gen),
dos = nlevels(dos),
ue = nlevels(ue)))## gen dos ue
## 29 10 290summary(da)## gen dose data
## 1 : 200 Min. :0.0000 Min. :2016-06-15
## 2 : 200 1st Qu.:0.0368 1st Qu.:2016-07-18
## 3 : 200 Median :0.2208 Median :2016-08-20
## 4 : 200 Mean :0.9402 Mean :2016-08-20
## 5 : 200 3rd Qu.:1.1776 3rd Qu.:2016-09-22
## 6 : 200 Max. :4.7104 Max. :2016-10-26
## (Other):4600
## dac alt mspa msr
## Min. : 23.30 Min. : 17.0 Min. : 2.96 Min. : 3.07
## 1st Qu.: 52.00 1st Qu.:106.0 1st Qu.:19.36 1st Qu.: 27.81
## Median : 62.00 Median :130.0 Median :36.83 Median : 40.01
## Mean : 67.92 Mean :141.5 Mean :35.76 Mean : 43.79
## 3rd Qu.: 79.03 3rd Qu.:157.0 3rd Qu.:48.67 3rd Qu.: 59.72
## Max. :149.70 Max. :528.0 Max. :93.43 Max. :133.07
## NA's :1728 NA's :1730 NA's :5643 NA's :5643
## dias dos dosep ue
## Min. : 2.00 0 : 580 Min. :0.0000 1.0 : 20
## 1st Qu.: 35.25 1 : 580 1st Qu.:0.3713 2.0 : 20
## Median : 68.50 2 : 580 Median :0.6279 3.0 : 20
## Mean : 68.50 3 : 580 Mean :0.7174 4.0 : 20
## 3rd Qu.:101.75 4 : 580 3rd Qu.:1.0503 5.0 : 20
## Max. :135.00 5 : 580 Max. :1.5919 6.0 : 20
## (Other):2320 (Other):5680# Obtém os intervalos em que as mudas morrem.
db <- ddply(da,
~gen + dose + dos + dosep + ue,
deathint,
timevar = "dias",
respvar = "alt")
# Áreas abaixo da curva (usando semanas e cm como unidades).
dc <- ddply(da,
~gen + dose + dos + dosep + ue,
summarise,
aacalt = auc(dias/7, alt/10),
aacdac = auc(dias/7, dac/10))
# Junta os dois.
db <- merge(db, dc)
# str(db)
# Gráfico de segmentos que indica o intervalo em que a muda morreu.
segplot(dos ~ left + right | gen,
data = db,
as.table = TRUE,
xlab = "Tempo após a primeira avaliação",
ylab = "Dose (codificada)") # +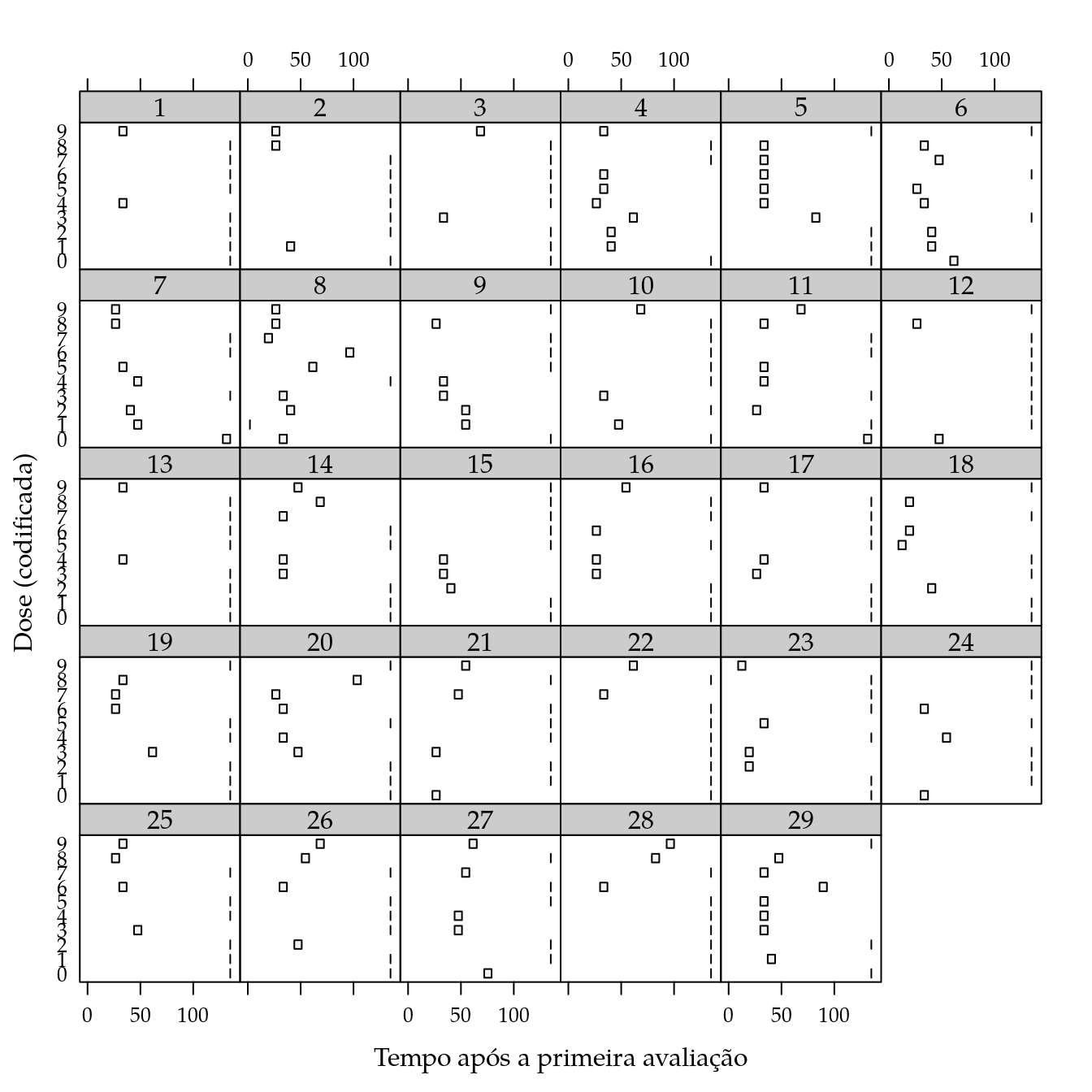
# layer(panel.text(x = x[subscripts],
# y = z[subscripts],
# labels = db$status[subscripts],
# pos = 2,
# cex = 0.6))Massa seca de parte áerea
A análise dos dados de massa seca de parte aérea foi feita apenas considerando genótipos com 3 ou mais registros, que correspondem ao número de doses de cálcio que restaram ao final do experimento.
dc <- subset(da, data == max(data))
dc <- dc[complete.cases(dc), ]
# Tabela de frequência do número de observações por cela experimental.
addmargins(xtabs(~gen + dos, data = dc))## dos
## gen 0 1 2 3 4 5 6 7 8 9 Sum
## 1 1 1 1 0 0 1 1 1 1 0 7
## 2 1 0 1 1 1 1 1 1 0 0 7
## 3 1 1 1 0 1 1 0 1 1 0 7
## 4 1 0 0 0 0 0 0 1 1 0 3
## 5 0 1 1 0 0 0 0 0 0 0 2
## 6 0 0 0 1 0 0 1 0 0 1 3
## 7 0 0 0 1 0 0 1 1 0 0 3
## 8 0 0 0 0 1 0 0 0 0 0 1
## 9 1 0 0 0 0 1 0 1 0 0 3
## 10 1 0 1 0 1 1 1 1 0 0 6
## 11 0 1 0 1 0 0 1 1 0 0 4
## 12 0 1 1 1 1 1 1 1 0 1 8
## 13 1 1 1 1 0 1 0 1 1 0 7
## 14 1 1 1 0 0 1 1 0 0 0 5
## 15 1 0 0 0 0 1 1 1 1 0 5
## 16 1 1 1 0 0 1 0 1 1 0 6
## 17 1 1 1 0 0 1 1 0 1 0 6
## 18 1 1 0 1 1 0 0 1 0 1 6
## 19 1 1 1 0 1 1 0 0 0 1 6
## 20 1 1 1 0 0 1 0 0 0 1 5
## 21 0 1 1 0 1 1 1 0 1 0 6
## 22 1 1 1 1 1 1 1 0 1 0 8
## 23 1 1 0 0 1 0 1 1 1 0 6
## 24 0 1 1 1 0 1 0 1 0 1 6
## 25 1 1 1 0 1 1 0 1 0 0 6
## 26 1 1 0 1 1 1 0 1 0 0 6
## 27 0 1 1 0 0 1 0 0 1 0 4
## 28 1 1 1 1 1 1 0 1 0 0 7
## 29 1 0 1 0 0 0 0 0 0 1 3
## Sum 20 20 19 11 13 20 13 18 11 7 152# Manter apenas genótipos com 3 ou mais registros.
k <- which(xtabs(~gen, data = dc) >= 3)
# Filtra para os genótipos com 3 ou mais registros.
dc <- droplevels(subset(dc, gen %in% names(k)))
xy1 <- xyplot(mspa + msr ~ dosep | gen,
outer = FALSE,
data = dc,
ylab = "Massa seca (g)",
xlab = "Dose de Ca (escala quase equidistante)",
auto.key = TRUE,
type = "o")
xy2 <- xyplot(mspa + msr ~ dosep | gen,
outer = FALSE,
data = dc,
ylab = "Massa seca (g)",
xlab = "Dose de Ca (escala quase equidistante)",
auto.key = TRUE,
type = c("p", "r"))
# grid.arrange(xy1, xy2, nrow = 1)
xy2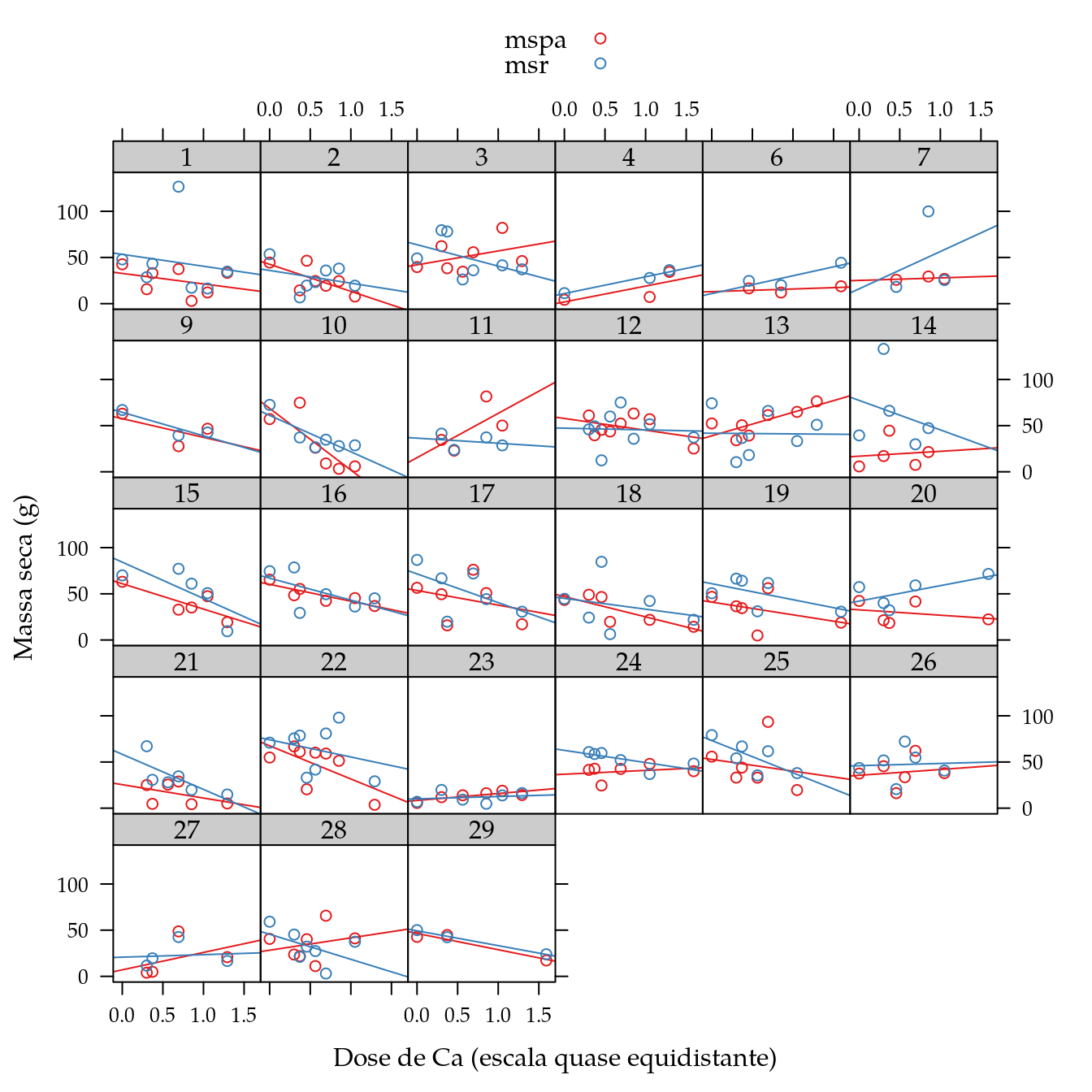
Foi considerado um modelo fatorial duplo contendo os efeitos do fator genótipo (níveis categóricos), do fator dose de cálcio (níveis métricos, expresso na escala transformada) e a interação entre estes fatores. Para representar o efeito de cálcio, foram utilizados polinômio de segundo grau, sendo o grau do polinômio aumentado ou diminuido conforme necessidade de melhorar ou simplificar o ajuste.
# Modelo de efeitos quadráticos por genótipo.
m0 <- lm(mspa ~ gen * (dosep + I(dosep^2)), data = dc)
# Verificação dos pressupostos.
par(mfrow = c(2, 2))
plot(m0); layout(1)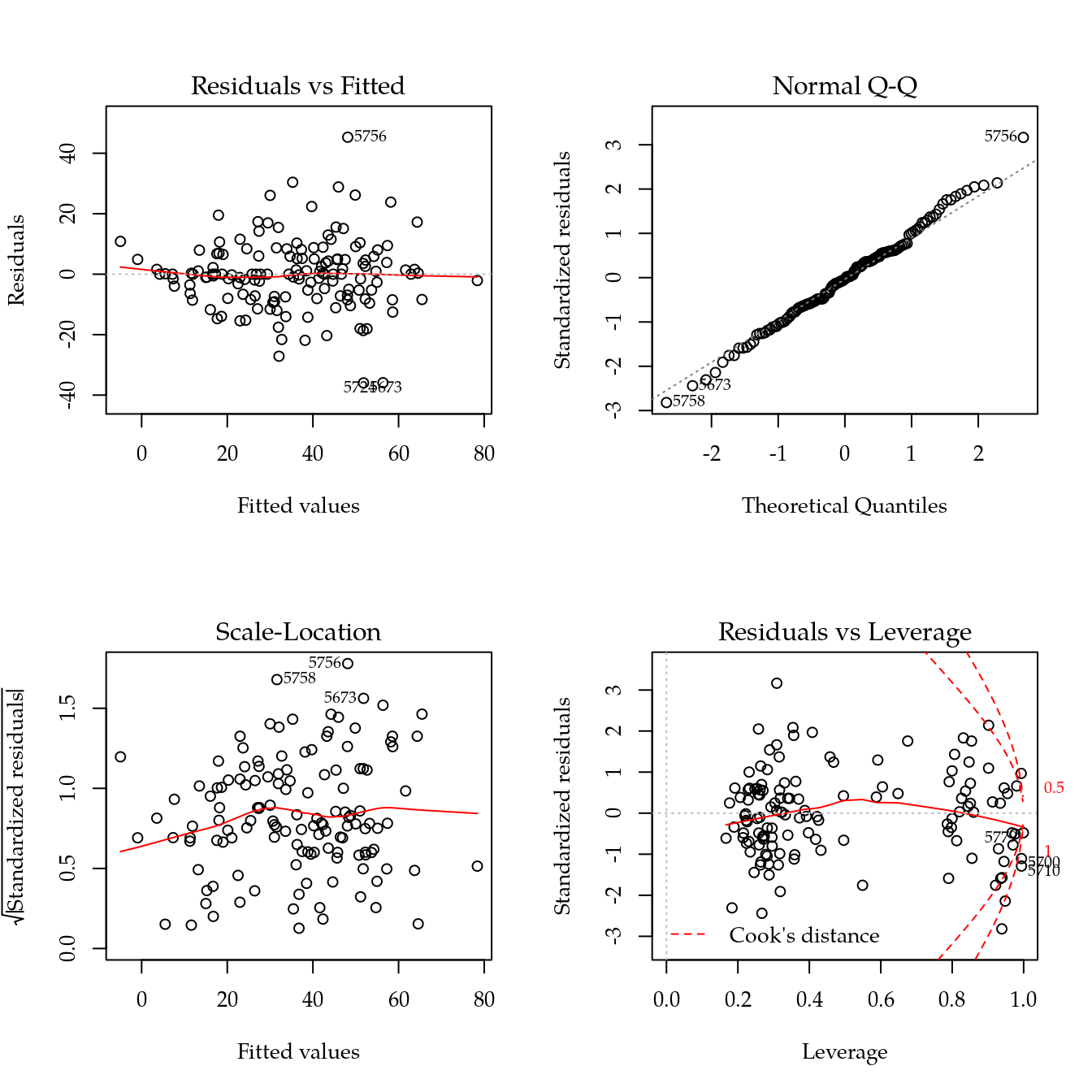
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: mspa
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 22105.4 850.21 2.8684 0.000272 ***
## dosep 1 1477.5 1477.53 4.9848 0.028866 *
## I(dosep^2) 1 27.4 27.37 0.0923 0.762168
## gen:dosep 26 10089.5 388.06 1.3092 0.187858
## gen:I(dosep^2) 26 4301.5 165.44 0.5582 0.950194
## Residuals 68 20155.5 296.41
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Reduzindo o modelo com a remoção dos termos quadráticos.
m0 <- update(m0, . ~ gen * dosep)
# par(mfrow = c(2, 2))
# plot(m0); layout(1)
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: mspa
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 22105.4 850.21 3.2839 1.278e-05 ***
## dosep 1 1477.5 1477.53 5.7069 0.01887 *
## gen:dosep 26 9978.3 383.78 1.4823 0.08749 .
## Residuals 95 24595.6 258.90
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## Analysis of Variance Table
##
## Response: mspa
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 22105 850.21 2.9755 3.03e-05 ***
## dosep 1 1478 1477.53 5.1710 0.02473 *
## Residuals 121 34574 285.73
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(m0)##
## Call:
## lm(formula = mspa ~ gen + dosep, data = dc)
##
## Residuals:
## Min 1Q Median 3Q Max
## -37.957 -12.348 0.087 9.066 48.438
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 30.26998 6.75309 4.482 1.69e-05 ***
## gen2 -0.03656 9.03960 -0.004 0.99678
## gen3 25.61210 9.03647 2.834 0.00538 **
## gen4 -9.04181 11.67273 -0.775 0.44008
## gen6 -7.09007 11.71266 -0.605 0.54609
## gen7 3.04863 11.67346 0.261 0.79441
## gen9 19.94146 11.66706 1.709 0.08998 .
## gen10 3.59749 9.40674 0.382 0.70281
## gen11 22.09697 10.59505 2.086 0.03912 *
## gen12 23.75822 8.75296 2.714 0.00761 **
## gen13 28.39832 9.03739 3.142 0.00211 **
## gen14 -7.59291 9.92236 -0.765 0.44562
## gen15 15.22280 9.90683 1.537 0.12700
## gen16 23.38485 9.40502 2.486 0.01427 *
## gen17 18.50569 9.40698 1.967 0.05145 .
## gen18 7.21872 9.40439 0.768 0.44423
## gen19 7.13165 9.40687 0.758 0.44985
## gen20 3.45686 9.89985 0.349 0.72756
## gen21 -9.50005 9.40479 -1.010 0.31445
## gen22 21.23611 8.75317 2.426 0.01674 *
## gen23 -11.64449 9.40472 -1.238 0.21806
## gen24 15.27988 9.40946 1.624 0.10700
## gen25 20.01100 9.41877 2.125 0.03566 *
## gen26 12.43185 9.41623 1.320 0.18924
## gen27 -5.59920 10.59503 -0.528 0.59814
## gen28 8.36645 9.05152 0.924 0.35716
## gen29 9.62461 11.66465 0.825 0.41093
## dosep -7.63221 3.35632 -2.274 0.02473 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 16.9 on 121 degrees of freedom
## Multiple R-squared: 0.4055, Adjusted R-squared: 0.2729
## F-statistic: 3.057 on 27 and 121 DF, p-value: 1.542e-05Não houve interação entre genótipo e dose de cálcio. Em função disso, o modelo obtido para a predição contém apenas os efeitos aditivos dos fatores. Foi necessário apenas o polinômio de grau um para representar o efeito de cálcio. Os resultados serão representados com as retas ajustadas para cada genótipo.
# Cria um conjunto de valores para predição.
pred <- with(dc,
expand.grid(# dosep = eseq(dose, f = 0)^0.3)
dosep = eseq(dosep, f = 0),
gen = levels(gen)))
ic <- predict(m0, newdata = pred, interval = "confidence")
pred <- cbind(pred, as.data.frame(ic))
xyplot(mspa ~ dosep | gen,
data = dc,
# type = c("p", "r"),
xlab = "Dose de cálcio (escala transformada)",
ylab = "Massa seca de parte áerea (g)") +
as.layer(xyplot(fit ~ dosep | gen,
data = pred,
type = "l",
ly = pred$lwr,
uy = pred$upr,
cty = "bands",
prepanel = prepanel.cbH,
panel = panel.cbH))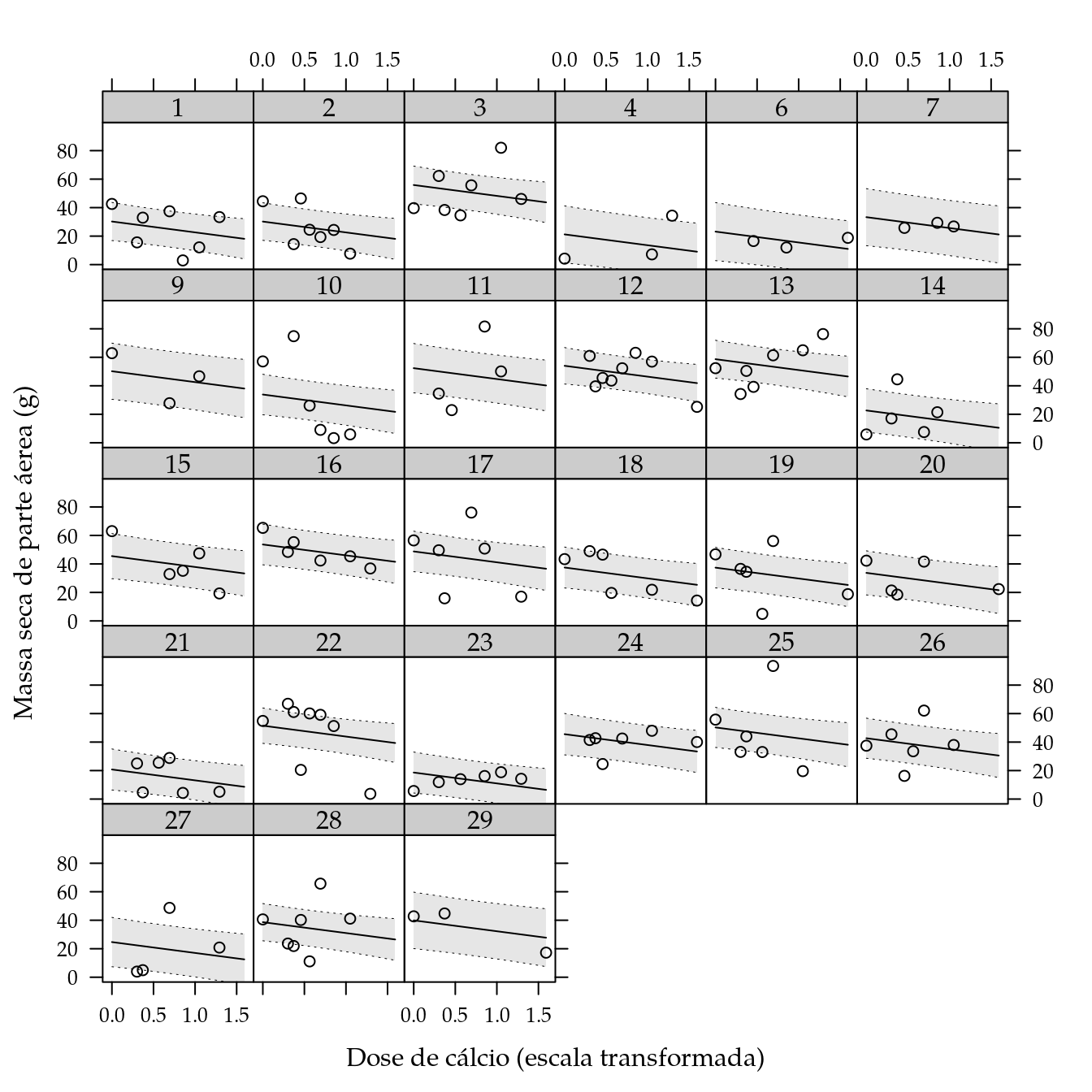
# Médias ajustadas, considerando um valor fixo de cálcio.
lsm <- LSmeans(m0, effect = "gen")
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 25.4849 6.3895 121.0000 3.9885 0.0001
## [2,] 25.4483 6.3919 121.0000 3.9814 0.0001
## [3,] 51.0970 6.3892 121.0000 7.9974 0.0000
## [4,] 16.4431 9.7730 121.0000 1.6825 0.0951
## [5,] 18.3948 9.8260 121.0000 1.8721 0.0636
## [6,] 28.5335 9.7741 121.0000 2.9193 0.0042
## [7,] 45.4264 9.7606 121.0000 4.6541 0.0000
## [8,] 29.0824 6.9021 121.0000 4.2135 0.0000
## [9,] 47.5819 8.4528 121.0000 5.6291 0.0000
## [10,] 49.2431 5.9874 121.0000 8.2245 0.0000
## [11,] 53.8832 6.3899 121.0000 8.4326 0.0000
## [12,] 17.8920 7.5845 121.0000 2.3590 0.0199
## [13,] 40.7077 7.5765 121.0000 5.3729 0.0000
## [14,] 48.8698 6.9010 121.0000 7.0816 0.0000
## [15,] 43.9906 6.9023 121.0000 6.3733 0.0000
## [16,] 32.7036 6.9018 121.0000 4.7384 0.0000
## [17,] 32.6166 6.9022 121.0000 4.7255 0.0000
## [18,] 28.9418 7.5605 121.0000 3.8280 0.0002
## [19,] 15.9849 6.9031 121.0000 2.3156 0.0223
## [20,] 46.7210 5.9798 121.0000 7.8131 0.0000
## [21,] 13.8404 6.9029 121.0000 2.0050 0.0472
## [22,] 40.7648 6.9121 121.0000 5.8976 0.0000
## [23,] 45.4959 6.9148 121.0000 6.5795 0.0000
## [24,] 37.9167 6.9119 121.0000 5.4857 0.0000
## [25,] 19.8857 8.4528 121.0000 2.3526 0.0203
## [26,] 33.8513 6.4053 121.0000 5.2849 0.0000
## [27,] 35.1095 9.7598 121.0000 3.5974 0.0005# Dose usada para obter as médias (é a média de cálcio dos dados
# observados).
d <- lsm$grid$dosep[1]^(1/0.3)
d## [1] 0.210926# Atribui nomes às linhas da matriz.
L <- lsm$L
rownames(L) <- levels(dc$gen)
# Obtém os contrastes dois a dois.
grid <- apmc(X = L,
model = m0,
focus = "gen",
test = "fdr",
cld2 = FALSE)
grid <- grid[order(grid$fit, decreasing = TRUE), ]
ocld <- ordered_cld(grid$cld, grid$fit)
grid$cld <- ocld
# x <- attr(ocld, "ind") * 1
# print.table(local({x[x == 0] <- NA; x}))
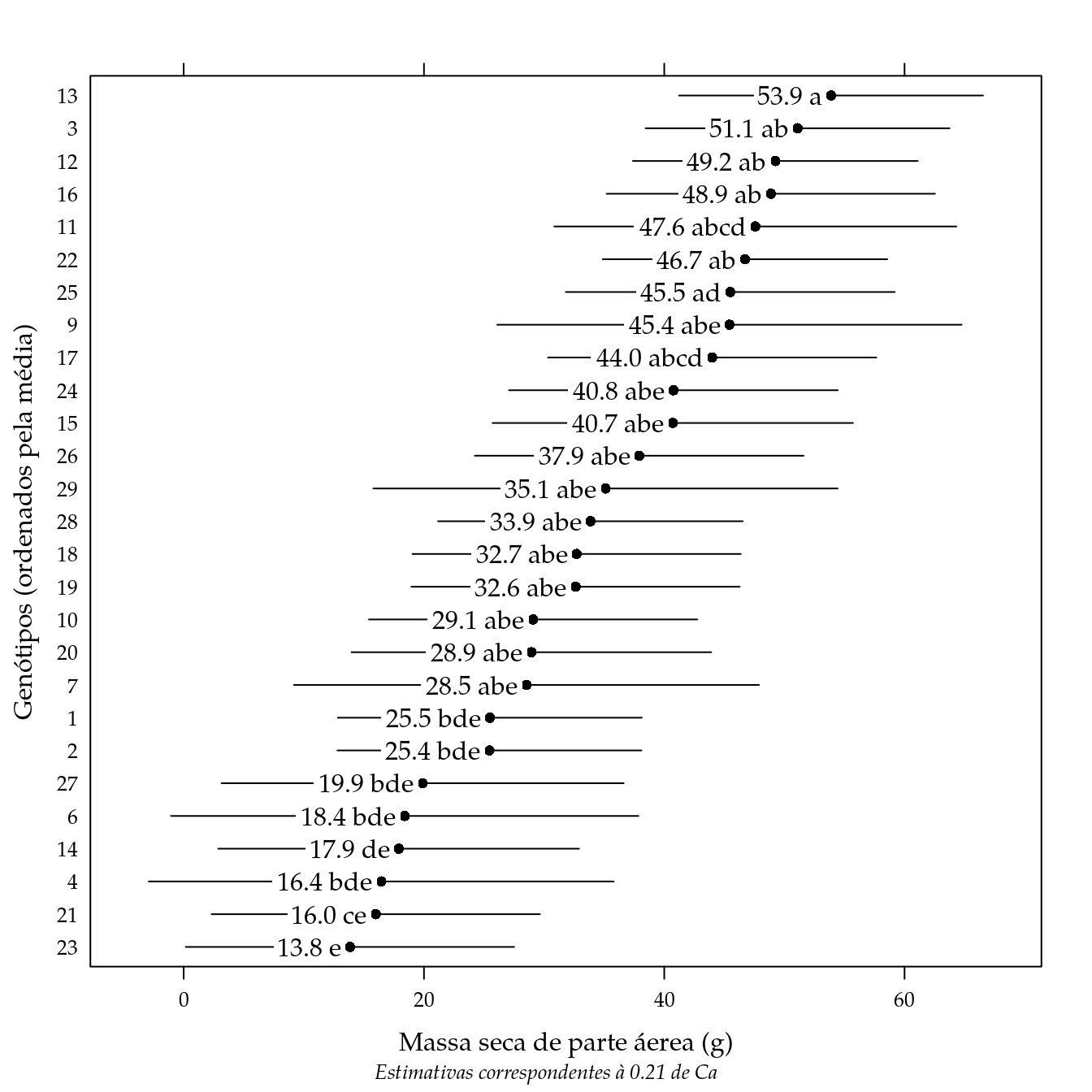
# Gráfico de segmentos com as letras resumo das comparações múltiplas.
segplot(reorder(gen, fit) ~ lwr + upr,
centers = fit,
data = grid,
draw = FALSE,
xlab = "Massa seca de parte áerea (g)",
ylab = "Genótipos (ordenados pela média)",
# par.settings = list(layout.widths = list(right.padding = 10)),
sub = list(
sprintf("Estimativas correspondentes à %0.2f de Ca", d),
font = 3, cex = 0.8),
txt = sprintf("%0.1f %s", grid$fit, grid$cld)) +
layer({
x <- unit(centers, "native") - unit(0.005, "snpc")
y <- unit(z, "native")
grid.rect(x = x, y = y,
width = Reduce(unit.c,
lapply(txt,
FUN = unit,
x = 1.05,
units = "strwidth")) +
unit(0.005, "snpc"),
height = unit(1, "lines"),
just = "right",
gp = gpar(fill = "white",
col = NA,
fontsize = 12))
grid.text(label = txt,
just = "right",
x = x - unit(0.005, "snpc"),
y = y)
})
# x <- attr(ocld, "ind")
# index <- which(x[nrow(x):1, ], arr.ind = TRUE)
# trellis.focus("panel", column = 1, row = 1, clip.off = TRUE)
# xcor <- 1.03 + (index[, 2] - 1)/50
# grid.segments(x0 = unit(xcor, "npc"),
# x1 = unit(xcor, "npc"),
# y0 = unit(index[, 1] + 0.5, units = "native"),
# y1 = unit(index[, 1] - 0.5, units = "native"),
# gp = gpar(lwd = 2, col = "red"))
# trellis.unfocus()Massa seca de raízes
# Modelo de efeitos quadráticos por genótipo.
m0 <- lm(msr ~ gen * (dosep + I(dosep^2)), data = dc)
# Verificação dos pressupostos.
par(mfrow = c(2, 2))
plot(m0); layout(1)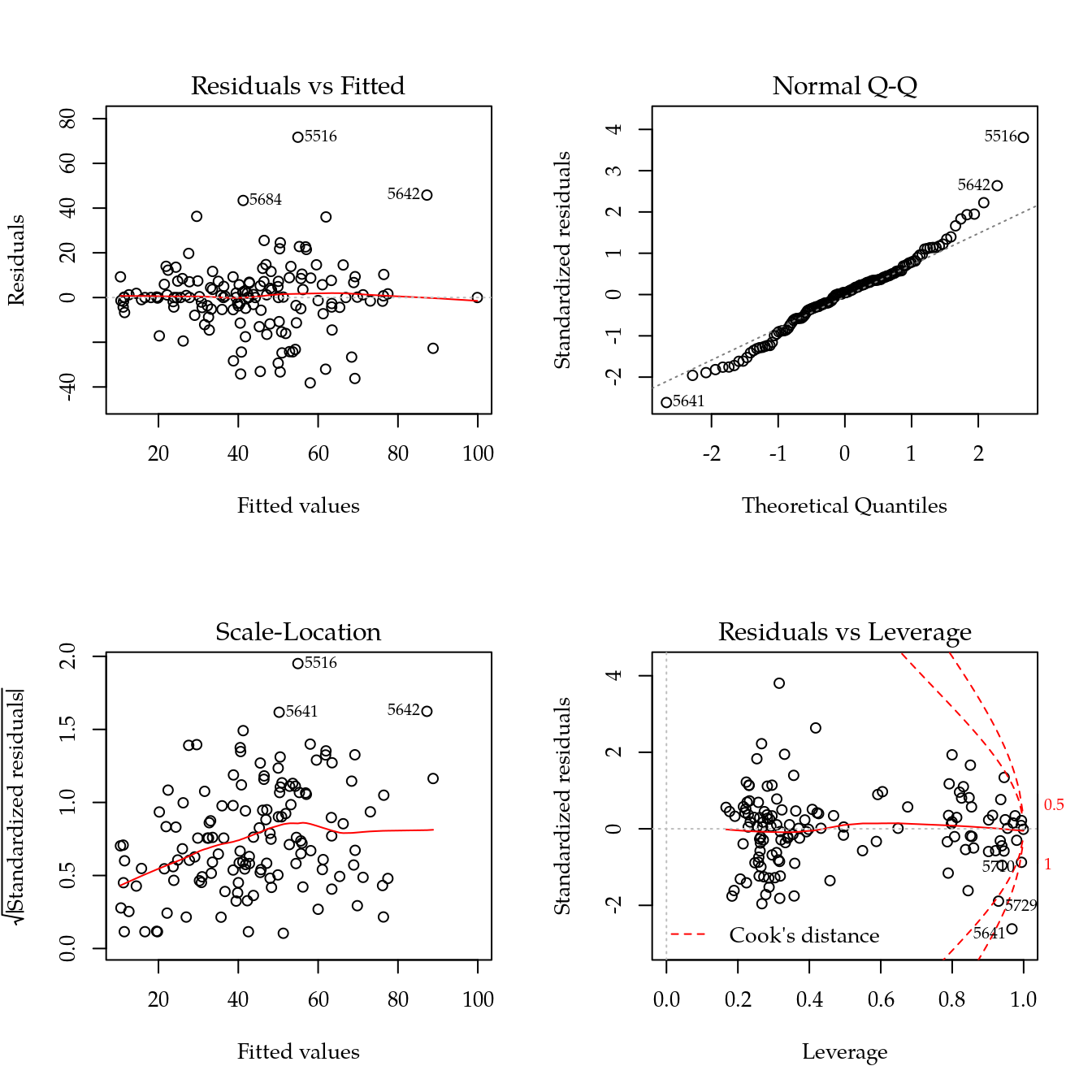
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: msr
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 23001 884.6 1.7046 0.041564 *
## dosep 1 3884 3884.1 7.4840 0.007933 **
## I(dosep^2) 1 377 376.8 0.7260 0.397171
## gen:dosep 26 6946 267.2 0.5148 0.969154
## gen:I(dosep^2) 26 12463 479.3 0.9236 0.576167
## Residuals 68 35291 519.0
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Reduzindo o modelo com a remoção dos termos quadráticos.
m0 <- update(m0, . ~ gen * dosep)
# par(mfrow = c(2, 2))
# plot(m0); layout(1)
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: msr
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 23001 884.6 1.7598 0.02569 *
## dosep 1 3884 3884.1 7.7265 0.00656 **
## gen:dosep 26 7320 281.5 0.5601 0.95354
## Residuals 95 47757 502.7
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## Analysis of Variance Table
##
## Response: msr
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 23001 884.6 1.9435 0.008623 **
## dosep 1 3884 3884.1 8.5331 0.004162 **
## Residuals 121 55077 455.2
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(m0)##
## Call:
## lm(formula = msr ~ gen + dosep, data = dc)
##
## Residuals:
## Min 1Q Median 3Q Max
## -37.807 -11.047 -1.707 9.904 82.182
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 53.0222 8.5234 6.221 7.34e-09 ***
## gen2 -17.9073 11.4093 -1.570 0.11914
## gen3 4.1755 11.4054 0.366 0.71493
## gen4 -18.2065 14.7327 -1.236 0.21893
## gen6 -11.4613 14.7831 -0.775 0.43968
## gen7 4.5578 14.7336 0.309 0.75759
## gen9 3.8148 14.7256 0.259 0.79603
## gen10 -7.8243 11.8727 -0.659 0.51114
## gen11 -12.0316 13.3725 -0.900 0.37005
## gen12 1.9572 11.0475 0.177 0.85967
## gen13 -4.2971 11.4065 -0.377 0.70704
## gen14 15.6134 12.5235 1.247 0.21490
## gen15 10.2654 12.5039 0.821 0.41327
## gen16 6.8593 11.8705 0.578 0.56444
## gen17 7.5859 11.8730 0.639 0.52408
## gen18 -7.5323 11.8697 -0.635 0.52690
## gen19 5.0121 11.8729 0.422 0.67367
## gen20 6.3360 12.4951 0.507 0.61302
## gen21 -12.1416 11.8702 -1.023 0.30841
## gen22 17.4641 11.0478 1.581 0.11654
## gen23 -32.8454 11.8702 -2.767 0.00655 **
## gen24 8.9385 11.8761 0.753 0.45312
## gen25 8.9716 11.8879 0.755 0.45190
## gen26 0.5503 11.8847 0.046 0.96314
## gen27 -22.2116 13.3725 -1.661 0.09930 .
## gen28 -14.7165 11.4244 -1.288 0.20015
## gen29 -6.1375 14.7225 -0.417 0.67751
## dosep -12.3745 4.2362 -2.921 0.00416 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 21.33 on 121 degrees of freedom
## Multiple R-squared: 0.328, Adjusted R-squared: 0.1781
## F-statistic: 2.188 on 27 and 121 DF, p-value: 0.002119Não houve interação entre genótipo e dose de cálcio. Em função disso, o modelo obtido para a predição contém apenas os efeitos aditivos dos fatores. Foi necessário apenas o polinômio de grau um para representar o efeito de cálcio. Os resultados serão representados com as retas ajustadas para cada genótipo.
# Cria um conjunto de valores para predição.
pred <- with(dc,
expand.grid(# dosep = eseq(dose, f = 0)^0.3)
dosep = eseq(dosep, f = 0),
gen = levels(gen)))
ic <- predict(m0, newdata = pred, interval = "confidence")
pred <- cbind(pred, as.data.frame(ic))
xyplot(mspa ~ dosep | gen,
data = dc,
# type = c("p", "r"),
xlab = "Dose de cálcio (escala transformada)",
ylab = "Massa seca de raízes (g)") +
as.layer(xyplot(fit ~ dosep | gen,
data = pred,
type = "l",
ly = pred$lwr,
uy = pred$upr,
cty = "bands",
prepanel = prepanel.cbH,
panel = panel.cbH))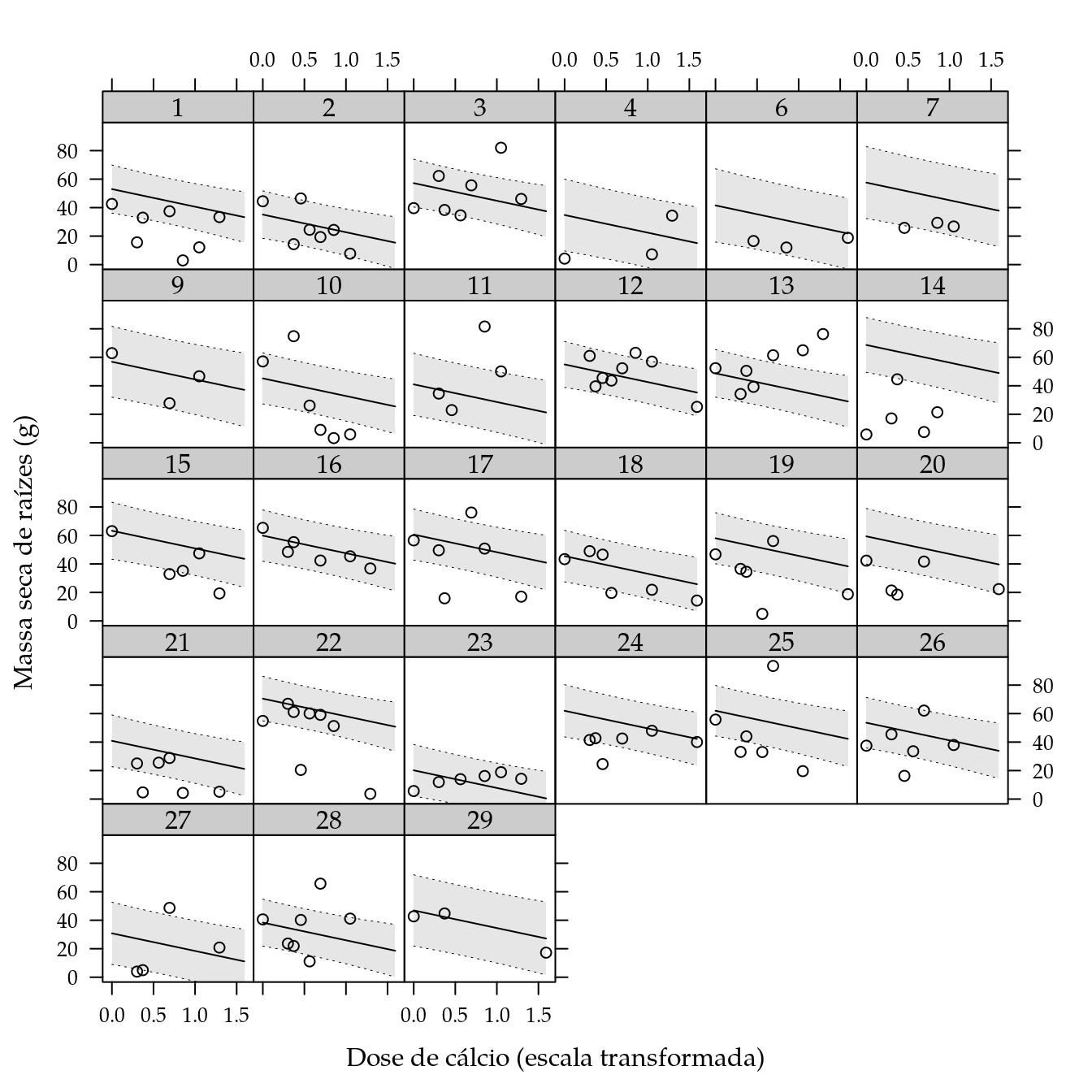
# Médias ajustadas, considerando um valor fixo de cálcio.
lsm <- LSmeans(m0, effect = "gen")
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 45.2639 8.0645 121.0000 5.6127 0.0000
## [2,] 27.3566 8.0675 121.0000 3.3910 0.0009
## [3,] 49.4393 8.0642 121.0000 6.1307 0.0000
## [4,] 27.0574 12.3350 121.0000 2.1935 0.0302
## [5,] 33.8026 12.4019 121.0000 2.7256 0.0074
## [6,] 49.8217 12.3364 121.0000 4.0386 0.0001
## [7,] 49.0787 12.3193 121.0000 3.9839 0.0001
## [8,] 37.4395 8.7115 121.0000 4.2977 0.0000
## [9,] 33.2323 10.6687 121.0000 3.1149 0.0023
## [10,] 47.2211 7.5570 121.0000 6.2487 0.0000
## [11,] 40.9668 8.0650 121.0000 5.0796 0.0000
## [12,] 60.8773 9.5728 121.0000 6.3594 0.0000
## [13,] 55.5293 9.5627 121.0000 5.8069 0.0000
## [14,] 52.1232 8.7100 121.0000 5.9843 0.0000
## [15,] 52.8498 8.7118 121.0000 6.0665 0.0000
## [16,] 37.7316 8.7111 121.0000 4.3314 0.0000
## [17,] 50.2759 8.7116 121.0000 5.7711 0.0000
## [18,] 51.5999 9.5425 121.0000 5.4074 0.0000
## [19,] 33.1223 8.7128 121.0000 3.8016 0.0002
## [20,] 62.7280 7.5474 121.0000 8.3112 0.0000
## [21,] 12.4184 8.7125 121.0000 1.4254 0.1566
## [22,] 54.2024 8.7241 121.0000 6.2129 0.0000
## [23,] 54.2355 8.7275 121.0000 6.2143 0.0000
## [24,] 45.8142 8.7239 121.0000 5.2516 0.0000
## [25,] 23.0523 10.6687 121.0000 2.1607 0.0327
## [26,] 30.5474 8.0844 121.0000 3.7785 0.0002
## [27,] 39.1264 12.3183 121.0000 3.1763 0.0019# Dose usada para obter as médias (é a média de cálcio dos dados
# observados).
d <- lsm$grid$dosep[1]^(1/0.3)
d## [1] 0.210926# Atribui nomes às linhas da matriz.
L <- lsm$L
rownames(L) <- levels(dc$gen)
# Obtém os contrastes dois a dois.
grid <- apmc(X = L,
model = m0,
focus = "gen",
test = "fdr",
cld2 = FALSE)
grid <- grid[order(grid$fit, decreasing = TRUE), ]
ocld <- ordered_cld(grid$cld, grid$fit)
grid$cld <- ocld
# Gráfico de segmentos com as letras resumo das comparações múltiplas.
segplot(reorder(gen, fit) ~ lwr + upr,
centers = fit,
data = grid,
draw = FALSE,
xlab = "Massa seca de raízes (g)",
ylab = "Genótipos (ordenados pela média)",
sub = list(
sprintf("Estimativas correspondentes à %0.2f de Ca", d),
font = 3, cex = 0.8),
txt = sprintf("%0.1f %s", grid$fit, grid$cld)) +
layer({
x <- unit(centers, "native") - unit(0.005, "snpc")
y <- unit(z, "native")
grid.rect(x = x, y = y,
width = Reduce(unit.c,
lapply(txt,
FUN = unit,
x = 1.05,
units = "strwidth")) +
unit(0.005, "snpc"),
height = unit(1, "lines"),
just = "right",
gp = gpar(fill = "white",
col = NA,
fontsize = 12))
grid.text(label = txt,
just = "right",
x = x - unit(0.005, "snpc"),
y = y)
})
Proporção de massa em raízes
# Cria variável que a porcentagem de massa seca de raízes em relação a
# massa seca total da muda.
dc$r <- with(dc, 100 * msr/(msr + mspa))# Modelo de efeitos quadráticos por genótipo.
m0 <- lm(r ~ gen * (dosep + I(dosep^2)), data = dc)
# Verificação dos pressupostos.
par(mfrow = c(2, 2))
plot(m0); layout(1)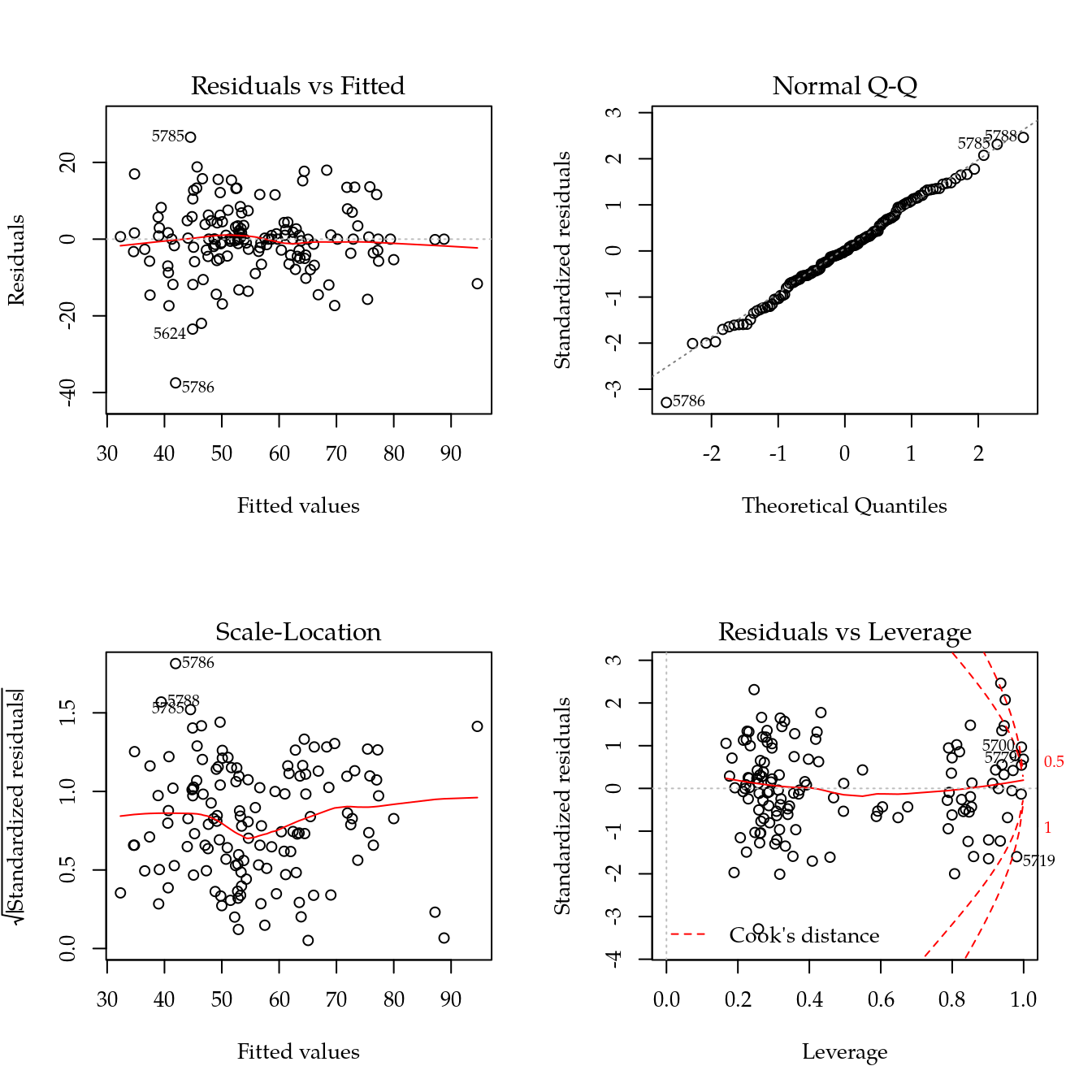
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: r
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 10846.5 417.17 2.3854 0.00225 **
## dosep 1 7.2 7.18 0.0410 0.84007
## I(dosep^2) 1 325.8 325.76 1.8627 0.17682
## gen:dosep 26 5523.2 212.43 1.2147 0.25788
## gen:I(dosep^2) 26 4777.1 183.73 1.0506 0.42085
## Residuals 68 11892.4 174.89
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1## Analysis of Variance Table
##
## Response: r
## Df Sum Sq Mean Sq F value Pr(>F)
## gen 26 10846 417.17 2.2594 0.001592 **
## Residuals 122 22526 184.64
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(m0)##
## Call:
## lm(formula = r ~ gen, data = dc)
##
## Residuals:
## Min 1Q Median 3Q Max
## -44.368 -6.919 0.295 9.255 28.680
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 63.62133 5.13582 12.388 < 2e-16 ***
## gen2 -11.83644 7.26315 -1.630 0.10575
## gen3 -15.17842 7.26315 -2.090 0.03871 *
## gen4 4.27467 9.37668 0.456 0.64928
## gen6 0.48032 9.37668 0.051 0.95923
## gen7 -7.83977 9.37668 -0.836 0.40474
## gen9 -10.97340 9.37668 -1.170 0.24417
## gen10 1.58445 7.55972 0.210 0.83434
## gen11 -20.34053 8.51679 -2.388 0.01846 *
## gen12 -16.13130 7.03251 -2.294 0.02351 *
## gen13 -23.41823 7.26315 -3.224 0.00162 **
## gen14 13.19129 7.95638 1.658 0.09990 .
## gen15 -9.46325 7.95638 -1.189 0.23660
## gen16 -13.08349 7.55972 -1.731 0.08604 .
## gen17 -8.13620 7.55972 -1.076 0.28394
## gen18 -13.76289 7.55972 -1.821 0.07113 .
## gen19 0.08582 7.55972 0.011 0.99096
## gen20 0.62266 7.95638 0.078 0.93775
## gen21 6.83064 7.55972 0.904 0.36801
## gen22 -3.54485 7.03251 -0.504 0.61512
## gen23 -17.66014 7.55972 -2.336 0.02112 *
## gen24 -6.71211 7.55972 -0.888 0.37635
## gen25 -7.21256 7.55972 -0.954 0.34193
## gen26 -8.66591 7.55972 -1.146 0.25390
## gen27 -2.26186 8.51679 -0.266 0.79101
## gen28 -14.78767 7.26315 -2.036 0.04392 *
## gen29 -10.05089 9.37668 -1.072 0.28588
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 13.59 on 122 degrees of freedom
## Multiple R-squared: 0.325, Adjusted R-squared: 0.1812
## F-statistic: 2.259 on 26 and 122 DF, p-value: 0.001592# Médias ajustadas, considerando um valor fixo de cálcio.
lsm <- LSmeans(m0, effect = "gen")
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 63.6213 5.1358 122.0000 12.3878 0
## [2,] 51.7849 5.1358 122.0000 10.0831 0
## [3,] 48.4429 5.1358 122.0000 9.4324 0
## [4,] 67.8960 7.8451 122.0000 8.6546 0
## [5,] 64.1016 7.8451 122.0000 8.1709 0
## [6,] 55.7816 7.8451 122.0000 7.1104 0
## [7,] 52.6479 7.8451 122.0000 6.7109 0
## [8,] 65.2058 5.5473 122.0000 11.7545 0
## [9,] 43.2808 6.7941 122.0000 6.3704 0
## [10,] 47.4900 4.8041 122.0000 9.8853 0
## [11,] 40.2031 5.1358 122.0000 7.8280 0
## [12,] 76.8126 6.0768 122.0000 12.6403 0
## [13,] 54.1581 6.0768 122.0000 8.9123 0
## [14,] 50.5378 5.5473 122.0000 9.1103 0
## [15,] 55.4851 5.5473 122.0000 10.0021 0
## [16,] 49.8584 5.5473 122.0000 8.9878 0
## [17,] 63.7071 5.5473 122.0000 11.4843 0
## [18,] 64.2440 6.0768 122.0000 10.5720 0
## [19,] 70.4520 5.5473 122.0000 12.7002 0
## [20,] 60.0765 4.8041 122.0000 12.5052 0
## [21,] 45.9612 5.5473 122.0000 8.2853 0
## [22,] 56.9092 5.5473 122.0000 10.2589 0
## [23,] 56.4088 5.5473 122.0000 10.1687 0
## [24,] 54.9554 5.5473 122.0000 9.9067 0
## [25,] 61.3595 6.7941 122.0000 9.0314 0
## [26,] 48.8337 5.1358 122.0000 9.5084 0
## [27,] 53.5704 7.8451 122.0000 6.8285 0# Atribui nomes às linhas da matriz.
L <- lsm$L
rownames(L) <- levels(dc$gen)
# Obtém os contrastes dois a dois.
grid <- apmc(X = L,
model = m0,
focus = "gen",
test = "fdr",
cld2 = FALSE)
grid <- grid[order(grid$fit, decreasing = TRUE), ]
ocld <- ordered_cld(grid$cld, grid$fit)
grid$cld <- ocld
# Gráfico de segmentos com as letras resumo das comparações múltiplas.
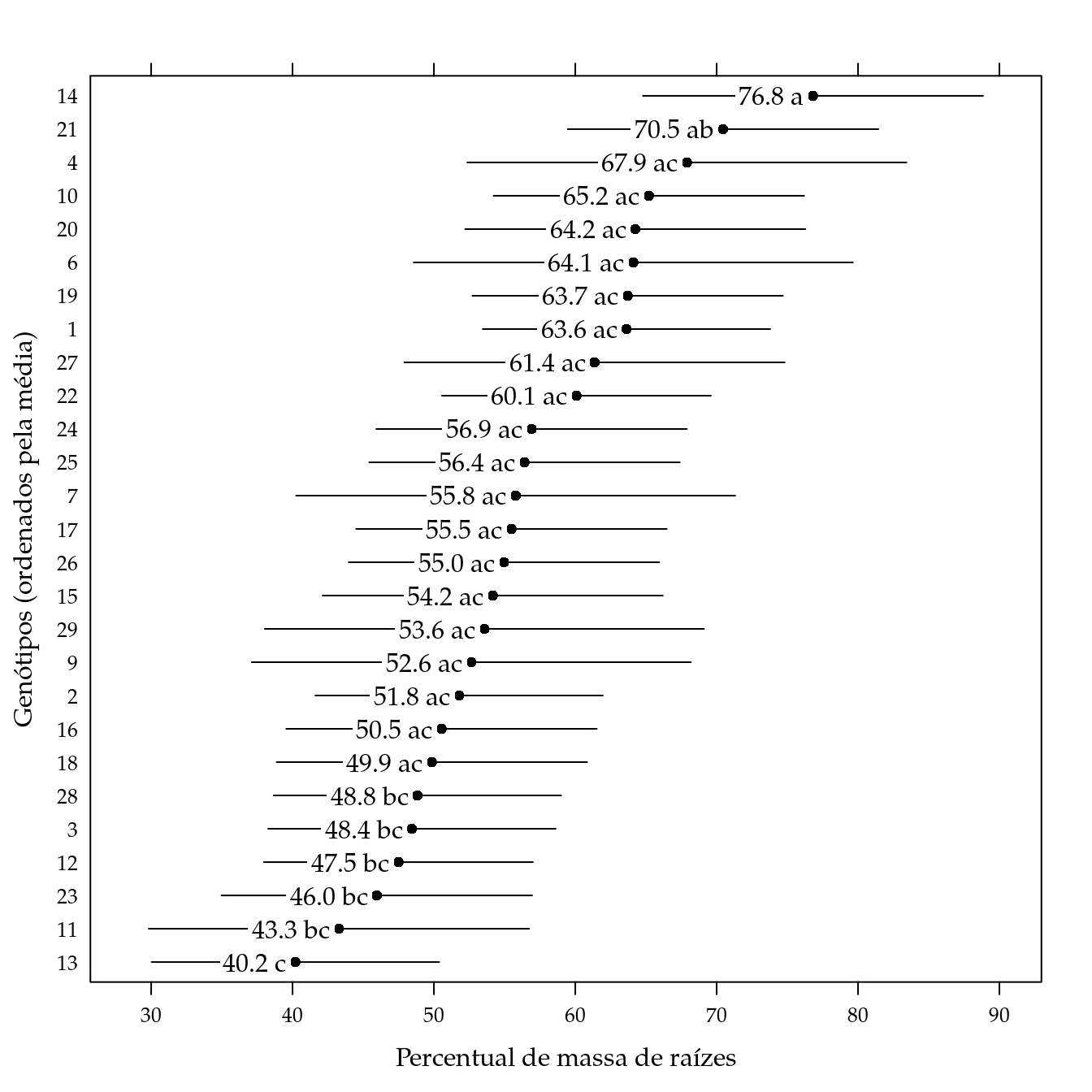
segplot(reorder(gen, fit) ~ lwr + upr,
centers = fit,
data = grid,
draw = FALSE,
xlab = "Percentual de massa de raízes",
ylab = "Genótipos (ordenados pela média)",
txt = sprintf("%0.1f %s", grid$fit, grid$cld)) +
layer({
x <- unit(centers, "native") - unit(0.005, "snpc")
y <- unit(z, "native")
grid.rect(x = x, y = y,
width = Reduce(unit.c,
lapply(txt,
FUN = unit,
x = 1.05,
units = "strwidth")) +
unit(0.005, "snpc"),
height = unit(1, "lines"),
just = "right",
gp = gpar(fill = "white",
col = NA,
fontsize = 12))
grid.text(label = txt,
just = "right",
x = x - unit(0.005, "snpc"),
y = y)
})
Altura
# Utilizar como variável concomitante a última data em foi medida em uma
# ancova.
str(da)## 'data.frame': 5800 obs. of 11 variables:
## $ gen : Factor w/ 29 levels "1","2","3","4",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ dose : num 0 0.0184 0.0368 0.0736 0.1472 ...
## $ data : Date, format: "2016-06-15" ...
## $ dac : num 38.6 40.2 43 40 39.8 23.8 33.9 37 40 41.8 ...
## $ alt : num 101 61.7 130 128 128 126 132 121 129 140 ...
## $ mspa : num NA NA NA NA NA NA NA NA NA NA ...
## $ msr : num NA NA NA NA NA NA NA NA NA NA ...
## $ dias : num 2 2 2 2 2 2 2 2 2 2 ...
## $ dos : Factor w/ 10 levels "0","1","2","3",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ dosep: num 0 0.302 0.371 0.457 0.563 ...
## $ ue : Factor w/ 290 levels "1.0","2.0","3.0",..: 1 30 59 88 117 146 175 204 233 262 ...# Função que extraí o registro completo mais velho de uma unidade
# experimental (última observação completa registrada).
last <- function(data, time, resp) {
tm <- data[, time]
data <- data[order(tm), ]
i <- max(which(!is.na(data[, resp])))
data[i, ]
}
# Reduz para uma tabela com uma linha por UE.
dc <- ddply(da,
~ue,
last, time = "data", resp = "alt")
str(dc)## 'data.frame': 290 obs. of 11 variables:
## $ gen : Factor w/ 29 levels "1","2","3","4",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ dose : num 0 0 0 0 0 0 0 0 0 0 ...
## $ data : Date, format: "2016-10-26" ...
## $ dac : num 88 76.1 105.9 51.6 109.2 ...
## $ alt : num 325 182 244 112 400 179 334 60 468 380 ...
## $ mspa : num 42.59 44.47 39.64 4.22 NA ...
## $ msr : num 47.9 53.5 49 11.4 78.2 ...
## $ dias : num 135 135 135 135 135 58 128 30 135 135 ...
## $ dos : Factor w/ 10 levels "0","1","2","3",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ dosep: num 0 0 0 0 0 0 0 0 0 0 ...
## $ ue : Factor w/ 290 levels "1.0","2.0","3.0",..: 1 2 3 4 5 6 7 8 9 10 ...xyplot(alt + dac ~ dias,
outer = TRUE,
data = da,
xlab = "Tempo após a primeira avaliação (dias)",
ylab = "Altura das mudas de teca (mm)",
col = "gray",
type = c("p", "smooth")) +
as.layer(xyplot(alt + dac ~ I(dias + 1),
data = dc,
outer = TRUE,
col = "blue",
pch = 19,
type = c("p", "smooth")))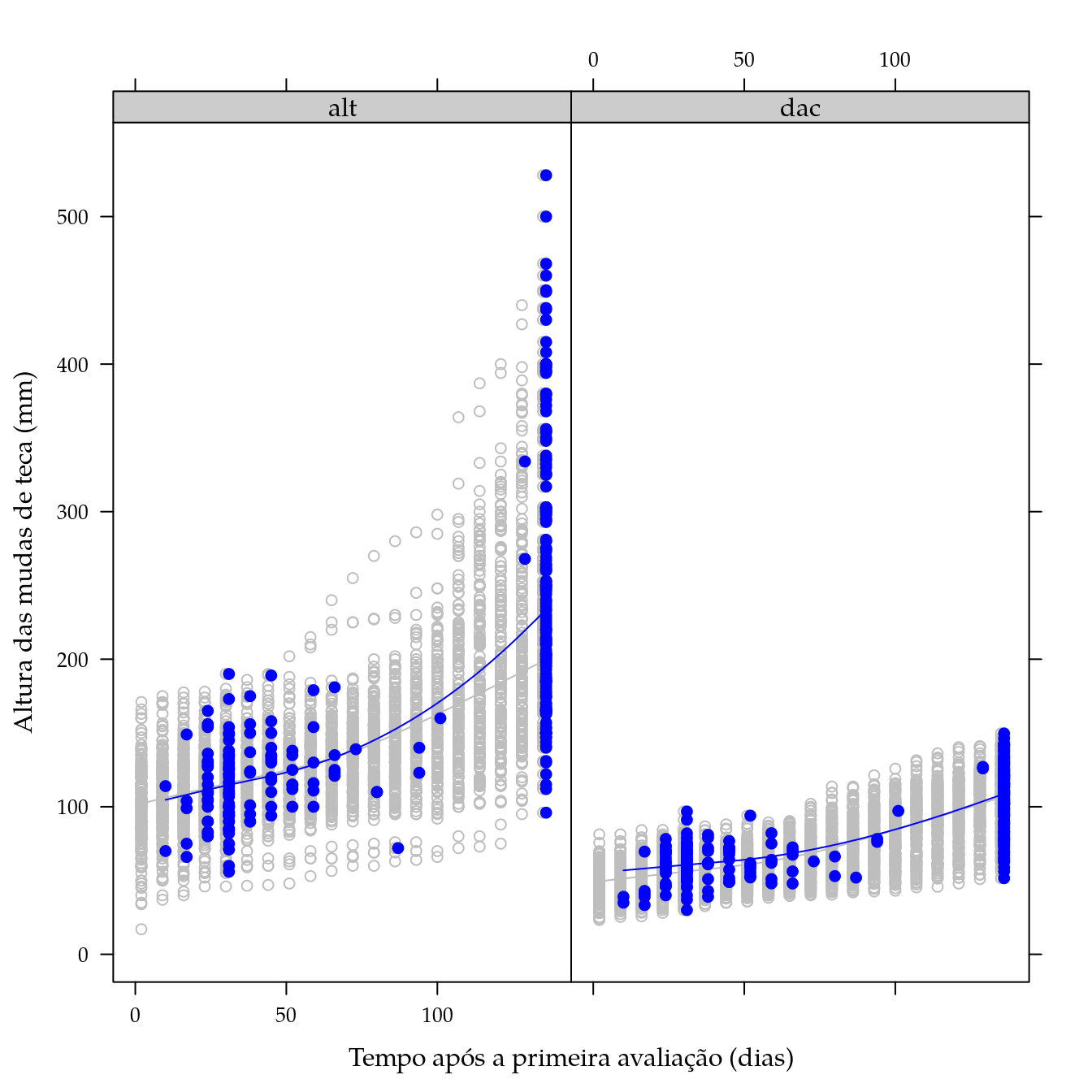
Para análise das variáveis altura e diâmetro à altura do colo será considerado análise de covariância. A covariável usada para corrigir os valores observados das respostas será o número de dias no último registro das variáveis. Dessa forma, todas as observações serão aproveitadas na análise, mesmo as que morreram antes do final do experimento.
Verfica-se que uma relação não linear entre a altura (diâmetro) em função do dia em que é medida. Cada ponto em azul no gráfico representa o último valor registrado das variáveis para uma unidade experimental, ou seja, é o valor das variáveis mais próximo do final do experimento, imediatamente antes da muda morrer. Dada a relação quadrática exibida, o efeito do tempo será considerado no modelo com um termo polinomial de grau 2.
# Modelo de efeitos quadráticos por genótipo.
m0 <- lm(alt ~ dias + I(dias^2) + gen * (dosep + I(dosep^2)),
data = dc)
# # Verificação dos pressupostos.
# par(mfrow = c(2, 2))
# plot(m0); layout(1)
# MASS::boxcox(m0)
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: alt
## Df Sum Sq Mean Sq F value Pr(>F)
## dias 1 1346945 1346945 310.0027 < 2.2e-16 ***
## I(dias^2) 1 26272 26272 6.0466 0.0147833 *
## gen 28 301451 10766 2.4778 0.0001522 ***
## dosep 1 107996 107996 24.8555 1.336e-06 ***
## I(dosep^2) 1 10278 10278 2.3656 0.1256161
## gen:dosep 28 114378 4085 0.9402 0.5564251
## gen:I(dosep^2) 28 73378 2621 0.6031 0.9435461
## Residuals 200 868989 4345
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Reduzindo o modelo com o abandono do termo quadrático e interação.
m0 <- update(m0, . ~ dias + I(dias^2) + gen + dosep)
anova(m0)## Analysis of Variance Table
##
## Response: alt
## Df Sum Sq Mean Sq F value Pr(>F)
## dias 1 1346945 1346945 324.4210 < 2.2e-16 ***
## I(dias^2) 1 26272 26272 6.3278 0.0125 *
## gen 28 301451 10766 2.5931 4.704e-05 ***
## dosep 1 107996 107996 26.0116 6.595e-07 ***
## Residuals 257 1067024 4152
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(m0)##
## Call:
## lm(formula = alt ~ dias + I(dias^2) + gen + dosep, data = dc)
##
## Residuals:
## Min 1Q Median 3Q Max
## -152.79 -37.90 -4.71 29.92 214.71
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 145.160655 31.157756 4.659 5.11e-06 ***
## dias -1.026481 0.839520 -1.223 0.22256
## I(dias^2) 0.013895 0.005057 2.747 0.00643 **
## gen2 -24.743539 28.834653 -0.858 0.39163
## gen3 20.672477 28.844344 0.717 0.47422
## gen4 4.959274 29.131254 0.170 0.86496
## gen5 9.890866 29.010954 0.341 0.73343
## gen6 28.156789 29.152219 0.966 0.33503
## gen7 22.478053 29.034382 0.774 0.43953
## gen8 0.041108 30.097746 0.001 0.99891
## gen9 64.167965 28.941270 2.217 0.02749 *
## gen10 12.765646 28.880980 0.442 0.65885
## gen11 14.896021 28.942090 0.515 0.60722
## gen12 27.494495 28.817248 0.954 0.34093
## gen13 75.100000 28.816118 2.606 0.00969 **
## gen14 77.156956 28.959852 2.664 0.00820 **
## gen15 63.562499 28.829519 2.205 0.02836 *
## gen16 44.974263 28.870276 1.558 0.12051
## gen17 19.892636 28.833911 0.690 0.49088
## gen18 21.056149 29.017472 0.726 0.46872
## gen19 40.135602 28.863130 1.391 0.16557
## gen20 32.522052 28.920526 1.125 0.26184
## gen21 14.874796 28.867703 0.515 0.60680
## gen22 34.150331 28.837773 1.184 0.23742
## gen23 -61.810695 29.056100 -2.127 0.03435 *
## gen24 10.487665 28.838070 0.364 0.71640
## gen25 79.385805 28.866006 2.750 0.00638 **
## gen26 83.053311 28.971415 2.867 0.00449 **
## gen27 14.482783 29.224624 0.496 0.62062
## gen28 -9.496780 28.944889 -0.328 0.74310
## gen29 -12.135498 29.147721 -0.416 0.67751
## dosep -42.705333 8.373344 -5.100 6.59e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 64.43 on 257 degrees of freedom
## (1 observation deleted due to missingness)
## Multiple R-squared: 0.6256, Adjusted R-squared: 0.5804
## F-statistic: 13.85 on 31 and 257 DF, p-value: < 2.2e-16Pelo quadro de ANOVA não existe interação entre genótipo e dose de cálcio. Verifica-se a existência apenas dos efeitos aditivos. No gráfico a seguir é representada a relação entre altura final e dose de cálcio para cada genótipo. Segundo o gráfico e também a estimativa do coeficiente do efeito de Ca, a altura do genótipo diminui com o aumento da dose de cálcio.
# Último dia de medição das mudas.
dmax <- max(dc$dias, na.rm = TRUE)
# Cria um conjunto de valores para predição.
pred <- with(dc,
expand.grid(
dosep = eseq(dosep, f = 0),
dias = dmax,
gen = levels(gen)))
ic <- predict(m0, newdata = pred, interval = "confidence")
pred <- cbind(pred, as.data.frame(ic))
xyplot(alt ~ dosep | gen,
data = subset(dc, dias == max(dias, na.rm = TRUE)),
# type = c("p", "r"),
xlab = "Dose de cálcio (escala transformada)",
ylab = "Altura das mudas (mm)") +
as.layer(xyplot(fit ~ dosep | gen,
data = pred,
type = "l",
ly = pred$lwr,
uy = pred$upr,
cty = "bands",
prepanel = prepanel.cbH,
panel = panel.cbH))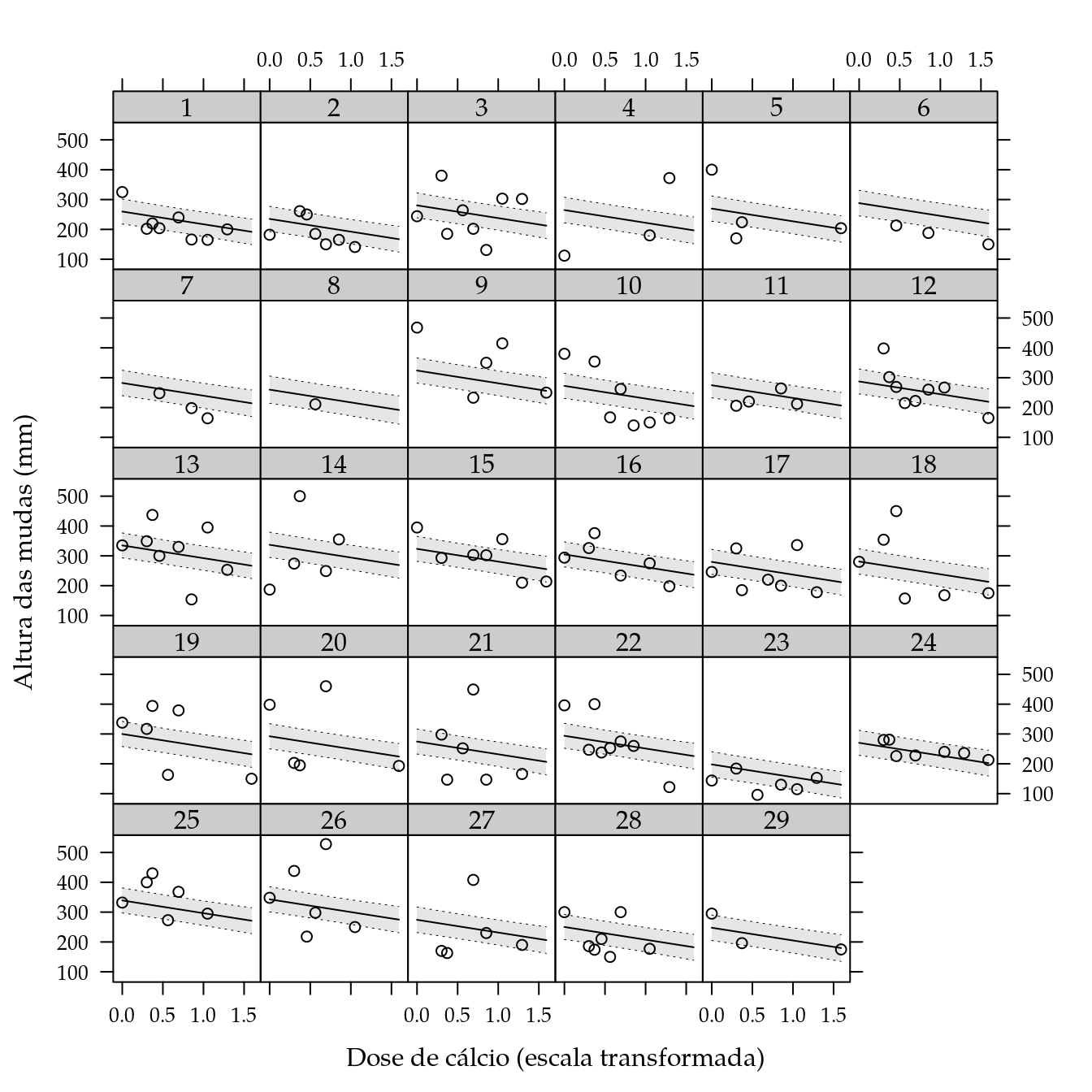
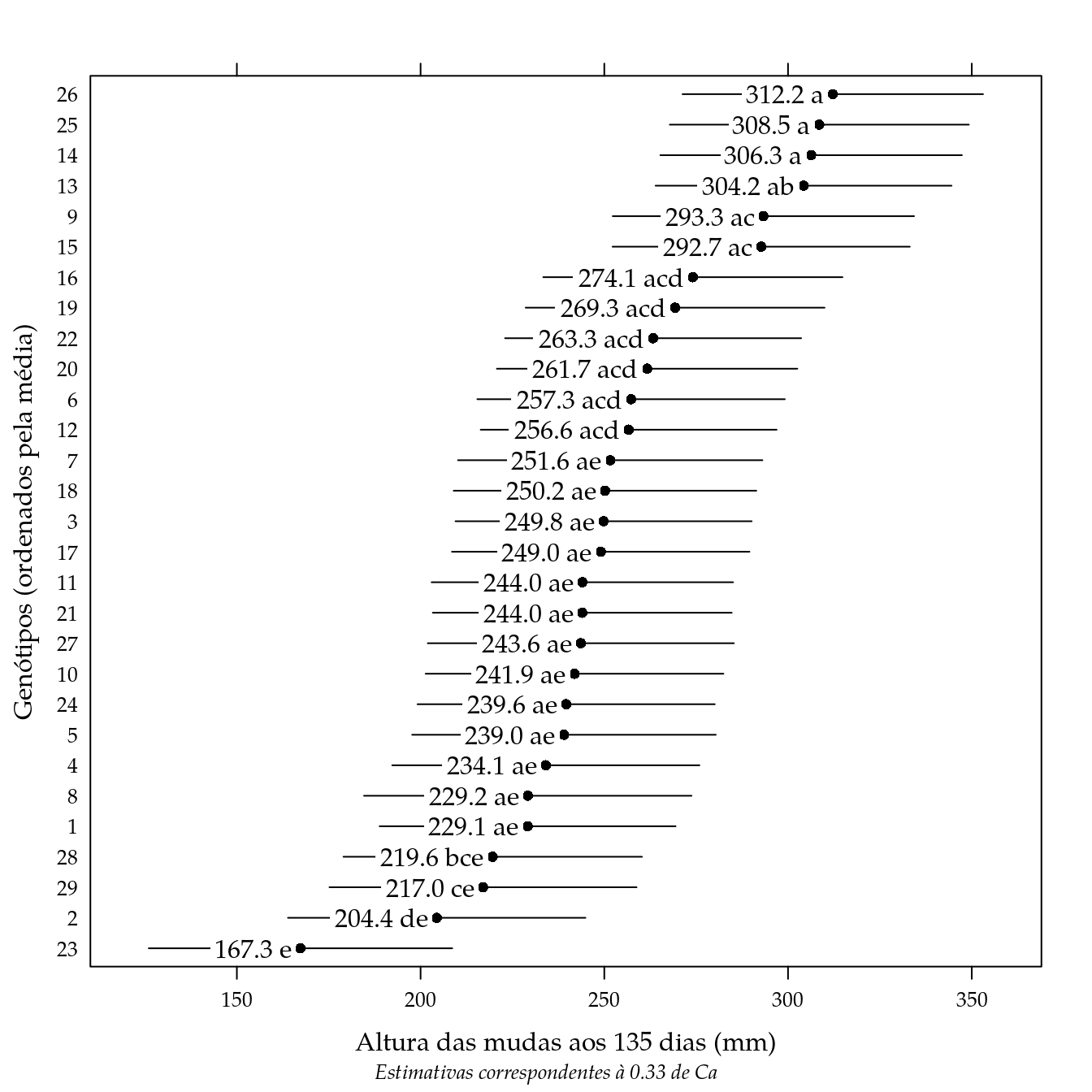
As médias de altura dos genótipos serão comparadas considerando a altura final, ou seja, aquela prevista para os 135 dias.
# Médias ajustadas, considerando um valor fixo de cálcio para o último
# dia de medição.
lsm <- LSmeans(m0,
effect = "gen",
at = list("dias" = dmax, "I(dias^2)" = dmax^2))
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 229.1299 20.4503 257.0000 11.2042 0
## [2,] 204.3863 20.5556 257.0000 9.9431 0
## [3,] 249.8023 20.4643 257.0000 12.2067 0
## [4,] 234.0891 21.2262 257.0000 11.0283 0
## [5,] 239.0207 20.9792 257.0000 11.3932 0
## [6,] 257.2867 21.2449 257.0000 12.1105 0
## [7,] 251.6079 21.0358 257.0000 11.9609 0
## [8,] 229.1710 22.6282 257.0000 10.1277 0
## [9,] 293.2978 20.8198 257.0000 14.0874 0
## [10,] 241.8955 20.5791 257.0000 11.7544 0
## [11,] 244.0259 20.8308 257.0000 11.7146 0
## [12,] 256.6244 20.4468 257.0000 12.5509 0
## [13,] 304.2299 20.4503 257.0000 14.8765 0
## [14,] 306.2868 20.8355 257.0000 14.7002 0
## [15,] 292.6924 20.5378 257.0000 14.2514 0
## [16,] 274.1041 20.6738 257.0000 13.2585 0
## [17,] 249.0225 20.5545 257.0000 12.1152 0
## [18,] 250.1860 20.9100 257.0000 11.9649 0
## [19,] 269.2655 20.6535 257.0000 13.0373 0
## [20,] 261.6519 20.7617 257.0000 12.6026 0
## [21,] 244.0047 20.6546 257.0000 11.8136 0
## [22,] 263.2802 20.4601 257.0000 12.8680 0
## [23,] 167.3192 20.9688 257.0000 7.9795 0
## [24,] 239.6175 20.5395 257.0000 11.6662 0
## [25,] 308.5157 20.6625 257.0000 14.9312 0
## [26,] 312.1832 20.7635 257.0000 15.0352 0
## [27,] 243.6126 21.1519 257.0000 11.5173 0
## [28,] 219.6331 20.6240 257.0000 10.6494 0
## [29,] 216.9944 21.2194 257.0000 10.2262 0# Dose usada para obter as médias (é a média de cálcio dos dados
# observados).
d <- lsm$grid$dosep[1]^(1/0.3)
d## [1] 0.3327573# Atribui nomes às linhas da matriz.
L <- lsm$L
rownames(L) <- levels(dc$gen)
# Obtém os contrastes dois a dois.
grid <- apmc(X = L,
model = m0,
focus = "gen",
test = "fdr",
cld2 = FALSE)
grid <- grid[order(grid$fit, decreasing = TRUE), ]
ocld <- ordered_cld(grid$cld, grid$fit)
grid$cld <- ocld
# Gráfico de segmentos com as letras resumo das comparações múltiplas.
segplot(reorder(gen, fit) ~ lwr + upr,
centers = fit,
data = grid,
draw = FALSE,
xlab = sprintf("Altura das mudas aos %d dias (mm)", dmax),
ylab = "Genótipos (ordenados pela média)",
sub = list(
sprintf("Estimativas correspondentes à %0.2f de Ca", d),
font = 3, cex = 0.8),
txt = sprintf("%0.1f %s", grid$fit, grid$cld)) +
layer({
x <- unit(centers, "native") - unit(0.005, "snpc")
y <- unit(z, "native")
grid.rect(x = x, y = y,
width = Reduce(unit.c,
lapply(txt,
FUN = unit,
x = 1.05,
units = "strwidth")) +
unit(0.005, "snpc"),
height = unit(1, "lines"),
just = "right",
gp = gpar(fill = "white",
col = NA,
fontsize = 12))
grid.text(label = txt,
just = "right",
x = x - unit(0.005, "snpc"),
y = y)
})
Diâmetro à altura do colo
A análise para diâmetro à altura do colo segue o mesmo roteiro da análise para altura das mudas.
# Modelo de efeitos quadráticos por genótipo.
m0 <- lm(dac ~ dias + I(dias^2) + gen * (dosep + I(dosep^2)),
data = dc)
# # Verificação dos pressupostos.
# par(mfrow = c(2, 2))
# plot(m0); layout(1)
# MASS::boxcox(m0)
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: dac
## Df Sum Sq Mean Sq F value Pr(>F)
## dias 1 140642 140642 512.5683 < 2.2e-16 ***
## I(dias^2) 1 1680 1680 6.1240 0.01417 *
## gen 28 27876 996 3.6284 5.305e-08 ***
## dosep 1 466 466 1.6977 0.19409
## I(dosep^2) 1 231 231 0.8426 0.35977
## gen:dosep 28 10831 387 1.4097 0.09281 .
## gen:I(dosep^2) 28 5488 196 0.7143 0.85411
## Residuals 200 54877 274
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Reduzindo o modelo com o abandono do termo quadrático e interação.
m0 <- update(m0, . ~ dias + I(dias^2) + gen)
anova(m0)## Analysis of Variance Table
##
## Response: dac
## Df Sum Sq Mean Sq F value Pr(>F)
## dias 1 140642 140642 504.7161 < 2.2e-16 ***
## I(dias^2) 1 1680 1680 6.0302 0.01472 *
## gen 28 27876 996 3.5728 3.223e-08 ***
## Residuals 258 71893 279
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(m0)##
## Call:
## lm(formula = dac ~ dias + I(dias^2) + gen, data = dc)
##
## Residuals:
## Min 1Q Median 3Q Max
## -55.793 -9.264 0.755 9.833 49.427
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 56.600728 7.891835 7.172 7.8e-12 ***
## dias -0.263033 0.217329 -1.210 0.22727
## I(dias^2) 0.004164 0.001308 3.183 0.00164 **
## gen2 -8.018830 7.470035 -1.073 0.28406
## gen3 15.496212 7.472634 2.074 0.03910 *
## gen4 -5.283248 7.544041 -0.700 0.48436
## gen5 6.101576 7.514009 0.812 0.41752
## gen6 14.493647 7.549360 1.920 0.05598 .
## gen7 20.382237 7.519841 2.710 0.00717 **
## gen8 4.675344 7.795536 0.600 0.54920
## gen9 12.473500 7.496640 1.664 0.09735 .
## gen10 6.614733 7.481964 0.884 0.37747
## gen11 17.733836 7.496809 2.366 0.01874 *
## gen12 19.657242 7.465616 2.633 0.00897 **
## gen13 21.320000 7.465323 2.856 0.00464 **
## gen14 8.587985 7.501422 1.145 0.25333
## gen15 18.410475 7.468671 2.465 0.01435 *
## gen16 14.878431 7.478959 1.989 0.04772 *
## gen17 14.821973 7.469839 1.984 0.04829 *
## gen18 -5.479486 7.517263 -0.729 0.46671
## gen19 11.124523 7.477072 1.488 0.13802
## gen20 6.664457 7.491505 0.890 0.37451
## gen21 7.794979 7.478214 1.042 0.29822
## gen22 21.090577 7.470931 2.823 0.00513 **
## gen23 -18.818336 7.527289 -2.500 0.01304 *
## gen24 20.405763 7.470871 2.731 0.00674 **
## gen25 17.930494 7.477796 2.398 0.01720 *
## gen26 12.710496 7.504954 1.694 0.09155 .
## gen27 -2.040574 7.569789 -0.270 0.78771
## gen28 8.197417 7.498590 1.093 0.27533
## gen29 3.192171 7.548354 0.423 0.67272
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 16.69 on 258 degrees of freedom
## (1 observation deleted due to missingness)
## Multiple R-squared: 0.703, Adjusted R-squared: 0.6685
## F-statistic: 20.36 on 30 and 258 DF, p-value: < 2.2e-16# Último dia de medição das mudas.
dmax <- max(dc$dias, na.rm = TRUE)
# Cria um conjunto de valores para predição.
pred <- with(dc,
expand.grid(
dosep = eseq(dosep, f = 0),
dias = dmax,
gen = levels(gen)))
ic <- predict(m0, newdata = pred, interval = "confidence")
pred <- cbind(pred, as.data.frame(ic))
xyplot(dac ~ dosep | gen,
data = subset(dc, dias == max(dias, na.rm = TRUE)),
# type = c("p", "r"),
xlab = "Dose de cálcio (escala transformada)",
ylab = "Altura das mudas (mm)") +
as.layer(xyplot(fit ~ dosep | gen,
data = pred,
type = "l",
ly = pred$lwr,
uy = pred$upr,
cty = "bands",
prepanel = prepanel.cbH,
panel = panel.cbH))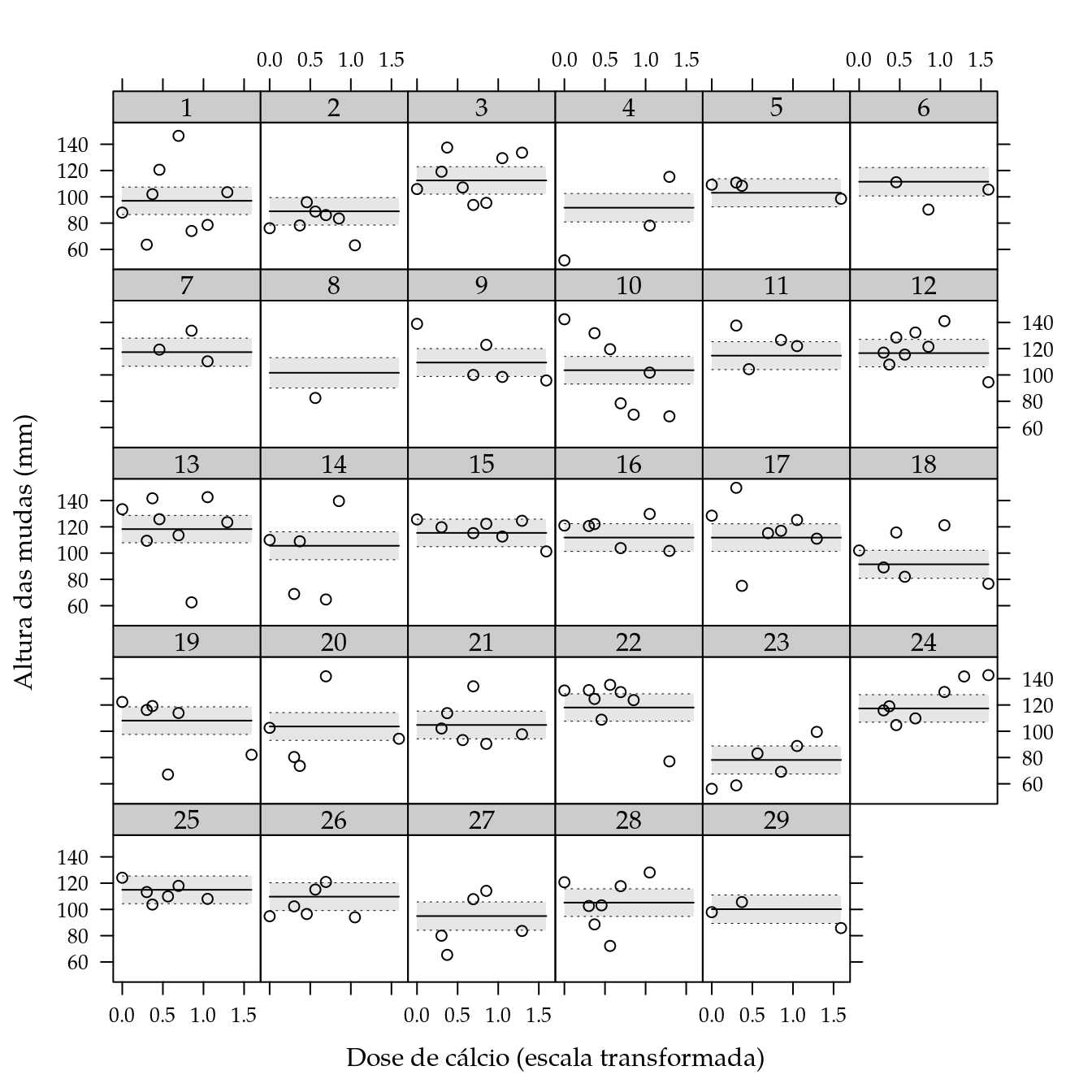
# Médias ajustadas, considerando um valor fixo de cálcio para o último
# dia de medição.
lsm <- LSmeans(m0,
effect = "gen",
at = list("dias" = dmax, "I(dias^2)" = dmax^2))
lsm## Coefficients:
## estimate se df t.stat p.value
## [1,] 96.9733 5.2973 258.0000 18.3060 0
## [2,] 88.9544 5.3239 258.0000 16.7085 0
## [3,] 112.4695 5.3009 258.0000 21.2171 0
## [4,] 91.6900 5.4911 258.0000 16.6978 0
## [5,] 103.0748 5.4294 258.0000 18.9847 0
## [6,] 111.4669 5.4958 258.0000 20.2823 0
## [7,] 117.3555 5.4435 258.0000 21.5587 0
## [8,] 101.6486 5.8568 258.0000 17.3557 0
## [9,] 109.4468 5.3895 258.0000 20.3073 0
## [10,] 103.5880 5.3297 258.0000 19.4360 0
## [11,] 114.7071 5.3923 258.0000 21.2724 0
## [12,] 116.6305 5.2964 258.0000 22.0207 0
## [13,] 118.2933 5.2973 258.0000 22.3307 0
## [14,] 105.5612 5.3935 258.0000 19.5719 0
## [15,] 115.3837 5.3192 258.0000 21.6921 0
## [16,] 111.8517 5.3535 258.0000 20.8932 0
## [17,] 111.7952 5.3236 258.0000 20.9999 0
## [18,] 91.4938 5.4152 258.0000 16.8956 0
## [19,] 108.0978 5.3481 258.0000 20.2123 0
## [20,] 103.6377 5.3750 258.0000 19.2813 0
## [21,] 104.7682 5.3483 258.0000 19.5891 0
## [22,] 118.0638 5.2998 258.0000 22.2771 0
## [23,] 78.1549 5.4305 258.0000 14.3918 0
## [24,] 117.3790 5.3195 258.0000 22.0657 0
## [25,] 114.9037 5.3504 258.0000 21.4758 0
## [26,] 109.6837 5.3761 258.0000 20.4019 0
## [27,] 94.9327 5.4750 258.0000 17.3393 0
## [28,] 105.1707 5.3416 258.0000 19.6889 0
## [29,] 100.1654 5.4895 258.0000 18.2468 0# Atribui nomes às linhas da matriz.
L <- lsm$L
rownames(L) <- levels(dc$gen)
# Obtém os contrastes dois a dois.
grid <- apmc(X = L,
model = m0,
focus = "gen",
test = "fdr",
cld2 = FALSE)
grid <- grid[order(grid$fit, decreasing = TRUE), ]
ocld <- ordered_cld(grid$cld, grid$fit)
grid$cld <- ocld
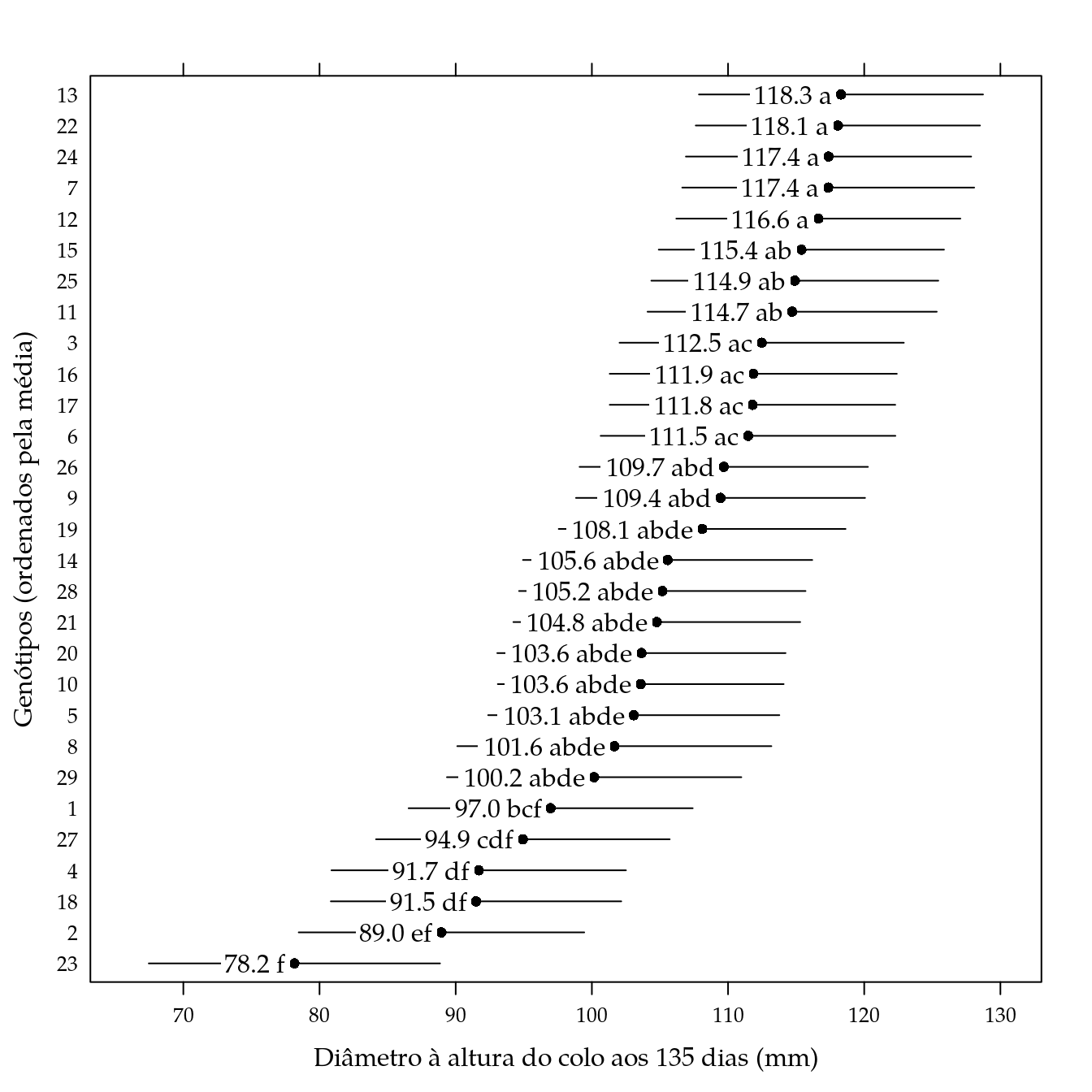
# Gráfico de segmentos com as letras resumo das comparações múltiplas.
segplot(reorder(gen, fit) ~ lwr + upr,
centers = fit,
data = grid,
draw = FALSE,
xlab = sprintf("Diâmetro à altura do colo aos %d dias (mm)", dmax),
ylab = "Genótipos (ordenados pela média)",
# sub = list(
# sprintf("Estimativas correspondentes à %0.2f de Ca", d),
# font = 3, cex = 0.8),
txt = sprintf("%0.1f %s", grid$fit, grid$cld)) +
layer({
x <- unit(centers, "native") - unit(0.005, "snpc")
y <- unit(z, "native")
grid.rect(x = x, y = y,
width = Reduce(unit.c,
lapply(txt,
FUN = unit,
x = 1.05,
units = "strwidth")) +
unit(0.005, "snpc"),
height = unit(1, "lines"),
just = "right",
gp = gpar(fill = "white",
col = NA,
fontsize = 12))
grid.text(label = txt,
just = "right",
x = x - unit(0.005, "snpc"),
y = y)
})
Informações da Sessão
## Atualizado em 11 de julho de 2019.
##
## R version 3.6.1 (2019-07-05)
## Platform: x86_64-pc-linux-gnu (64-bit)
## Running under: Ubuntu 18.04.2 LTS
##
## Matrix products: default
## BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.7.1
## LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.7.1
##
## locale:
## [1] LC_CTYPE=pt_BR.UTF-8 LC_NUMERIC=C
## [3] LC_TIME=pt_BR.UTF-8 LC_COLLATE=en_US.UTF-8
## [5] LC_MONETARY=pt_BR.UTF-8 LC_MESSAGES=en_US.UTF-8
## [7] LC_PAPER=pt_BR.UTF-8 LC_NAME=C
## [9] LC_ADDRESS=C LC_TELEPHONE=C
## [11] LC_MEASUREMENT=pt_BR.UTF-8 LC_IDENTIFICATION=C
##
## attached base packages:
## [1] grid stats graphics grDevices utils datasets
## [7] methods base
##
## other attached packages:
## [1] multcomp_1.4-10 TH.data_1.0-10 MASS_7.3-51.4
## [4] survival_2.44-1.1 mvtnorm_1.0-11 doBy_4.6-2
## [7] plyr_1.8.4 gridExtra_2.3 EACS_0.0-7
## [10] wzRfun_0.91 captioner_2.2.3 latticeExtra_0.6-28
## [13] RColorBrewer_1.1-2 lattice_0.20-38 knitr_1.23
##
## loaded via a namespace (and not attached):
## [1] zoo_1.8-6 tidyselect_0.2.5 xfun_0.8
## [4] remotes_2.1.0 purrr_0.3.2 splines_3.6.1
## [7] testthat_2.1.1 htmltools_0.3.6 usethis_1.5.1
## [10] yaml_2.2.0 rlang_0.4.0 pkgbuild_1.0.3
## [13] pkgdown_1.3.0 pillar_1.4.2 glue_1.3.1
## [16] withr_2.1.2 sessioninfo_1.1.1 stringr_1.4.0
## [19] commonmark_1.7 gtable_0.3.0 devtools_2.1.0
## [22] codetools_0.2-16 memoise_1.1.0 evaluate_0.14
## [25] callr_3.3.0 ps_1.3.0 Rcpp_1.0.1
## [28] backports_1.1.4 desc_1.2.0 pkgload_1.0.2
## [31] fs_1.3.1 digest_0.6.20 stringi_1.4.3
## [34] processx_3.4.0 dplyr_0.8.3 rprojroot_1.3-2
## [37] cli_1.1.0 tools_3.6.1 sandwich_2.5-1
## [40] magrittr_1.5 tibble_2.1.3 crayon_1.3.4
## [43] pkgconfig_2.0.2 Matrix_1.2-17 xml2_1.2.0
## [46] prettyunits_1.0.2 assertthat_0.2.1 rmarkdown_1.13
## [49] roxygen2_6.1.1 rstudioapi_0.10 R6_2.4.0
## [52] compiler_3.6.1