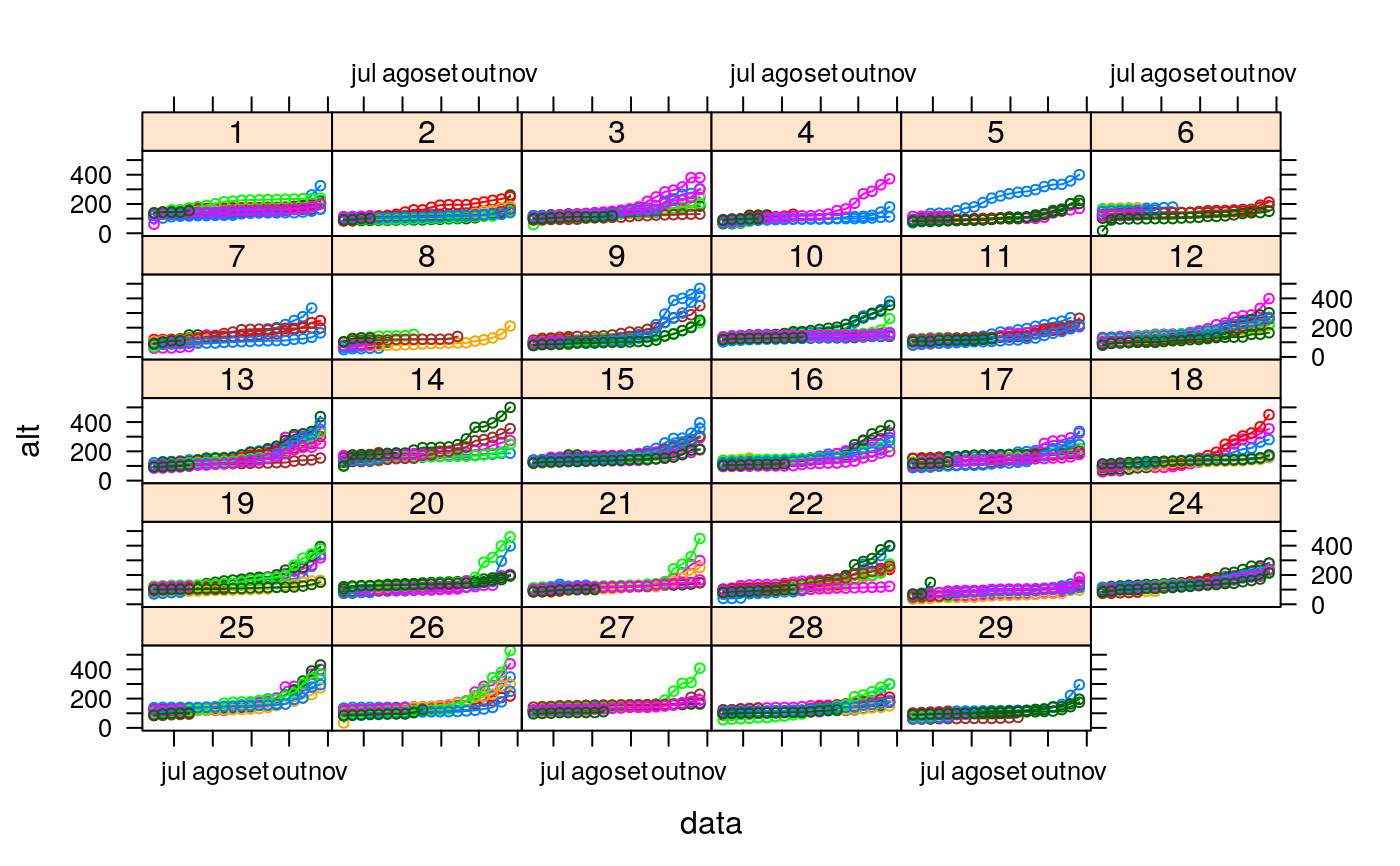
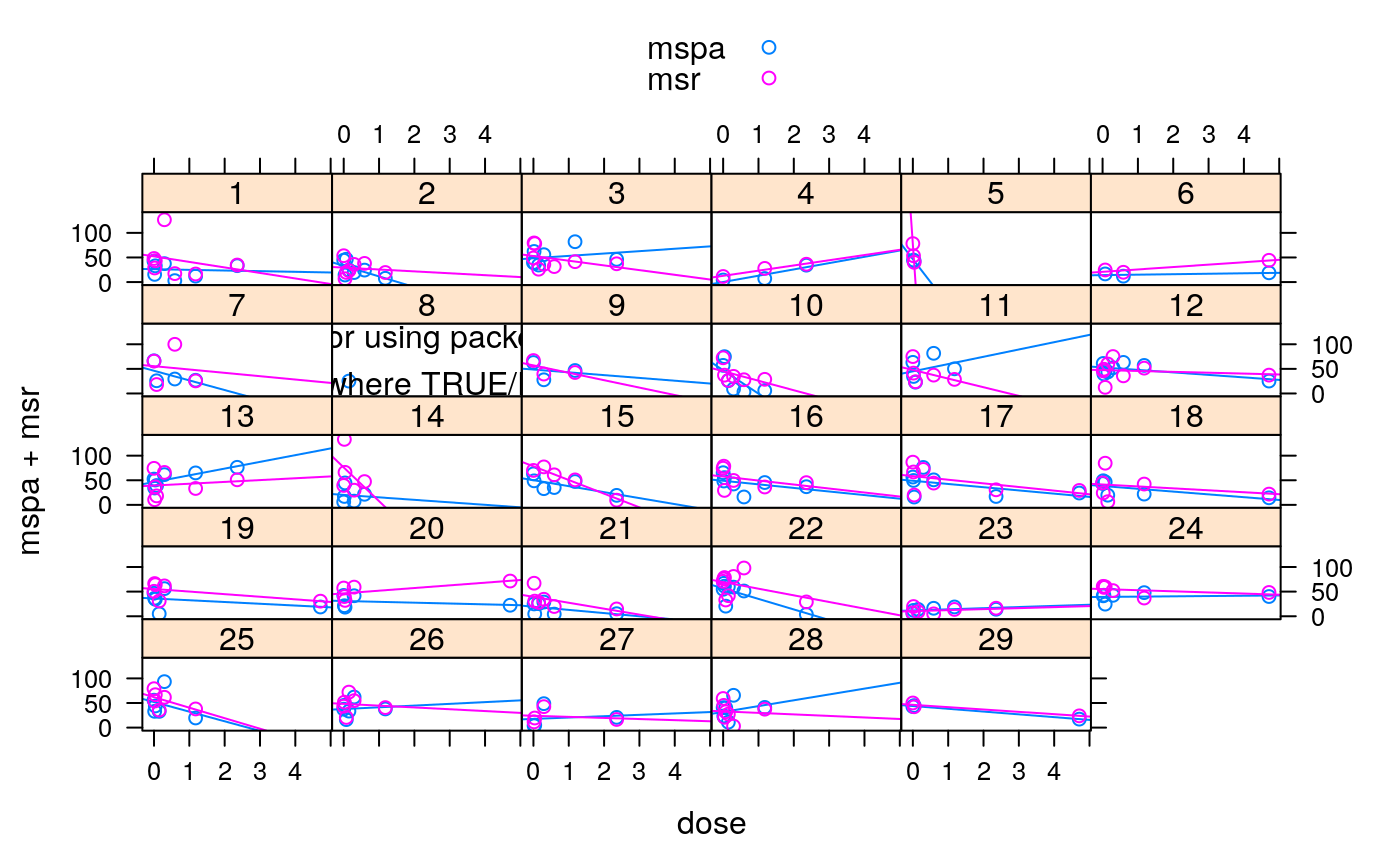
O experimento foi instalado em casa de vegetação no mês de Junho do ano de 2016, utilizando vasos de polietileno com 5 dm\(^3\) de solo. A umidade do solo foi mantida ente 60-70% da capacidade de campo. Foram estudadas doses crescentes de cálcio (Ca) para estudar o desenvolvimento de mudas de teca de diferentes genótipos. Os fatores de estudo foram 3: genótipos de teca, doses de Ca, e o tempo. Os níveis do fator genótipo foram 29, identificados com números de 1 a 29. Os níveis do fator Ca foram 10, espaçados na escala log base 2 com inclusão da dose 0. Os níveis do fator tempo foram 20 medidas a partir da data do plantio. O experimento foi instalado em casa de vegetação com delineamento inteiramente casualizado e a unidade experimental foi um vaso com uma planta de teca. No experimento foram obtidas as seguintes váriaveis respostas: altura de planta (mm) medida semanalmente, diâmetro altura do colo (mm) medida semanalmente, massa seca de parte aérea (g) e massa seca de raiz (g), ambos medidos ao final do experimento, já que a determinação é destrutiva. Ao longo do experimento algumas plantas morreram então o período de sobrevivência pode ser determinado pela ultima data em que foi realizado registro das variáveis. Essa variável tem censura intervalar pois sabe-se que a planta morreu entre dois instantes de tempo conhecido.
Format
Um data.frame com 5800 observações e 7 variáveis, em
que
genFator de níveis categóricos que representa os genótipos de teca estudados.
doseFator de níveis métricos que representa a dose de Ca aplicada ao solo na forma de adubação para desenvolvimento das mudas, em cmol\(_c\) dm\(^{-3}\).
dataVariável cronológica que representa as datas de avaliação das unidades experimentais para as variáveis longitudinais altura e diâmetro a altura do colo.
dacDiâmetro à altura do colo (mm) das mudas de teca
altAltura total (mm) das mudas de teca.
mspaMassa seca de parte aérea (g) das mudas ao final do experimento.
msrMassa seca de raízes (g) das mudas ao final do experimento.
Source
Serafim, Milson Evaldo (Orientador), Valteir Siani (TCC Engenharia florestal) (2016). Resposta de genótipos de teca em resposta a doses de Ca.
Examples
#> 'data.frame': 5800 obs. of 7 variables: #> $ gen : Factor w/ 29 levels "1","2","3","4",..: 1 1 1 1 1 1 1 1 1 1 ... #> $ dose: num 0 0.0184 0.0368 0.0736 0.1472 ... #> $ data: Date, format: "2016-06-15" "2016-06-15" ... #> $ dac : num 38.6 40.2 43 40 39.8 23.8 33.9 37 40 41.8 ... #> $ alt : num 101 61.7 130 128 128 126 132 121 129 140 ... #> $ mspa: num NA NA NA NA NA NA NA NA NA NA ... #> $ msr : num NA NA NA NA NA NA NA NA NA NA ...#> dose #> gen 0 0.0184 0.0368 0.0736 0.1472 0.2944 0.5888 1.1776 2.3552 4.7104 #> 1 20 20 20 20 5 20 20 20 20 5 #> 2 20 6 20 20 20 20 20 20 4 4 #> 3 20 20 20 5 20 20 20 20 20 10 #> 4 20 6 6 9 4 5 5 20 20 5 #> 5 20 20 20 12 5 5 5 5 5 20 #> 6 9 6 6 20 5 4 20 7 5 20 #> 7 19 7 6 20 7 5 20 20 4 4 #> 8 5 0 6 5 20 9 14 3 4 4 #> 9 20 8 8 5 5 20 20 20 4 20 #> 10 20 7 20 5 20 20 20 20 20 10 #> 11 19 20 4 20 5 5 20 20 5 10 #> 12 7 20 20 20 20 20 20 20 4 20 #> 13 20 20 20 20 5 20 20 20 20 5 #> 14 20 20 20 5 5 20 20 5 10 7 #> 15 20 20 6 5 5 20 20 20 20 20 #> 16 20 20 20 4 4 20 4 20 20 8 #> 17 20 20 20 4 5 20 20 20 20 5 #> 18 20 20 6 20 20 2 3 20 3 20 #> 19 20 20 20 9 20 20 4 4 5 20 #> 20 20 20 20 7 5 20 5 4 13 20 #> 21 4 20 20 4 20 20 20 7 20 8 #> 22 20 20 20 20 20 20 20 5 20 9 #> 23 20 20 3 3 20 5 20 20 20 2 #> 24 5 20 20 20 8 20 5 20 19 20 #> 25 20 20 20 7 20 20 5 20 4 5 #> 26 20 20 7 20 20 20 5 20 8 10 #> 27 11 20 20 7 7 20 20 8 20 9 #> 28 20 20 20 20 20 20 5 20 12 14 #> 29 20 6 20 5 5 5 13 5 7 20#> dose #> gen 0 0.0184 0.0368 0.0736 0.1472 0.2944 0.5888 1.1776 2.3552 4.7104 Sum #> 1 1 1 1 0 0 1 1 1 1 0 7 #> 2 1 0 1 1 1 1 1 1 0 0 7 #> 3 1 1 1 0 1 1 0 1 1 0 7 #> 4 1 0 0 0 0 0 0 1 1 0 3 #> 5 0 1 1 0 0 0 0 0 0 0 2 #> 6 0 0 0 1 0 0 1 0 0 1 3 #> 7 1 0 0 1 0 0 1 1 0 0 4 #> 8 0 0 0 0 1 0 0 0 0 0 1 #> 9 1 0 0 0 0 1 0 1 0 0 3 #> 10 1 0 1 0 1 1 1 1 0 0 6 #> 11 1 1 0 1 0 0 1 1 0 0 5 #> 12 0 1 1 1 1 1 1 1 0 1 8 #> 13 1 1 1 1 0 1 0 1 1 0 7 #> 14 1 1 1 0 0 1 1 0 0 0 5 #> 15 1 1 0 0 0 1 1 1 1 0 6 #> 16 1 1 1 0 0 1 1 1 1 0 7 #> 17 1 1 1 0 0 1 1 0 1 1 7 #> 18 1 1 0 1 1 0 0 1 0 1 6 #> 19 1 1 1 0 1 1 0 0 0 1 6 #> 20 1 1 1 0 0 1 0 0 0 1 5 #> 21 0 1 1 0 1 1 1 0 1 0 6 #> 22 1 1 1 1 1 1 1 0 1 0 8 #> 23 1 1 0 0 1 0 1 1 1 0 6 #> 24 0 1 1 1 0 1 0 1 0 1 6 #> 25 1 1 1 0 1 1 0 1 0 0 6 #> 26 1 1 0 1 1 1 0 1 0 0 6 #> 27 0 1 1 0 0 1 0 0 1 0 4 #> 28 1 1 1 1 1 1 0 1 0 0 7 #> 29 1 0 1 0 0 0 0 0 0 1 3 #> Sum 22 21 19 11 13 20 14 18 11 8 157xyplot(mspa + msr ~ dose | gen, data = gen_teca, auto.key = TRUE, type = c("p", "r"), as.table = TRUE)