Efeito da Época de Colheita e Adubação Nitro-Fosfatada na Carobinha
Source:R/np_carobinha.R
np_carobinha.RdResultados de um experimento realizado para estudar o efeito da época de colheita e níveis de adubação nitro-fosfatada sobre a produção e teores de nutrientes na carobinha (subsp. Symmetrifoliolata).
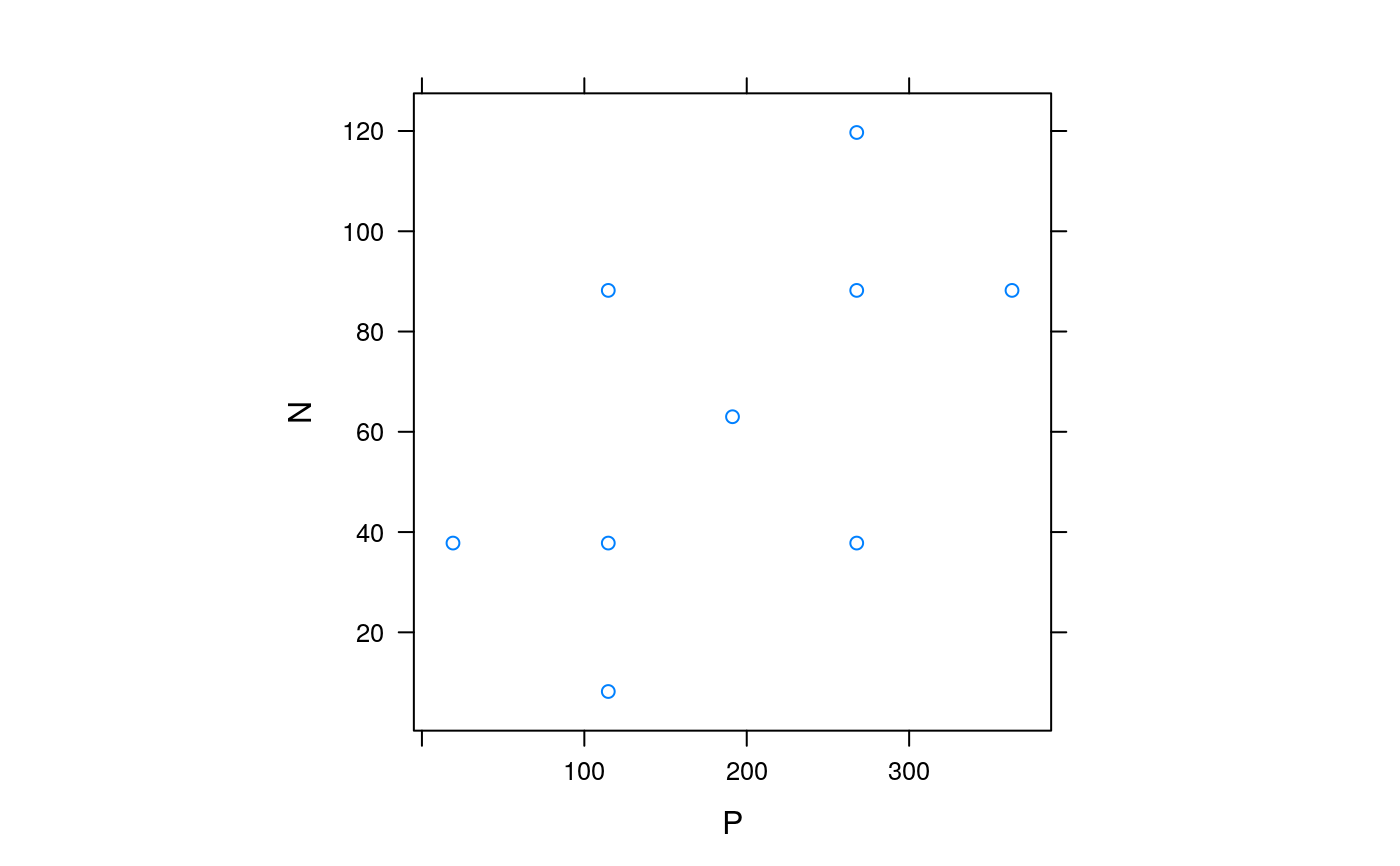
O experimento foi instalado em ambiente protegido em maio/2012. 4 vasos preenchidos com Latossolo vermelho distroférrico muito argiloso, com peso de 5.5 kg, foram adubados com nitrogênio (5 níveis, kg ha^-1) e fósforo (5 níveis, kg ha^-1). Um total de 25 combinações de N e P resultam do cruzamento completo dos níveis. No entanto, para reduzir a dimensão do experimento, utilizou-se 9 combinações ao considerar a matriz experimental de Pan Plueba III. O delineamento experimental foi de blocos casualizados (DBC). Cada unidade experimental foi constituida de 4 plantas, contendo 2 plantas úteis por parcela.
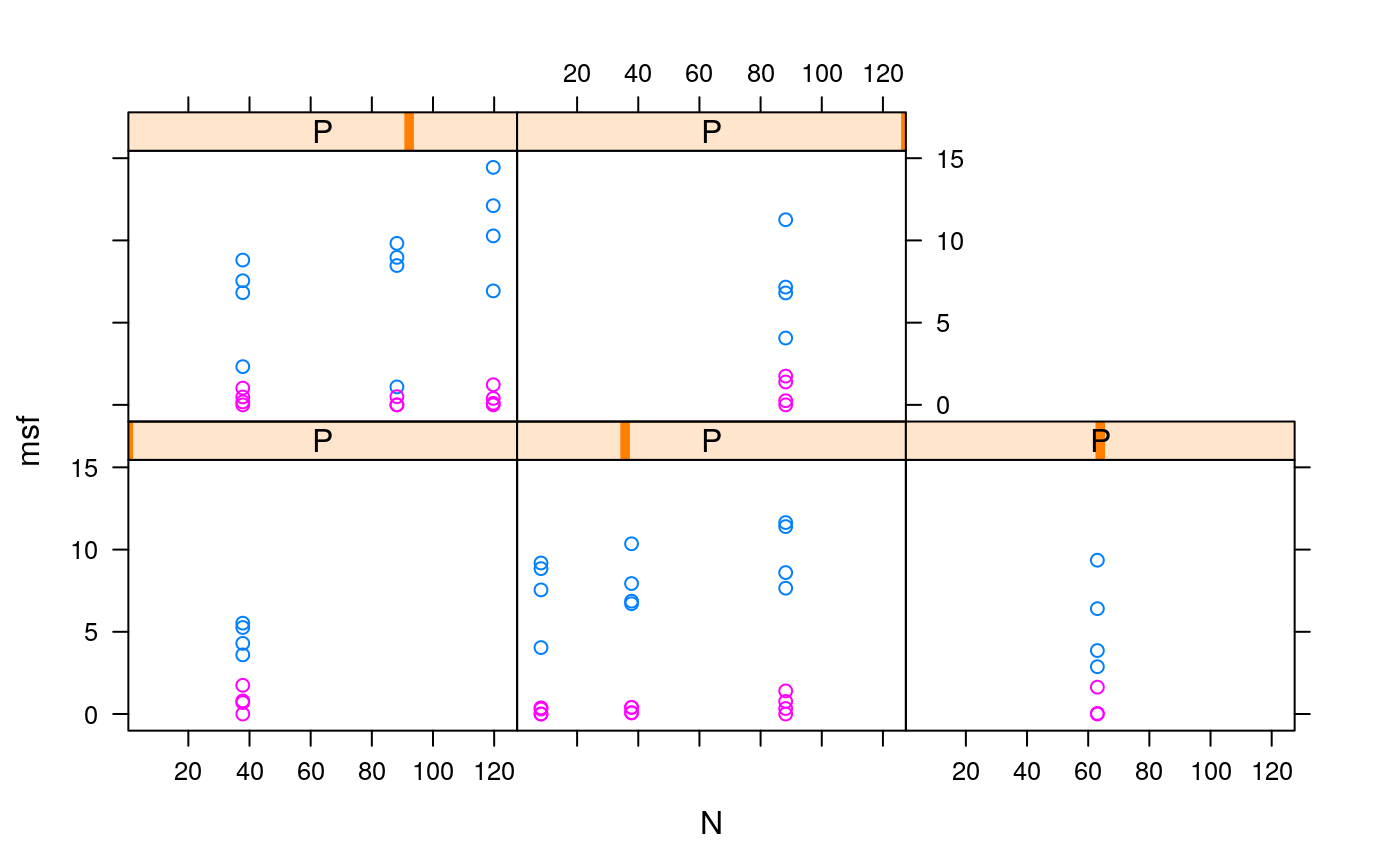
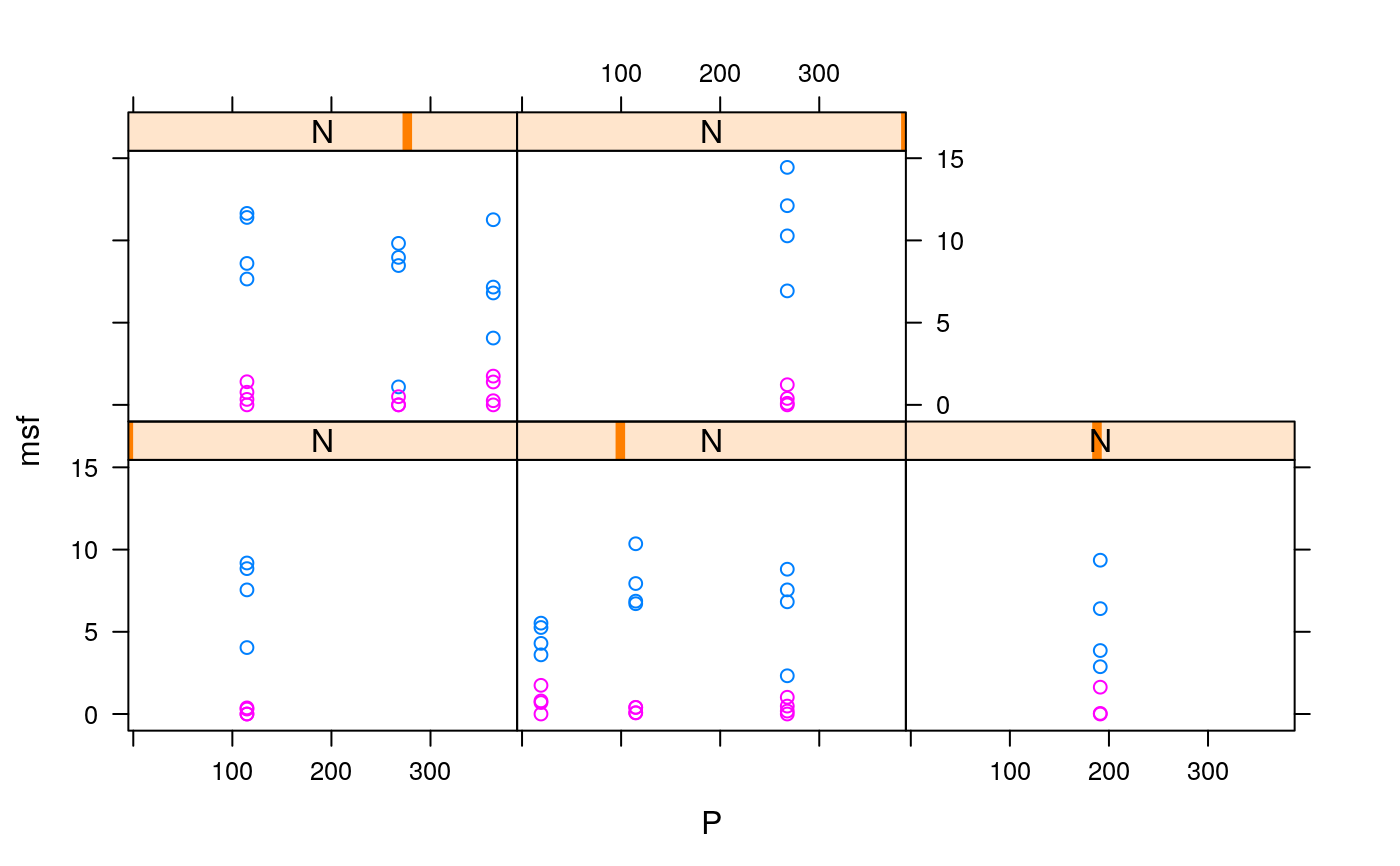
Colheram-se duas plantas em janeiro/2013 (259 dias após o transplantio - DAT) e em junho/2016 (770 DAT), quando as produções foram mensuradas. Posteriormente, determinou-se o teor de nutrientes das folhas e raízes.
Format
Um data.frame com 72 observações e 7 variáveis, em que
epocÉpoca de colheita da carobinha e determinação das variáveis resposta, em dias após o transplantio.
blocFator categórico que indica os blocos do experimento, TODO formado pelas parcelas no mesmo canteiro.
NNível de nitrogênio aplicado na adubação, kg ha\(^{-1}\).
PNível de fósforo aplicado na adubação, kg ha\(^{-1}\).
msfMassa fresca das folhas, g.
KraizTeor de potássio nas raízes, TODO grandeza.
KfolhTeor de potássio nas folhas, TODO grandeza.
Examples
#> 'data.frame': 72 obs. of 7 variables: #> $ epoc : int 259 259 259 259 259 259 259 259 259 259 ... #> $ bloc : Factor w/ 4 levels "I","II","III",..: 1 1 1 1 1 1 1 1 1 2 ... #> $ N : num 8.19 37.8 37.8 37.8 63 ... #> $ P : num 114.8 19.1 114.8 267.7 191.2 ... #> $ msf : num 9.18 4.3 6.86 8.81 9.35 ... #> $ Kraiz: num 0.682 0.633 0.534 0.633 0.534 ... #> $ Kfolh: num 0.434 0.782 0.534 0.633 0.583 ...#> [1] 8.19 37.80 63.00 88.20 119.70#> [1] 19.12322 114.75044 191.23150 267.72402 363.33977#> N P #> 1 8.19 114.75044 #> 2 37.80 19.12322 #> 3 37.80 114.75044 #> 4 37.80 267.72402 #> 5 63.00 191.23150 #> 6 88.20 114.75044 #> 7 88.20 267.72402 #> 8 88.20 363.33977 #> 9 119.70 267.72402#> bloc #> epoc I II III IV #> 259 9 9 9 9 #> 770 9 9 9 9#> P #> N 19.12322488 114.75043597 191.23149716 267.72402086 363.33976944 #> 8.19 0 8 0 0 0 #> 37.8 8 8 0 8 0 #> 63 0 0 8 0 0 #> 88.2 0 8 0 8 8 #> 119.7 0 0 0 8 0# Codificar os níveis de N e P para mesma escala centrada. cod <- function(x) { u <- unique(x) stopifnot(length(u) == 5) u <- sort(u) m <- u[3] d <- diff(u[c(2, 4)])/2 z <- (x - m)/d return(z) } # Criando versões codificadas de N e P. np_carobinha <- transform(np_carobinha, nn = cod(N), pp = cod(P)) cbn <- unique(np_carobinha[, c("nn", "pp")]) round(cbn, 3)#> nn pp #> 1 -2.175 -1.00 #> 2 -1.000 -2.25 #> 3 -1.000 -1.00 #> 4 -1.000 1.00 #> 5 0.000 0.00 #> 6 1.000 -1.00 #> 7 1.000 1.00 #> 8 1.000 2.25 #> 9 2.250 1.00