Capítulo 11 Análise de covariância
11.1 Motivação
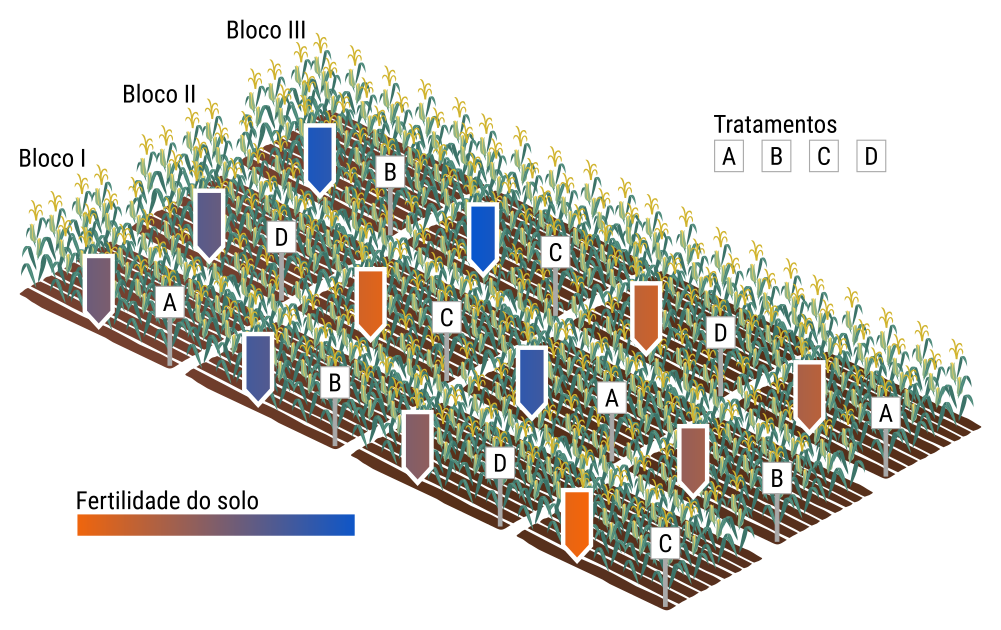
Figura 11.1: Ilustração de um experimento em delineamento de blocos completos casualizados para estudar um fator de 4 níveis com 3 repetições no rendimento de grãos de milho. Um experimento de adubação foi feito antes no local. Por conta disso, as parcelas apresentam nível de fertilidade não uniforme.

Figura 11.2: Esquema de um experimento em delineamento de blocos completos casualizados para estudar o efeito do valor energético da ração no ganho de peso de suínos com 6 repetições. Os blocos acomodam o efeito de grupos contemporâneos, que são animais nascidos no mesmo período e mantidos sob as mesmas condições de instalação e manejo. No entando, dentro de um grupo há variação no peso inicial dos animais.
- Incluir variáveis de controle local contínuas.
- Assumir uma relação funcional.
- Independe dos tratamentos aplicados.
- Independe da variável resposta.
Diferença entre regressora e covariável. Suposições.
Exemplos:
- Acomodar o efeito da variação de stand causado por uma chuva de granizo.
- Acomodar o efeito de variação de fertilidade do solo.
- Acomodar variação de umidade dos grãos.
- De peso inicial dos animais.
- De idade dos indivíduos.
A covariável pode manisfestar efeito e ser medida no começo do experimento (idade, peso inicial, fertilidade). Mas pode ser medida no meio ou quando se manifestar, como uma chuva de granizo que compromete o número planejado de plantas por parcela ou o ataque de alguma praga.
Atenção: o uso de uma covariável de efeito não aditivo com o(s) fator(es) sob investigação pode produzir resultados enganosos.
Suponha que a variação no stand tenha sido causada por uma chuva forte que quebrou o caule das plantas no florescimento da cultura levando-as a morte. Espera-se o efeito causado por esse evento (chuva) seja independente do nível/efeito dos tratamentos. Ou seja, assumimos que os cultivares são igualmente prejudicados pela chuva. Porém, se a resistência do caule depender da cultivar, mesmo que a exposição à chuva tenha sido a mesma nas cultivares, o efeito da chuva no stand será diferente. Em consequência, as cultivares mais resistentes ao tombamento terão produzido mais por terem mais plantas na parcela já que são mais resistentes ao tombamento. Então usar ou não stand como termo para modelar a produtividade poderá favorescer ou não cultivares resistentes ao tombamento.
Em muitos contextos, o nome offset é usando para a covariável. IMPROVE.
rm(list = objects())
library(car) # linearHypothesis(), Anova(), ResidualPlots().
library(emmeans) # emmeans().
library(multcomp) # glht().
library(tidyverse) # Manipulação de dados.
library(labestData) # Tabelas de dados.
source("mpaer_functions.R")11.2 Competição de cultivares de feijão em Zimmermann
O exemplo disponível em ZIMMERMANN (2004) é de um experimento de competição de cultivares de feijão. O experimento foi realizado no delineamento de blocos casualizados completos e avaliou a produtividade de 7 cultivares com 4 repetições. Ao final do experimento, registrou-se o número de plantas na área útil da parcela, variável que é chamada de stand. As plantas da área útil foram, então, colhidas para determinação da produtividade. Havendo mais plantas na área útil, teremos maior produção.
11.2.1 Análise exploratória
Como preconizado, começamos pela análise exploratória dos dados. Com ela fazemos a inspeção de conformidade que i) pode revelar vestígios de problema nos dados que precisarão de reparo, ii) permite antecipar a forma dos efeitos dos tratamentos e covariáveis, iii) permite verificar se os pressupostos do modelo indicado para o experimento são razoáveis.
Nos gráficos a seguir serão vistas as relações entre a resposta e as cultivares, entre a resposta e a covariável stand.
# Abre a documentação.
if (interactive()) {
help(ZimmermannTb14.3, help_type = "html")
}
# Nomes curtos dão mais agilidade.
tb <- ZimmermannTb14.3
tb <- tb %>%
mutate(cult = factor(str_replace_all(cult, "\\W", "")))
# Estrutura da tabela de dados.
str(tb)## 'data.frame': 28 obs. of 4 variables:
## $ bloc : Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 2 2 2 ...
## $ cult : Factor w/ 7 levels "CNFP8018","CNFP8019",..: 1 2 3 4 5 6 7 1 2 3 ...
## $ stand: int 95 124 106 92 101 98 103 103 114 116 ...
## $ prod : num 1588 2012 1975 1838 1825 ...# Resumo descritivo.
summary(tb)## bloc cult stand prod
## 1:7 CNFP8018 :4 Min. : 77.0 Min. :1375
## 2:7 CNFP8019 :4 1st Qu.: 92.0 1st Qu.:1856
## 3:7 CNFP8020 :4 Median :101.0 Median :2056
## 4:7 CNFP8021 :4 Mean :100.7 Mean :2007
## CNFP8022 :4 3rd Qu.:108.8 3rd Qu.:2138
## CNFP8023 :4 Max. :124.0 Max. :2588
## GBrilhante:4# Diagrama de dispersão priorizando o efeito de cultivar.
ggplot(data = tb,
mapping = aes(x = reorder(cult, prod),
y = prod,
color = bloc)) +
geom_point(mapping = aes(size = stand)) +
geom_line(mapping = aes(group = bloc),
show.legend = FALSE)
# Diagrama de dispersão priorizando o efeito de stand.
ggplot(data = tb,
mapping = aes(x = stand, y = prod)) +
facet_wrap(facets = ~cult) +
geom_point() +
geom_rug(sides = "b",
mapping = aes(x = mean(stand)),
color = "black") +
geom_rug(data = aggregate(cbind(stand, prod) ~ cult, tb, mean),
sides = "b",
mapping = aes(x = stand),
color = "orange") +
geom_smooth(se = TRUE, method = "lm", color = "orange") +
geom_smooth(data = select(tb, -cult),
mapping = aes(x = stand, y = prod),
size = 0.7, color = "gray",
inherit.aes = FALSE,
se = FALSE, method = "lm")
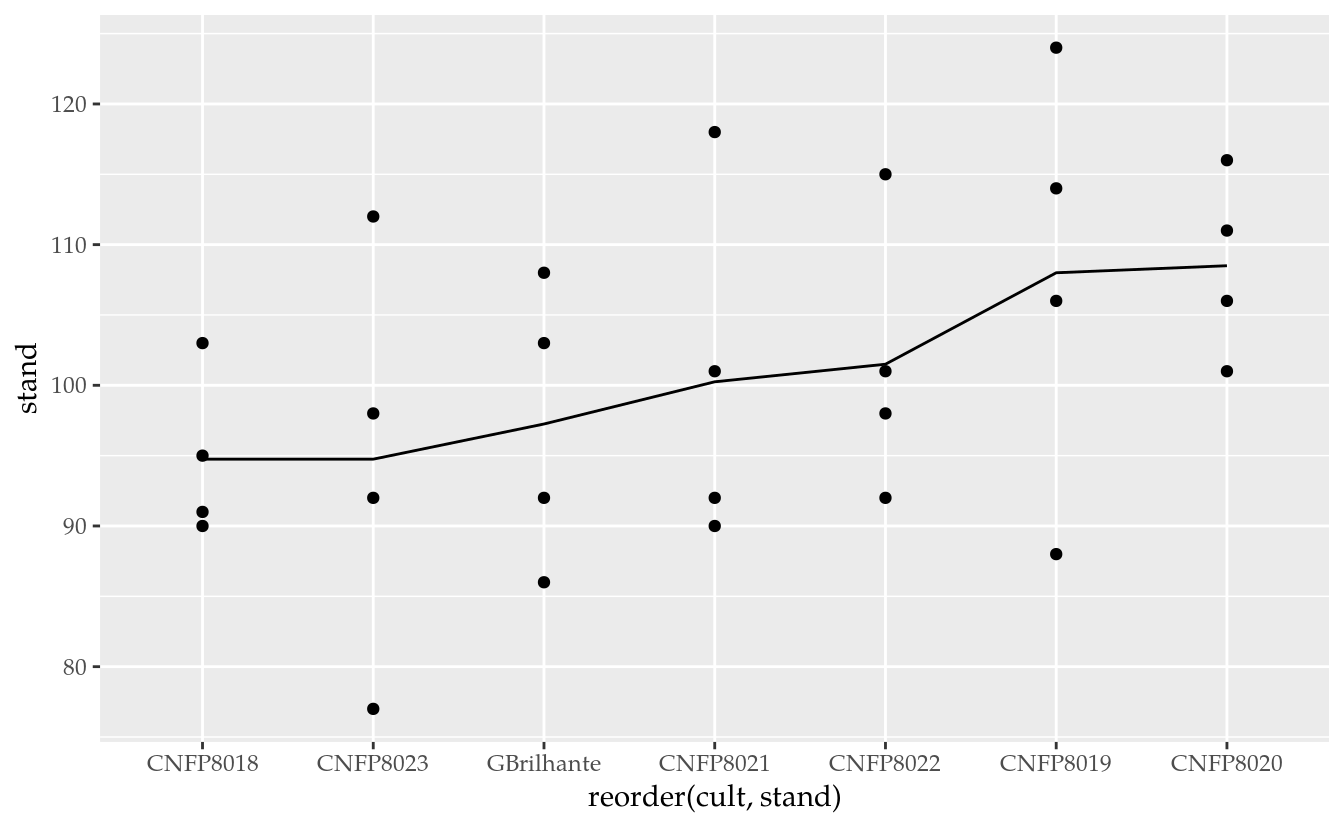
# Gráfico para verificar o efeito de cultivar no stand.
ggplot(data = tb,
mapping = aes(x = reorder(cult, stand),
y = stand)) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")
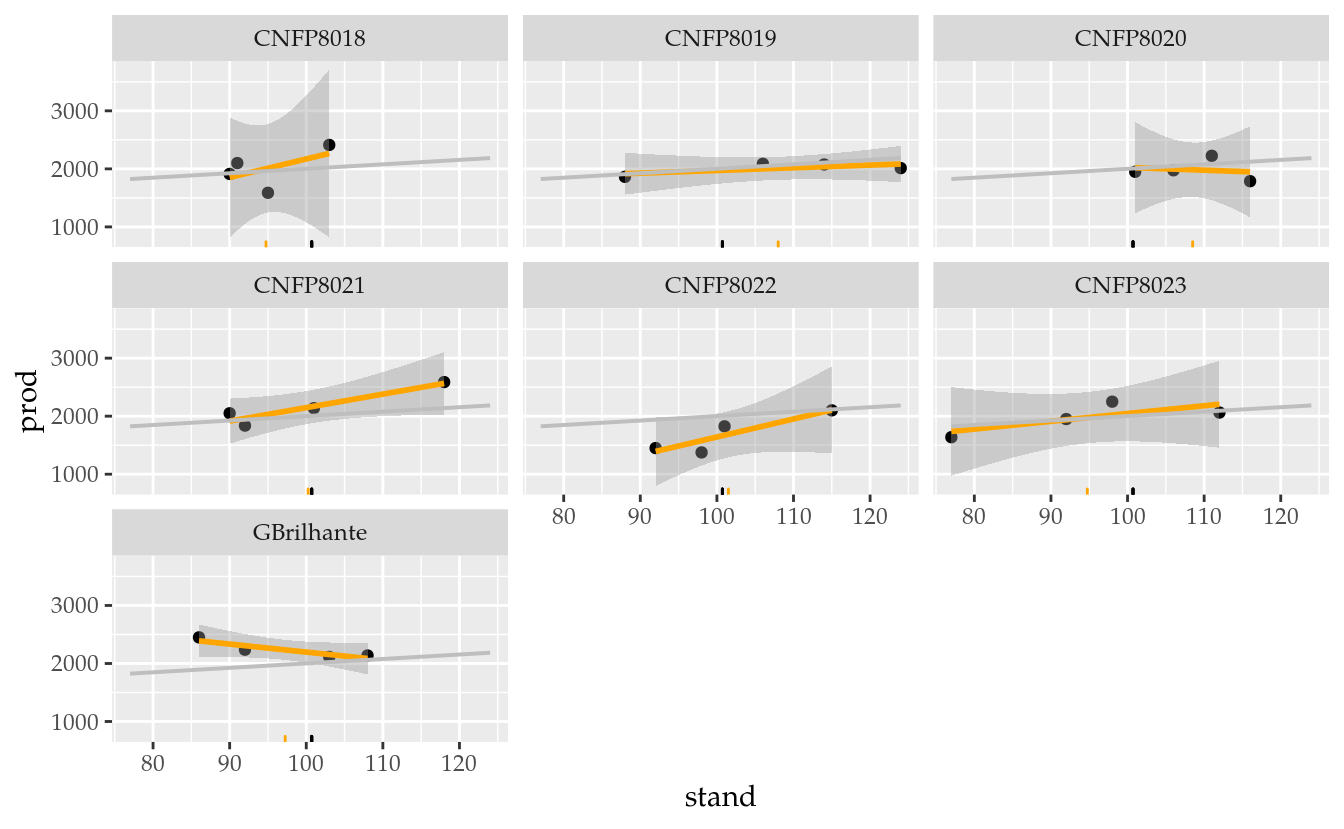
O primeiro gráfico indica que pode efeito de cultivar na produtividade. As maiores produtividades parecem ser de unidades experimentais com maior stand. O efeito de bloco não deve ser muito pronunciado.
O segundo gráfico ilustra melhor o efeito de stand na produtividade indicando que a contribuição é linear e positiva na maioria das cultivares. É importante destacar que, embora será um termo do modelo, o stand não é um fator com níveis controlados pelo pesquisador. O gráfico mostra que o conjunto de valores de stand em cada cultivar é diferente. Isso terá implicação na obtenção das médias marginais estimadas e seu erro padrão. As marcas preta e laranja no eixo x são o stand médio do experimento e o stand por cultivar. A linha inclinada em cinza é o ajuste a produtividade em função do stand (ignorando efeito de cultivar e bloco). Apensar de termos ajustado funções, são apenas para auxiliar a análise visual. Portanto, não considere tais impressões como definitivas.
O último gráfico é se suma importância. Com ele julgamos a hipótese de stand depender de cultivar. Se o stand for uma variável influenciada pela cultivar, usá-la na análise de covariância poderá produzir resultados enganosos se interpretados imediatamente.
Essa análise exploratória não parece indicar existência de observações influentes ou qualquer outro tipo de ameaça em relação aos pressupostos do modelo. Portanto, presseguimos sem modificação nos dados.
11.2.2 Ajuste do modelo
A variável resposta é a produtividade que é medida em uma escala contínua. Dessa forma, podemos assumir distribuição Gaussiana. Bloco e cultivar são fatores qualitativos e seus efeitos serão acomodados por um conjunto \(k - 1\) parâmetros, em que \(k\) é o número de níveis de cada fator.
O modelo considerando apenas o efeito de bloco e cultivar é \[ \begin{aligned} Y_{ij} &= \text{Normal}(\mu_{ij}, \sigma^2) \\ \mu_{ij} &= \mu + \alpha_{i} + \tau_{j}\\ \sigma^2 &\propto 1 \end{aligned} \] em que
- \(Y_{ij}\) é o valor observado da variável resposta na unidade experimental \(ij\) que tem distribuição normal com média \(\mu_{ij}\) e variância \(\sigma^2\). A média, portanto, depende do bloco e cultivar e a variância não depende de termo algum (suposição de homocedasticidade).
- \(\mu\) é uma constante que pertence a todas as observações.
- \(\alpha_{i}\) é o efeito do bloco \(i\).
- \(\tau_{j}\) é o efeito da cultivar \(j\).
É razoável assumir que o efeito de stand seja bem descrito por alguma função \(f(x, \beta)\), em que \(x\) é o stand e \(\beta\) os parâmetros dessa função \(f\). Dessa forma, assuminfo que o efeito de stand aditivo aos demais, a expressão do modelo é \[ \begin{aligned} Y_{ij} &= \text{Normal}(\mu_{ij}, \sigma^2) \\ \mu_{ij} &= \mu + \alpha_{i} + \tau_{j} + f(x_{ij}, \beta)\\ \sigma^2 &\propto 1. \end{aligned} \]
Como a análise exploratória indicou, o efeito de stand na produtividade é linear, então a função \(f\) assume uma forma simples e ao incluí-la no modelo tem-se \[ \begin{aligned} \mu_{ij} &= \mu + \alpha_{i} + \tau_{j} + \beta_{0} + \beta_{1} x_{ij}\\ &= (\mu + \beta_{0}) + \alpha_{i} + \tau_{j} + \beta_{1} x_{ij}\\ &= \mu^{*} + \alpha_{i} + \tau_{j} + \beta_{1} x_{ij}. \end{aligned} \]
Após o ajuste do modelo serão verificadas as suposições feitas. Pode ser que seja necessário mudar a expressão de \(f\) para incluir um termo de segunda ordem ou declarar um modelo não aditivo, por exemplo.
No bloco a seguir dois modelos são ajustados. O último contém o efeito de stand, além do efeito de bloco e cultivar já presentes no precedente. É feita análise dos resíduos e exibido o quadro de análise de variância para julgarmos a relevância dos termos do modelo.
# Ajuste do modelo sem declarar `stand`.
m0 <- lm(prod ~ bloc + cult, data = tb)
# Modelo que contém o anterior e mais o efeito linear de `stand`.
m1 <- lm(prod ~ bloc + stand + cult, data = tb)
# Análise de resíduos.
par(mfrow = c(2, 2)); plot(m1); layout(1)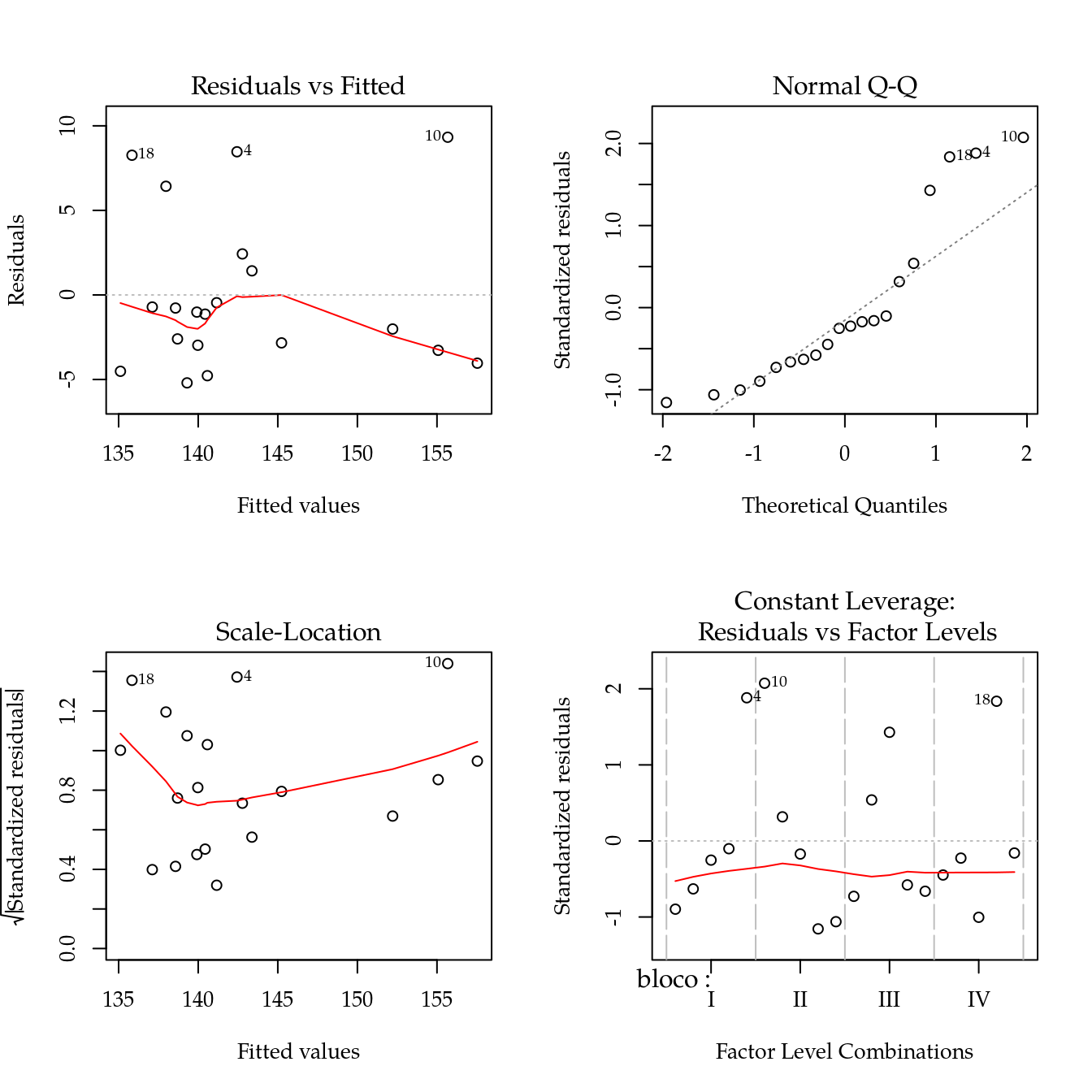
# Resíduos parciais.
# residualPlots(m1)
# Verifica a relevância do(s) termo(s) extra(s), o efeito de `stand`,
# usando a estatística F da redução na soma de quadrados residual entre
# modelos encaixados.
anova(m0, m1)## Analysis of Variance Table
##
## Model 1: prod ~ bloc + cult
## Model 2: prod ~ bloc + stand + cult
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 18 1277757
## 2 17 929430 1 348326 6.3712 0.02184 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Quadro de análise de variância com testes para hipóteses sob o efeito
# marginal de cada termo.
Anova(m1)## Anova Table (Type II tests)
##
## Response: prod
## Sum Sq Df F value Pr(>F)
## bloc 116917 3 0.7128 0.55769
## stand 348326 1 6.3712 0.02184 *
## cult 899421 6 2.7419 0.04741 *
## Residuals 929430 17
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1O teste F para a redução na soma de quadrados residual aponta que existe efeito de stand. O quadro de análise de variância apresenta a mesma estatística F para o teste do efeito do stand. Porém, em modelos mais gerais, como modelos lineares generalizados, por exemplo, o teste baseado da redução da deviance e o teste F do quadro de deviance podem diferir.
O quadro de anova também indica efeito de cultivar. Nas subseções seguintes será feito estudo mais detalhado do efeito de cultivar com uso de comparações múltiplas de médias.
O efeito de stand é representado pelo coeficiente de taxa do modelo (\(\beta\)). Por ser uma taxa, a unidade de medida é kg ha\(^{-1}\) planta\(^{-1}\). Dessa forma, a interpretamos que cada planta a mais na área útil da parcela aumenta em 14.9 kg ha\(^{-1}\) a produtividade. O stand médio do experimento foi de 100.7 plantas.
# Estimativa pontual e intervalar para o efeito de `stand`.
cbind(est = coef(m1)["stand"], confint(m1, parm = "stand"))## est 2.5 % 97.5 %
## stand 14.90935 2.447168 27.37153# Stand médio do experimento.
mean(tb$stand)## [1] 100.714311.2.3 Médias marginais e comparações múltiplas
Neste experimento, as médias marginais amostrais não são estimadores apropriados das médias marginais populacionais. Isso porque o efeito de stand não é ortogonal ao efeito dos demais termos do modelo tal como acontece com outros delineamentos em que está presente alguma covariável de níveis não controlados.
O código abaixo ilustra essa afirmação. As matrizes de projeção do espaço coluna dos termos bloco, cultivar e stand são obtidas. O produto matricial entre matrizes de projeção ortogonais é uma matriz de zeros. Caso contrário, ao menos um elemento serão diferente de zero. Verifica-se que apenas os efeitos de bloco e cultivar são ortogonais.
#-----------------------------------------------------------------------
# Matrizes de delineamento para cada termo separado.
X_mu <- model.matrix(~1, data = tb)
X_bloc <- model.matrix(~bloc, data = tb)
X_cult <- model.matrix(~cult, data = tb)
X_stand <- model.matrix(~stand, data = tb)
# Matrizes de projeção.
P_bloc <- (proj(X_bloc) - proj(X_mu))
P_cult <- (proj(X_cult) - proj(X_mu))
P_stand <- (proj(X_stand) - proj(X_mu))
# Verifica se são espaços ortogonais.
all(round(P_bloc %*% P_cult, digits = 3) == 0) # São ortogonais.## [1] TRUEall(round(P_bloc %*% P_stand, digits = 3) == 0) # Não são.## [1] FALSEall(round(P_cult %*% P_stand, digits = 3) == 0) # Não são.## [1] FALSENos fragmentos de código abaixo calculamos a média amostral e as médias marginais com os dois modelos definidos: aquele que não incluiu efeito de stand (m0) e o modelo que incluiu (m1).
# Média marginal amostral de produtividade por cultivar.
smm <- tb %>%
group_by(cult) %>%
summarise(smean = mean(prod)) %>%
as.data.frame()
# Média marginal estimada por cultivar com cada modelo.
emm0 <- emmeans(m0, specs = ~cult)
emm1 <- emmeans(m1, specs = ~cult)
# Coloca na mesma tabela para comparação.
v <- c("emmean", "SE")
bind_cols(smm,
as.data.frame(emm0)[, v] %>% rename_all(paste0, "_m0"),
as.data.frame(emm1)[, v] %>% rename_all(paste0, "_m1")) %>%
arrange(smean)## cult smean emmean_m0 SE_m0 emmean_m1 SE_m1
## 1 CNFP8022 1687.500 1687.500 133.2164 1675.786 117.0027
## 2 CNFP8023 1975.000 1975.000 133.2164 2063.924 122.1033
## 3 CNFP8020 1984.375 1984.375 133.2164 1868.295 125.6305
## 4 CNFP8018 2003.125 2003.125 133.2164 2092.049 122.1033
## 5 CNFP8019 2009.375 2009.375 133.2164 1900.750 124.5797
## 6 CNFP8021 2153.125 2153.125 133.2164 2160.047 116.9428
## 7 GBrilhante 2234.375 2234.375 133.2164 2286.025 118.6879Pela inspeção da tabela acima pode-se ver que:
- As médias amostrais marginais são iguais às médias marginais do modelo
m0. Sem a covariávelstand, a matriz de delineamento fica balanceada e com termos de efeito ortogonal, assim as médias marginais amostrais coincidem com as médias marginais estimadas. - O erro padrão das médias marginais é o mesmo para todas as cultivares no modelo
m0. Neste modelo, o erro padrão da média é função apenas do número de repetições que foi o mesmo para cultivares. - O erro padrão das médias marginais muda com a cultivar no modelo
m1. Neste modelo o erro padrão depende do conjunto de valores de stand observados. Os erros padrões seriam iguais se todas as cultivares de um mesmo bloco apresentassem o mesmo stand. Algo que na prática só seria razoável se stand fosse um fator controlável que o pesquisador pudesse fixar os níveis para observar a resposta. - As médias marginais diferem entre os modelos
m0em1. Muda inclusive o ordenamento das médias. Esse resultado justifica a preocupação colocada anteriormente com a contextualização da chuva. As médias marginais foram corrigidas para o efeito de stand. Se isso deve ser feito não é uma questão estatística mas prática, da natureza do fenômeno e das hipóteses.
Atenção: o modelo que assumimos i) não tem função de ligação, ii) não tem função de variância (suposição de variância constante), ii) não tem efeitos aleatórios e iv) as observações são independentes. Logo, para modelos em que algum desse itens for modificado, as conclusões podem não ser válidas.
Em resumo, as diferenças acima comentadas são resultado da inclusão do stand como termo do modelo. As médias marginais são diferentes por que usam o mesmo valor de referência para o stand para serem calculadas. O erro padrão variável também é função da disposição dos pontos de suporte no stand.
# Diferença entre o stand médio geral e por cultivar.
tb %>%
mutate(m_geral = mean(stand)) %>%
group_by(cult) %>%
summarise(m_geral = m_geral[1],
m_cult = mean(stand),
diff = m_cult - m_geral) %>%
arrange(m_cult)## # A tibble: 7 x 4
## cult m_geral m_cult diff
## <fct> <dbl> <dbl> <dbl>
## 1 CNFP8018 101. 94.8 -5.96
## 2 CNFP8023 101. 94.8 -5.96
## 3 GBrilhante 101. 97.2 -3.46
## 4 CNFP8021 101. 100. -0.464
## 5 CNFP8022 101. 102. 0.786
## 6 CNFP8019 101. 108 7.29
## 7 CNFP8020 101. 108. 7.79# O stand médio é o valor usado por padrão para obter as médias
# marginais.
attr(emm1, "linfct") %>%
t() %>%
print.table(zero.print = ".", digits = 2)## [,1] [,2] [,3] [,4] [,5] [,6] [,7]
## (Intercept) 1.00 1.00 1.00 1.00 1.00 1.00 1.00
## bloc2 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## bloc3 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## bloc4 0.25 0.25 0.25 0.25 0.25 0.25 0.25
## stand 100.71 100.71 100.71 100.71 100.71 100.71 100.71
## cultCNFP8019 . 1.00 . . . . .
## cultCNFP8020 . . 1.00 . . . .
## cultCNFP8021 . . . 1.00 . . .
## cultCNFP8022 . . . . 1.00 . .
## cultCNFP8023 . . . . . 1.00 .
## cultGBrilhante . . . . . . 1.00As médias marginais podem ser obtidas com outros valores de referência para condicionar a estimação. Pode fixar outro valor de stand ou outra função a ser aplicada na amostra, como a mediana. Também pode-se fixar outro nível de bloco, se fizer sentido.
# É possível usar por outro valor, i.e. 120 plantas.
emm <- emmeans(m1, specs = ~cult, at = list(stand = 120))
emm <- emmeans(m1, specs = ~cult, at = list(stand = 120, bloc = "3"))
emm <- emmeans(m1, specs = ~cult, cov.reduce = list(stand = median))
as.data.frame(emm)
attr(emm, "linfct")Para estudar o efeito de cultivar em mais detalhes, serão feitas as comparações múltiplas de médias marginais. O código abaixo obtém a matriz com coeficientes para calcular as médias marginais que é extraído do atributo retornado pela emmeans(). A matriz de contrastes é construída e passada para a glht(). Métodos são chamados para obter o intevalor de confiança para a média e adicionar a representação compacta de letras. Por fim, um gráfico de segmentos é feito para apreciar os resultados.
# Pontos experimentais e matriz para obter as médias marginais.
grid <- attr(emm1, "grid")
L <- attr(emm1, "linfct")
rownames(L) <- grid$cult# Contrastes par a par entre médias marginais.
K <- all_pairwise(L, collapse = " - ")
# Constraste de médias (contrasts of means).
com <- glht(model = m1, linfct = K)
# com
# Quando `mcp(. = "Tukey")` é usado, tem-se a representação com letras
# para os contrastes par a par. Só coincide com o uso de `K` porque
# todos os efeitos são aditivos.
com <- summary(glht(model = m1,
linfct = mcp(cult = "Tukey")),
test = adjusted(type = "fdr"))
com
# Resumo compacto de letras.
a <- cld(com)
# O intervalo de confiança é o mesmo returnado pela `emmeans()`.
b <- confint(glht(m1, linfct = L), calpha = univariate_calpha())
# Reúne tudo em uma tabela.
results <- cbind(grid, b$confint, cld = a$mcletters$Letters) %>%
rename("fit" = "Estimate")
results# A função `wzRfun::apmc()` contém as etapas acima:
# glht -> summary -> cld -> confint.
results <- wzRfun::apmc(X = L, model = m1, focus = "cult", test = "fdr")
# Exibição dos resultados.
ggplot(data = results,
mapping = aes(x = fit, y = reorder(cult, fit, mean))) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.0f %s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = sprintf("Produtividade (para stand de %0.1f plantas)",
L[1, "stand"]),
y = "Cultivares")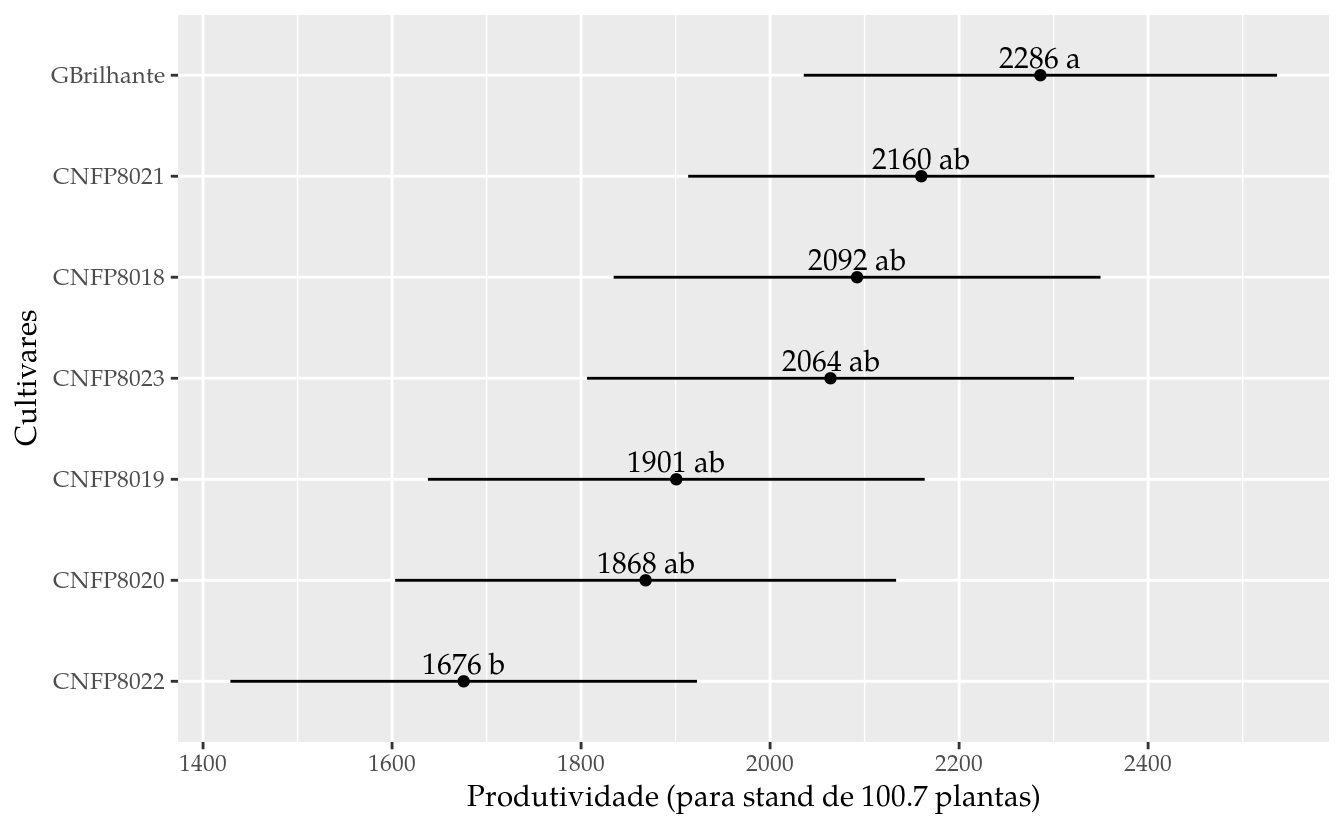
O gráfico indica que apenas o contraste entre as cultivares da extremidade é diferente de zero. Nenhum contraste envolvendo o restante das cultivares é significativo.
As estimativas intervalares de produtividade utilizaram o stand médio do experimento como referência. O erro padrão da média marginal aumenta quando o valor do stand usado para condicionar afasta-se do stand médio do experimento. O interessante é, no modelo Gaussiano, a incerteza na estimação do efeito de stand desaparece pela subtração feita na obtenção do contraste. Em modelos mais gerais, por ter função de ligação e função de variância, por exemplo, essa regra pode não se manter.
No bloco abaixo apenas as cultivares mais contrastantes tem a produtividade predita para diferentes valores de stand.
# Obtém as cultivares mais contrastantes.
cult_ext <- results %>%
arrange(fit) %>%
slice(c(1, n())) %>%
pull(cult)
# Cria sequência de valores de `stand`.
stand_seq <- seq(75, 125, by = 5)
# Obtém as médias marginais para esse grid de pontos experimentais.
emm_rg <- emmeans(m1,
specs = ~cult + stand,
at = list(stand = stand_seq,
cult = cult_ext))
grid <- as.data.frame(emm_rg)
head(grid)## cult stand emmean SE df lower.CL upper.CL
## 1 CNFP8022 75 1292.402 195.3701 17 880.2074 1704.597
## 2 GBrilhante 75 1902.642 175.8998 17 1531.5259 2273.758
## 3 CNFP8022 80 1366.949 172.6150 17 1002.7633 1731.135
## 4 GBrilhante 80 1977.189 155.0806 17 1649.9972 2304.380
## 5 CNFP8022 85 1441.496 152.2066 17 1120.3679 1762.624
## 6 GBrilhante 85 2051.736 137.4909 17 1761.6550 2341.816# Gráfico apenas para as cultivares mais contrastantes.
pd <- position_dodge(1)
ggplot(data = grid,
mapping = aes(x = stand, y = emmean,
color = cult, group = cult)) +
geom_point(position = pd) +
geom_line(position = pd) +
geom_errorbar(mapping = aes(ymin = lower.CL, ymax = upper.CL),
position = pd,
width = 1)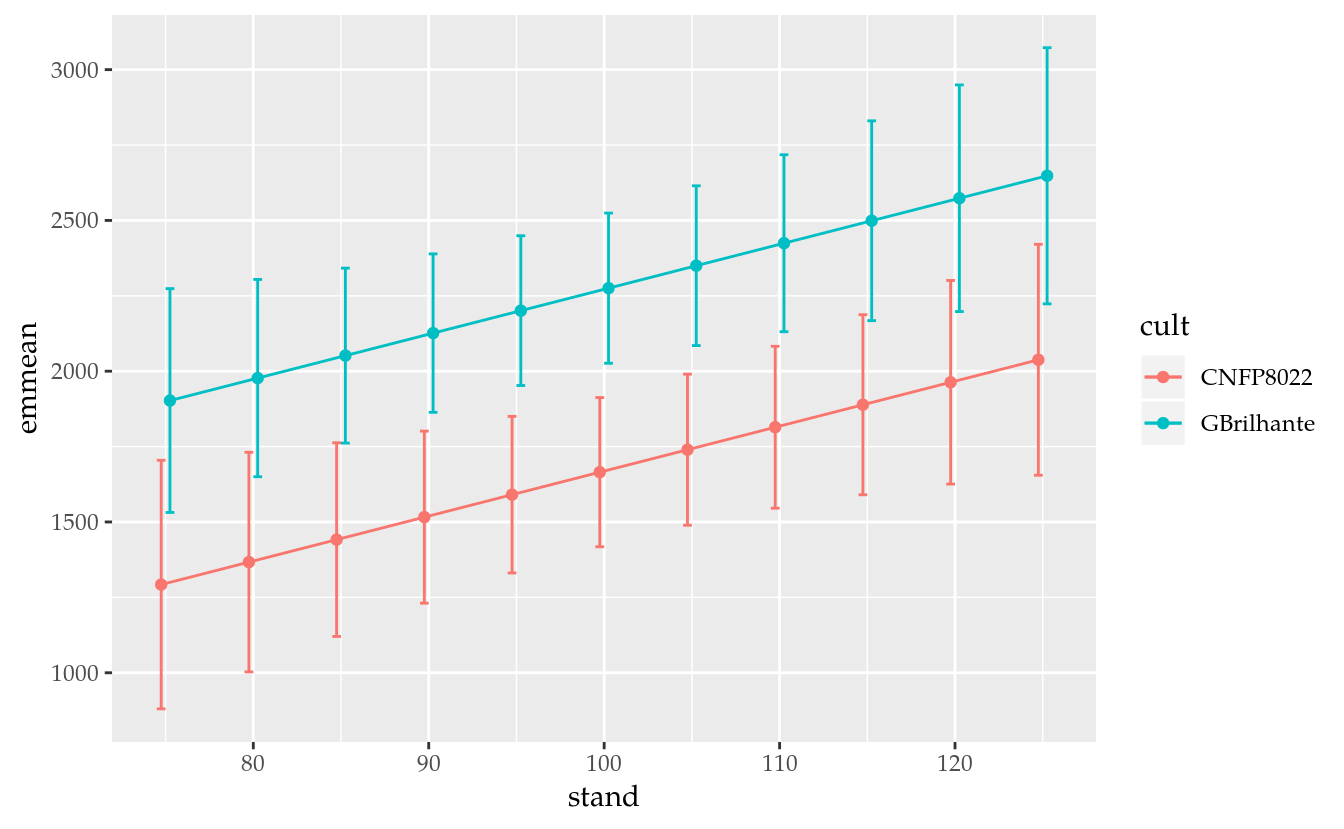
# Avalia o contraste entre médias para alguns valores de `stand`.
L <- attr(emm_rg, "linfct")
# Matriz para o contrastes entre cultivares em cada `stand`.
K <- by(L,
INDICES = grid$stand,
FUN = function(x) x[2, ] - x[1, ]) %>%
do.call(what = rbind) %>%
as.matrix()
K[1, , drop = FALSE]## (Intercept) bloc2 bloc3 bloc4 stand cultCNFP8019 cultCNFP8020
## 75 0 0 0 0 0 0 0
## cultCNFP8021 cultCNFP8022 cultCNFP8023 cultGBrilhante
## 75 0 -1 0 1# Resultados.
summary(glht(m1, linfct = K), test = adjusted(type = "none"))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = prod ~ bloc + stand + cult, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 75 == 0 610.2 167.2 3.649 0.00199 **
## 80 == 0 610.2 167.2 3.649 0.00199 **
## 85 == 0 610.2 167.2 3.649 0.00199 **
## 90 == 0 610.2 167.2 3.649 0.00199 **
## 95 == 0 610.2 167.2 3.649 0.00199 **
## 100 == 0 610.2 167.2 3.649 0.00199 **
## 105 == 0 610.2 167.2 3.649 0.00199 **
## 110 == 0 610.2 167.2 3.649 0.00199 **
## 115 == 0 610.2 167.2 3.649 0.00199 **
## 120 == 0 610.2 167.2 3.649 0.00199 **
## 125 == 0 610.2 167.2 3.649 0.00199 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- none method)A estimativa da diferença em produtividade das duas cultivares é a mesma. Visto que o feito de stand é aditivo (e a função de ligação é a indentidade) as retas são paralelas. Os erros padrões dos contrastes foram os mesmos também. É que ao construir a matriz de contraste das médias marginais, a incerteza sobre o efeito de stand na média marginal de uma cultivar subtrai o mesmo em outra. Com isso, a incerteza sobre o efeito de stand (e também sobre o efeito de bloco) não aparece no contraste.
11.2.4 Modelos alternativos
E se o efeito de primeira ordem não for o correto? Um modelo mais flexível pode ser ajustado. O modelo com termo quadrático do stand é ajustado no bloco a seguir. O teste da soma de quadrados extra pode ser aplicado para avaliar a contribuição do parâmetro adicional.
# Modelo com termo quadrático de `stand`.
m2 <- lm(prod ~ bloc + stand + I(stand^2) + cult, data = tb)
anova(m1, m2)## Analysis of Variance Table
##
## Model 1: prod ~ bloc + stand + cult
## Model 2: prod ~ bloc + stand + I(stand^2) + cult
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 17 929430
## 2 16 914210 1 15220 0.2664 0.6128Pelo teste da redução na soma de quadrados residual, não há necessidade de inclusão de um termo quadrático para acomomodar o efeito de stand na produtividade.
E se a suposição de aditividade não for razoável? Neste caso um modelo mais geral pode ser especificado. Por exemplo, pode-se declarar o termo de interação entre cultivar o termo linear de stand. O teste da soma de quadrados extra pode ser aplicado para a avaliar a nulinidade dos parâmetros adicionais.
# Modelo com inclinações por cultivar (efeito multiplicativo).
m3 <- lm(prod ~ bloc + stand * cult, data = tb)
anova(m1, m3)## Analysis of Variance Table
##
## Model 1: prod ~ bloc + stand + cult
## Model 2: prod ~ bloc + stand * cult
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 17 929430
## 2 11 488368 6 441062 1.6557 0.2217Pelo teste da redução na soma de quadrados residual, não há necessidade de um modelo que seja de efeitos multiplicativos entre cultivar e stand.
Na seção seguinte será feita a análise de covariância para outros dados com a finalidade de fixar melhor os procedimentos e enfatizar aspectos práticos.
11.3 Código com dados adicionais
Abaixo tem-se os códigos. Os resultados não são exibidos e não é feita discussão. Fica a cargo do leitor interessado proceder com a execução e entendimento da análise.
tb <- RamalhoTb13.6
str(tb)
summary(tb)
ggplot(data = tb,
mapping = aes(x = cult, y = prod, color = bloc)) +
geom_point(mapping = aes(size = teor)) +
geom_line(mapping = aes(group = bloc),
show.legend = FALSE)
ggplot(data = tb,
mapping = aes(x = teor, y = prod)) +
facet_wrap(facets = ~cult) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")
ggplot(data = tb,
mapping = aes(x = reorder(cult, teor),
y = teor)) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")
m1 <- lm(prod ~ bloc + teor + cult, data = tb)
par(mfrow = c(2, 2)); plot(m1); layout(1)
residualPlots(m1)
Anova(m1)
coef(m1)["teor"]
emm1 <- emmeans(m1, specs = ~cult)
as.data.frame(emm1)
grid <- attr(emm1, "grid")
L <- attr(emm1, "linfct")
rownames(L) <- grid$cult
results <- wzRfun::apmc(X = L, model = m1, focus = "cult", test = "fdr")
ggplot(data = results,
mapping = aes(x = fit, y = reorder(cult, fit, mean))) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = "Produtividade",
y = "Cultivares")tb <- BanzattoQd9.2.1
str(tb)
summary(tb)
ggplot(data = tb,
mapping = aes(x = varied, y = prod, color = bloco)) +
geom_point(mapping = aes(size = nppp)) +
geom_line(mapping = aes(group = bloco),
show.legend = FALSE)
ggplot(data = tb,
mapping = aes(x = nppp, y = prod)) +
facet_wrap(facets = ~varied) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")
ggplot(data = tb,
mapping = aes(x = reorder(varied, nppp),
y = nppp)) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")
m1 <- lm(prod ~ bloco + nppp + varied, data = tb)
par(mfrow = c(2, 2)); plot(m1); layout(1)
residualPlots(m1)
Anova(m1)
coef(m1)["nppp"]
emm1 <- emmeans(m1, specs = ~varied)
as.data.frame(emm1)
grid <- attr(emm1, "grid")
L <- attr(emm1, "linfct")
rownames(L) <- grid$varied
results <- wzRfun::apmc(X = L, model = m1,
focus = "varied", test = "fdr")
ggplot(data = results,
mapping = aes(x = fit, y = reorder(varied, fit, mean))) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = "Produtividade",
y = "Variedade")11.4 ANCOVA em experimento fatorial
A análise de covariância para um experimento com estrutura fatorial difere da anterior apenas TODO.
Os dados usados nessa seção são de um experimento de nutrição de suínos realizado em arranjo fatorial completo 3 \(\times\) 3. Foram estudados 3 níveis de energia da dieta (baixo, médio e alto) e 3 níveis do fator denominado sexo com níveis fêmea, macho com castração física e macho com castração hormonal, com 8 animais por ponto experimental. O experimento mediu diversas variáveis resposta, como ganho de peso, rendimento de carcaça, conversão alimentar, etc. Os dados foram cedidos para fins didáticos e o seu uso requer expresso consentimento daqueles que os produziram. A documentação dos dados está nas primeiras linhas do arquivo na forma de comentário.
O peso inicial dos animais (kg), antes da instalação do experimento, é conhecido bem como a idade (dias). Essas variáveis podem explicar parte da variação observada nas variáveis resposta, e por essa razão foram medidas. Então serão usadas na análise de covariância ilustrada a seguir.
11.4.1 Importação dos dados
Os dados são importados da web. Após importação, os níveis dos fatores são devidamente ordenados. Todas as combinações de sexo e dieta estão presentes, perfazendo 9 pontos experimentais.
# Lendo arquivos de dados.
url <- "http://leg.ufpr.br/~walmes/data/castracao.txt"
tb <- read_tsv(url, comment = "#")## Parsed with column specification:
## cols(
## .default = col_double(),
## sexo = col_character()
## )## See spec(...) for full column specifications.attr(tb, "spec") <- NULL# Converte variáveis para fator.
tb <- tb %>%
mutate(sexo = factor(sexo, labels = c("Fem", "MCastr", "MImuno")),
dieta = factor(ener, labels = c("Baixo", "Medio", "Alto")))
# Descrição numérica das variáveis.
summary(tb[, c("sexo", "dieta", "pi", "id")])## sexo dieta pi id
## Fem :24 Baixo:24 Min. :87.30 Min. :124.0
## MCastr:24 Medio:24 1st Qu.:90.38 1st Qu.:134.0
## MImuno:24 Alto :24 Median :92.00 Median :137.0
## Mean :92.12 Mean :137.9
## 3rd Qu.:93.90 3rd Qu.:142.0
## Max. :97.60 Max. :147.0# Tabela de ocorrência dos pontos experimentais.
xtabs(~sexo + dieta, data = tb)## dieta
## sexo Baixo Medio Alto
## Fem 8 8 8
## MCastr 8 8 8
## MImuno 8 8 8São 8 animais por ponto experimental. No entanto, algumas medidas foram perdidas ao longo do experimento. Então para algumas variáveis resposta não se tem o valor observado em 8 unidades experimentais.
O conjunto de gráficos a seguir avalia se existe relação entre as covariáveis peso inicial e idade e os fatores sob investigação. Como mencionado anteriormente, o peso inicial e idade dos animais foi determinado antes da casualiazação dos pontos experimentais às unidades experimentais. Dessa forma, não se espera encontrar relação. Todavia, nada impede de verificar o efeito de sexo e dieta no peso inicial e idade pelo quadro de análise de variância.
# Gráficos de distribuições acumuladas empíricas.
gridExtra::grid.arrange(
ggplot(data = tb,
mapping = aes(x = id)) +
stat_ecdf() +
geom_rug(),
ggplot(data = tb,
mapping = aes(x = pi)) +
stat_ecdf() +
geom_rug(),
ncol = 1)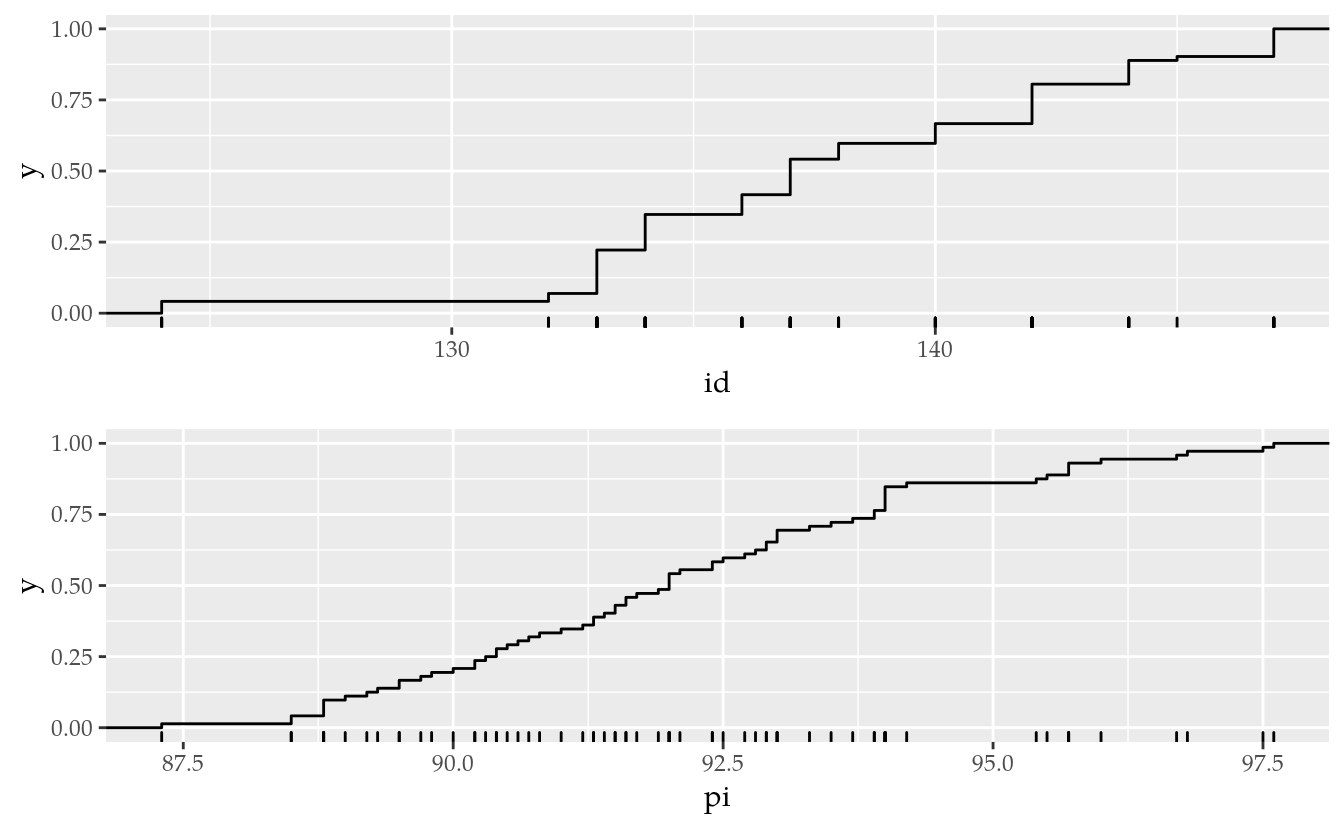
# Empilha as covariáveis `pi` e `id`.
tb_long <- tb %>%
gather(key = "variavel", value = "valor", pi, id)
# Calcula a média com intervalo de confiança por ponto experimental.
tb_desc <- tb_long %>%
group_by(sexo, dieta, variavel) %>%
drop_na() %>%
summarise(md = mean(valor),
se = sd(valor)/sqrt(n()),
lwr = md - 1.96 * se,
upr = md + 1.96 * se)
gg1 <- ggplot(data = tb_long) +
facet_grid(facets = variavel ~ dieta, scales = "free_y") +
geom_jitter(mapping = aes(x = sexo, y = valor),
width = 0.1) +
geom_errorbar(data = tb_desc,
mapping = aes(x = sexo, ymin = lwr, ymax = upr),
width = 0.1) +
geom_line(data = tb_desc,
mapping = aes(x = sexo, y = md, group = 1))
gg2 <- ggplot(data = tb_long) +
facet_grid(facets = variavel ~ sexo, scales = "free_y") +
geom_jitter(mapping = aes(x = dieta, y = valor),
width = 0.1) +
geom_errorbar(data = tb_desc,
mapping = aes(x = dieta, ymin = lwr, ymax = upr),
width = 0.1) +
geom_line(data = tb_desc,
mapping = aes(x = dieta, y = md, group = 1))
# Gráficos para verficar o efeito de sexo e dieta nas covariáveis.
gridExtra::grid.arrange(gg1, gg2, nrow = 1)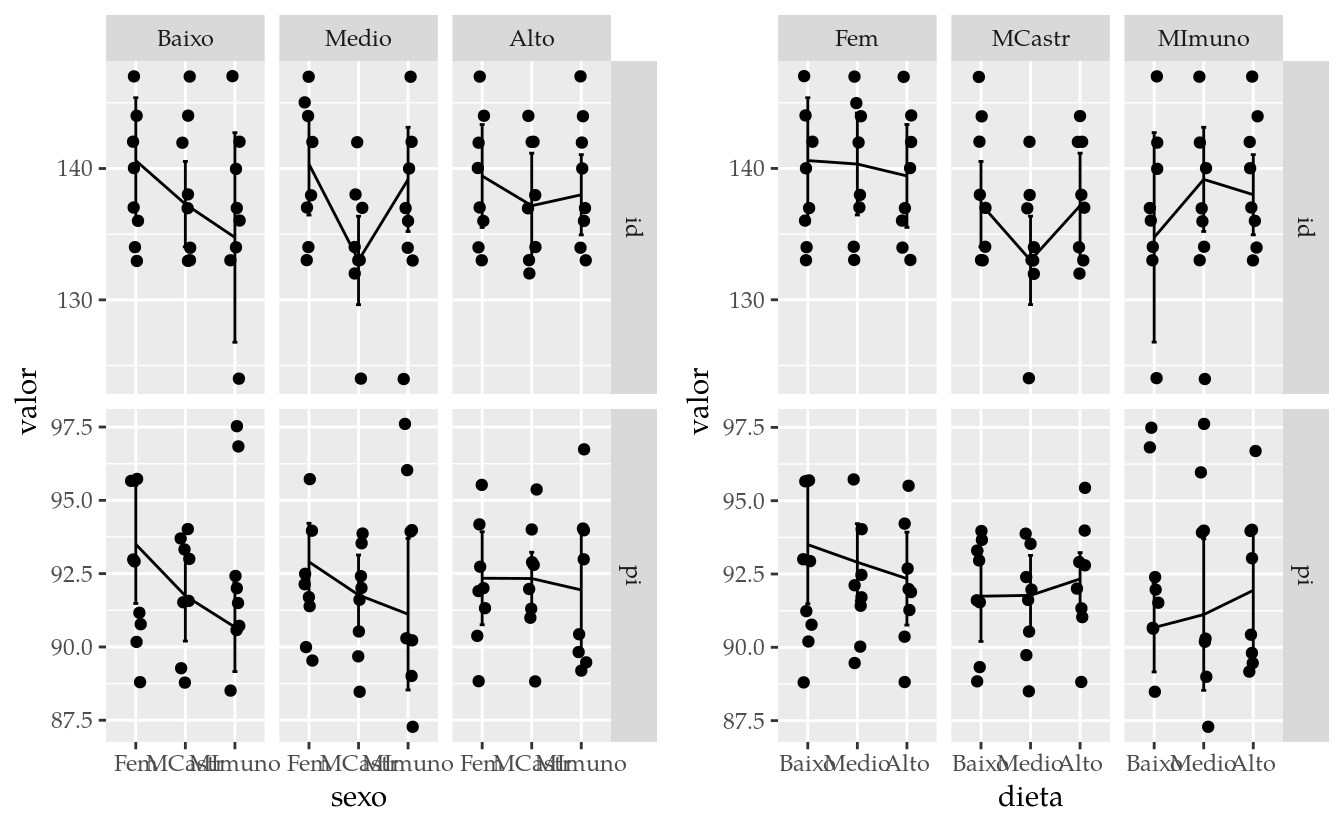
O último gráfico não fornece evidência de que o peso inicial e idade sejam afetados pelo sexo ou dieta. Todavia, poderia ocorrer das fêmeas serem mais leves/pesadas que os machos por uma questão natural em algumas espécies, como em frango de corte ou bovinos adultos.
Pode-se obter o quadro de análise de variância para as covariáveis como função dos termos experimentais para verificar se elas são explicadas pelos mesmos. Como as variáveis são medidas antes do inicio do experimento e como os animais foram aleatoriamente designinados aos níveis de dieta, espera-se resultado não significativo.
Não seria um problema, por exemplo, que as fêmeas fossem sistematicamente mais leves que os machos no experimento. O importante é que o efeito do peso inicial na variável resposta seja independente do sexo e do nível de dieta. O simples fato da covariável ser explicada pelos fatores experimentais não implica dizer que os efeitos são dependentes. Da mesma forma que o oposto, as covariáveis não serem explicadas pelos fatorires experimentais, não implica que os efeitos são independentes.
# Testa se `pi` e `id` são explicados por sexo * dieta.
m0 <- lm(cbind(id, pi) ~ sexo * dieta, data = tb)
# Anovas para cada variável resposta.
summary.aov(m0)## Response id :
## Df Sum Sq Mean Sq F value Pr(>F)
## sexo 2 89.36 44.681 1.4778 0.2359
## dieta 2 38.11 19.056 0.6303 0.5358
## sexo:dieta 4 86.89 21.722 0.7185 0.5825
## Residuals 63 1904.75 30.234
##
## Response pi :
## Df Sum Sq Mean Sq F value Pr(>F)
## sexo 2 1.93 0.9654 0.1541 0.8575
## dieta 2 0.84 0.4204 0.0671 0.9352
## sexo:dieta 4 2.38 0.5958 0.0951 0.9837
## Residuals 63 394.75 6.2658# Pode usar uma análise de variância multivariada.
# anova(m0)
Anova(m0)##
## Type II MANOVA Tests: Pillai test statistic
## Df test stat approx F num Df den Df Pr(>F)
## sexo 2 0.048895 0.78940 4 126 0.5341
## dieta 2 0.020527 0.32665 4 126 0.8597
## sexo:dieta 4 0.050446 0.40754 8 126 0.9145# Não significativo é o resultado esperado se os tratamentos foram
# casualizados aos animais.Nas seções a seguir serão analisas duas variáveis resposta. A primeira é ganho de peso e não apresenta interação entre sexo e dieta. Porém, a espessura do toucinho apresenta interação.
11.4.2 Peso aos 28 dias
Partimos com a análise exploratória para a variável resposta rendimento de pernil (rpernil). Os gráficos são para percebermos a relação entre as covariáveis e a resposta para declararmos da forma mais apropriada o seu efeito no modelo.
ggplot(data = tb,
mapping = aes(x = pi, y = rpernil)) +
facet_grid(facets = sexo ~ dieta) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")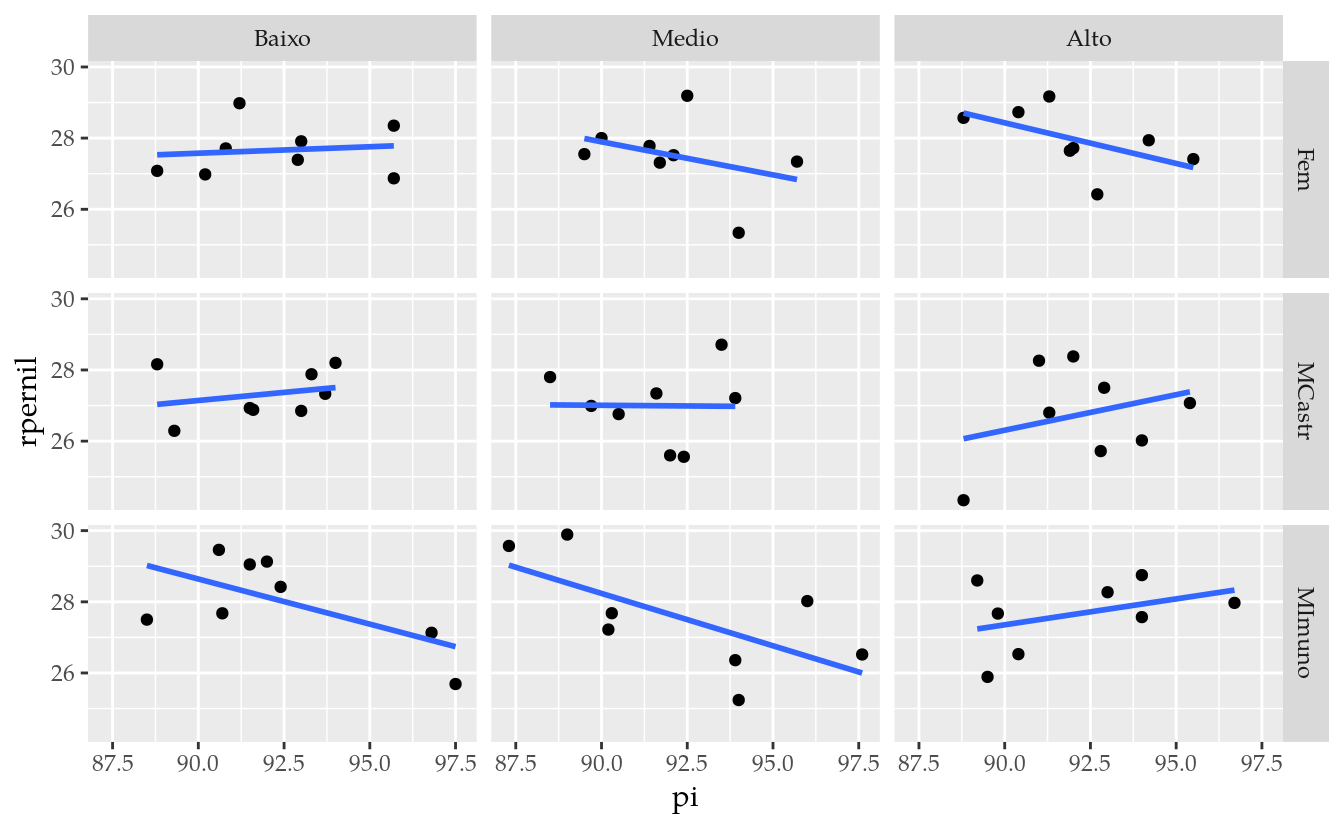
ggplot(data = tb,
mapping = aes(x = id, y = rpernil)) +
facet_grid(facets = sexo ~ dieta) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")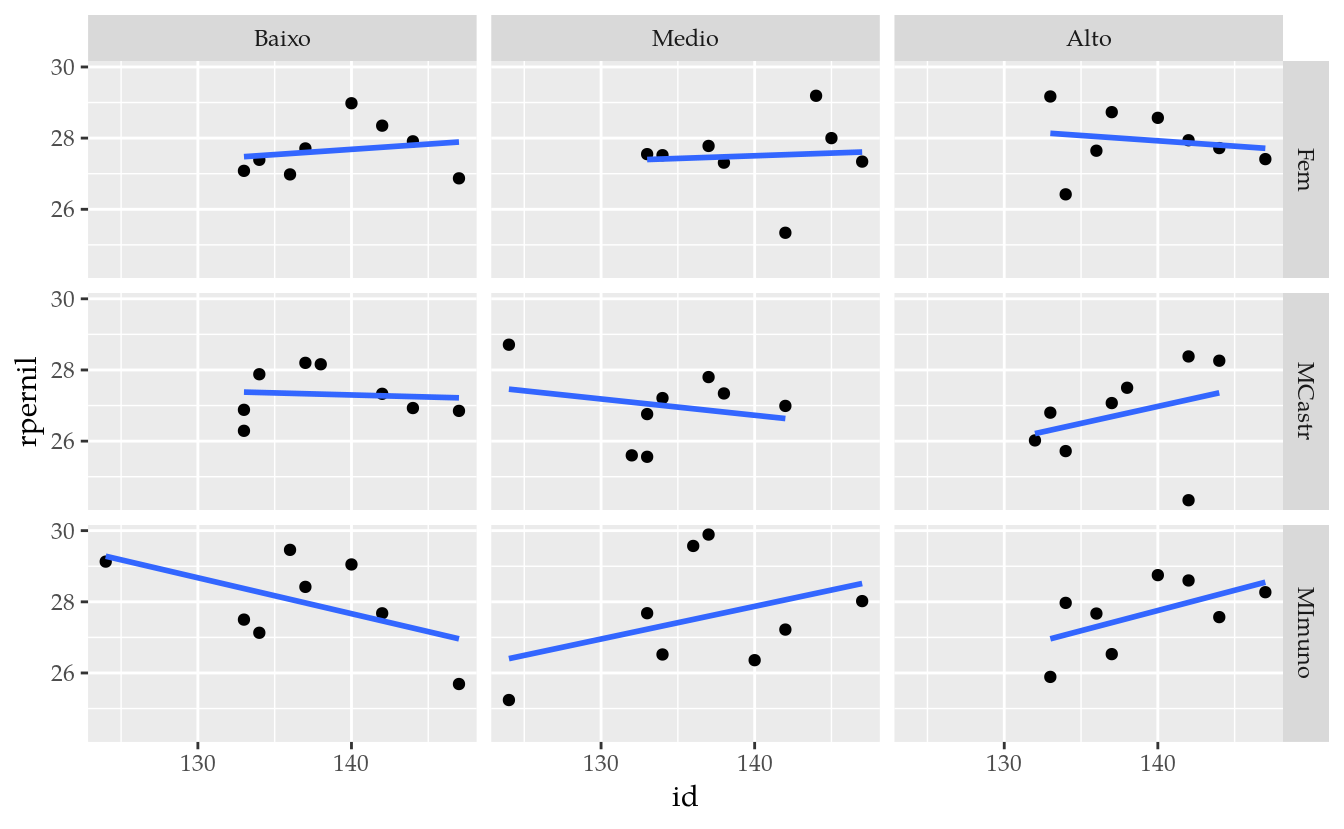
Os gráficos indicam haver moderado efeito do peso inicial, porém pouco efeito da idade no rendimento de pernil. De qualquer forma, a princípio o modelo linear de primeira ordem será suficiente para acomodar o efeito de cada covariável.
O modelo estatístico para esse experimento é \[ \begin{aligned} Y_{ijk} | \text{fontes de variação} &\sim \text{Normal}(\mu_{ijk}, \sigma^2) \newline \mu_{ijk} &= \mu + \beta_i + \alpha_j + \tau_k + \gamma \cdot \text{pi}_{ijk} + \psi \cdot \text{id}_{ijk} \end{aligned} \] em que \(Y_{ijk}\) é o valor observado da variável resposta, \(\beta_i\) é o efeito dos níveis de sexo \(i\), \(\alpha_j\) o efeito dos níveis de energia (Kcal) na dieta \(j\). \(\gamma\) é o acréscimo por unidade de peso inicial. \(\psi\) é o acréscimo por unidade de idade. $ é uma constante que incide em todas as observações. \(\mu_{ijk}\) é a média para as observações do ponto experimental \(ij\) na repetição \(k\) e \(\sigma^2\) é a variância das observações ao redor desse valor médio.
No bloco abaixo o modelo ajustados. Gráficos para diagnóstico baseados nos resíduos são feitos para avaliar a conformidade das suposições.
# Análise de variância (em experimentos não ortogonais a ordem dos
# termos é importante!).
m1 <- lm(rpernil ~ pi + id + sexo * dieta, data = tb)
# Avaliação dos pressupostos.
par(mfrow = c(2, 2)); plot(m1); layout(1)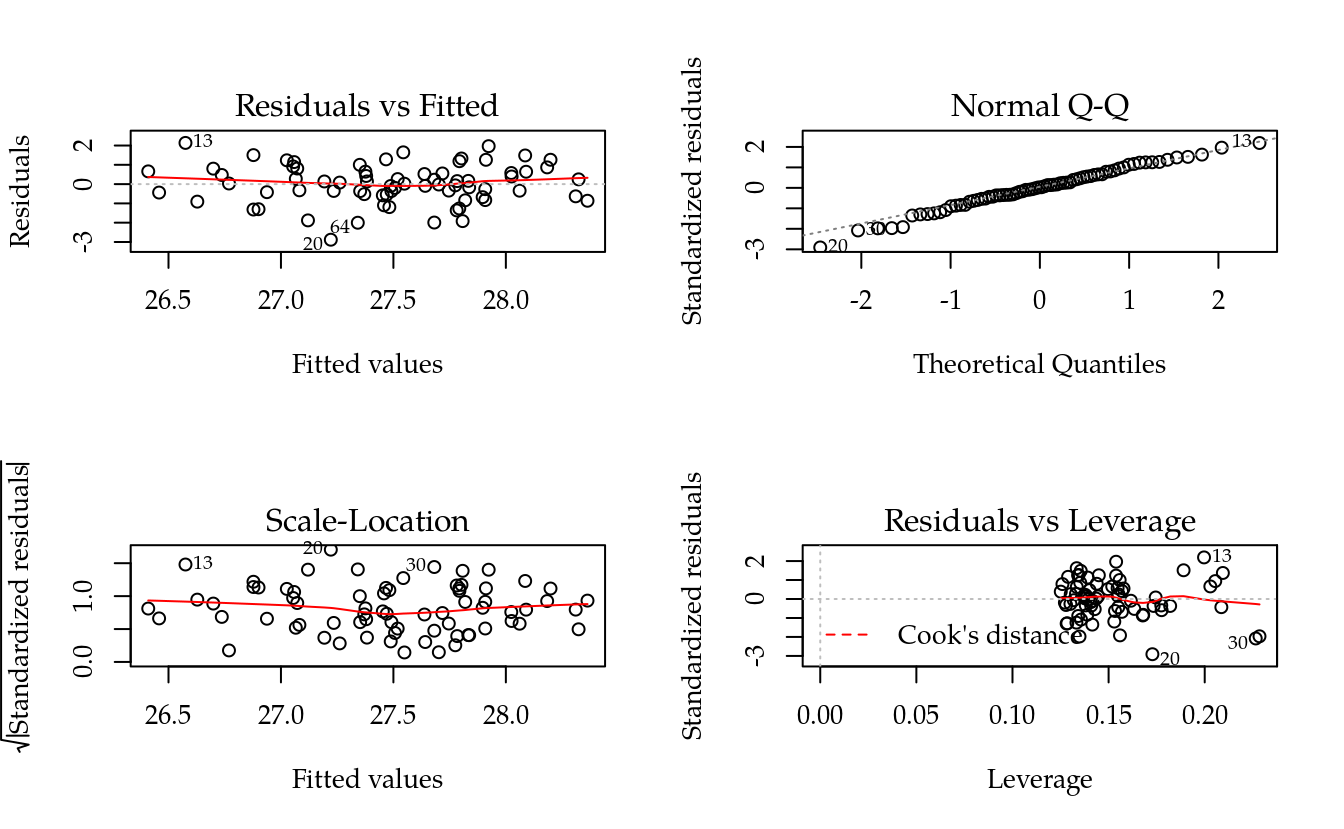
# Resíduos contra cada termo do modelo.
residualPlots(m1, layout = c(2, 3))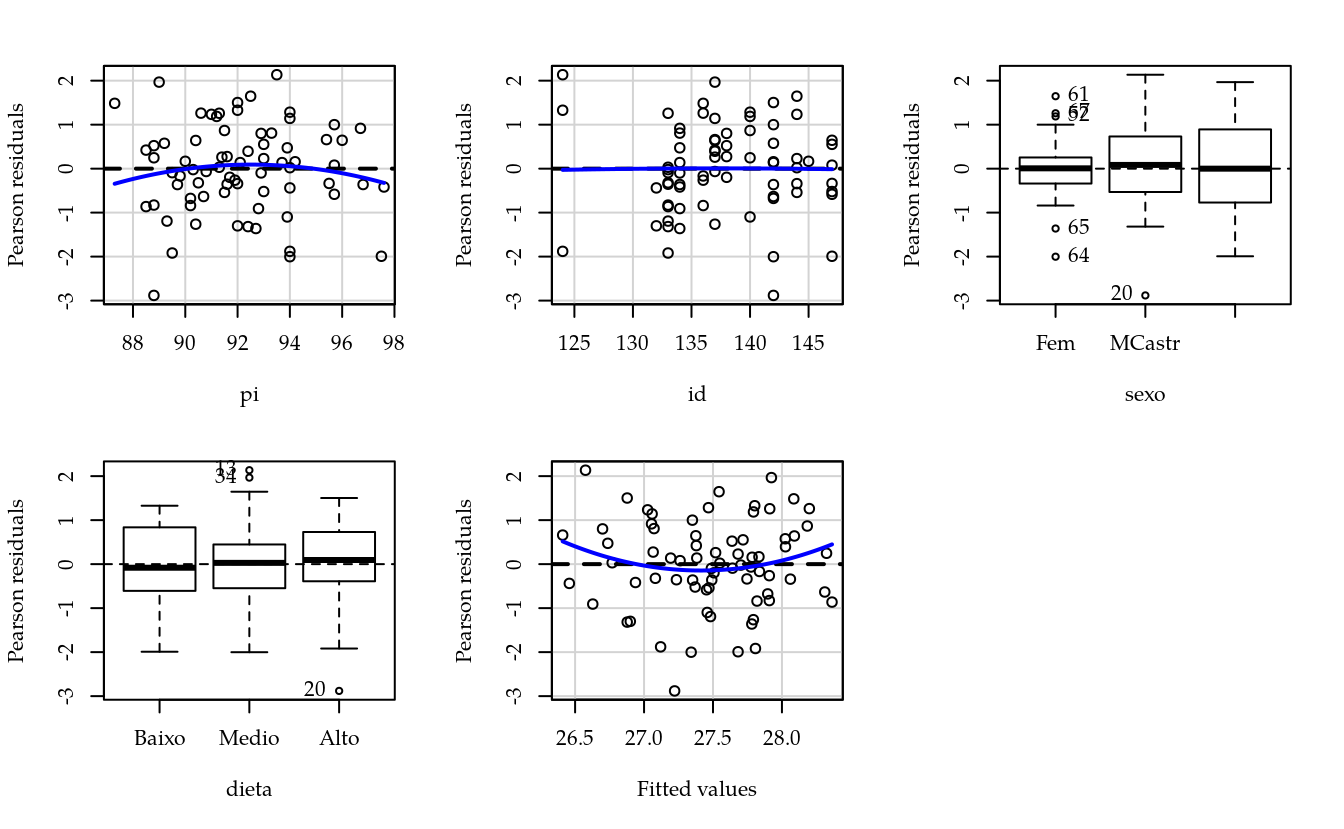
## Test stat Pr(>|Test stat|)
## pi -0.9040 0.36960
## id -0.0647 0.94866
## sexo
## dieta
## Tukey test 1.7619 0.07809 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Quadro de análise de variância.
# anova(m1)
# drop1(m1, test = "F", scope = . ~ .)
Anova(m1)## Anova Table (Type II tests)
##
## Response: rpernil
## Sum Sq Df F value Pr(>F)
## pi 4.413 1 3.7221 0.05835 .
## id 0.771 1 0.6500 0.42325
## sexo 7.908 2 3.3344 0.04224 *
## dieta 1.244 2 0.5244 0.59457
## sexo:dieta 1.735 4 0.3657 0.83208
## Residuals 72.331 61
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Ajuste do modelo reduzido.
m2 <- lm(rpernil ~ pi + sexo, data = tb)
anova(m2, m1)## Analysis of Variance Table
##
## Model 1: rpernil ~ pi + sexo
## Model 2: rpernil ~ pi + id + sexo * dieta
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 68 76.034
## 2 61 72.331 7 3.703 0.4461 0.869# Efeito do peso inicial.
coef(m2)["pi"]## pi
## -0.09907416Os gráficos de resíduos não revelam nada preocupante. Não há evidências contra as suposições de homocedasticidade e normalidade. Apenas o gráfico de resíduos contra os níveis de dieta parece indicar que a dispersão cresce com o nível de dieta. Um estudo mais detalhado poderia ser conduzido para averiguar essa potencial perturbação dos pressupostos, mas para não perder o foco, não feremos isso aqui.
O quadro de análise de variância não apontou existência de interação entre sexo e dieta nem o efeito de dieta. Existe apenas o efeito de sexo e do peso inicial no rendimento de pernil. Não contribuição do efeito de idade. Um modelo apenas com os termos relevantes foi ajustado. O teste da redução da soma de quadrados residual não geou evidências que de modelo mais simples é inferior ao modelo completo.
Nos fragmentos de código abaixo serão estudado em mais detalhe o efeito de sexo com o emprego de comparações múltiplas de médias.
# Médias marginais estimadas pelo modelo.
emm2 <- emmeans(m2, specs = ~sexo)
emm2 %>% as.data.frame()## sexo emmean SE df lower.CL upper.CL
## 1 Fem 27.70937 0.2158617 68 27.27863 28.14012
## 2 MCastr 27.00229 0.2161626 68 26.57094 27.43363
## 3 MImuno 27.75888 0.2160353 68 27.32779 28.18998# Constrastes dois a dois entre médias marginais. O modelo tem que ser
# de efeitos aditivos para os resultados da `mcp()` serem seguros.
summary(glht(m2, linfct = mcp(sexo = "Tukey")))##
## Simultaneous Tests for General Linear Hypotheses
##
## Multiple Comparisons of Means: Tukey Contrasts
##
##
## Fit: lm(formula = rpernil ~ pi + sexo, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## MCastr - Fem == 0 -0.70708 0.30559 -2.314 0.0606 .
## MImuno - Fem == 0 0.04951 0.30532 0.162 0.9856
## MImuno - MCastr == 0 0.75660 0.30596 2.473 0.0416 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)# Condições experimentais das médias marginais e matriz associada.
grid <- attr(emm2, "grid")
L <- attr(emm2, "linfct")
rownames(L) <- grid$sexo
L## (Intercept) pi sexoMCastr sexoMImuno
## Fem 1 92.11667 0 0
## MCastr 1 92.11667 1 0
## MImuno 1 92.11667 0 1# Comparações multiplas entre médias marginais.
results <- wzRfun::apmc(L, model = m2, focus = "sexo",
test = "single-step") %>%
mutate(sexo = factor(sexo, levels = sexo))
# Gráfico de segmentos para o intervalo de confiança.
ggplot(data = results,
mapping = aes(x = fit, y = sexo)) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = "Rendimento de pernil",
y = "Sexo")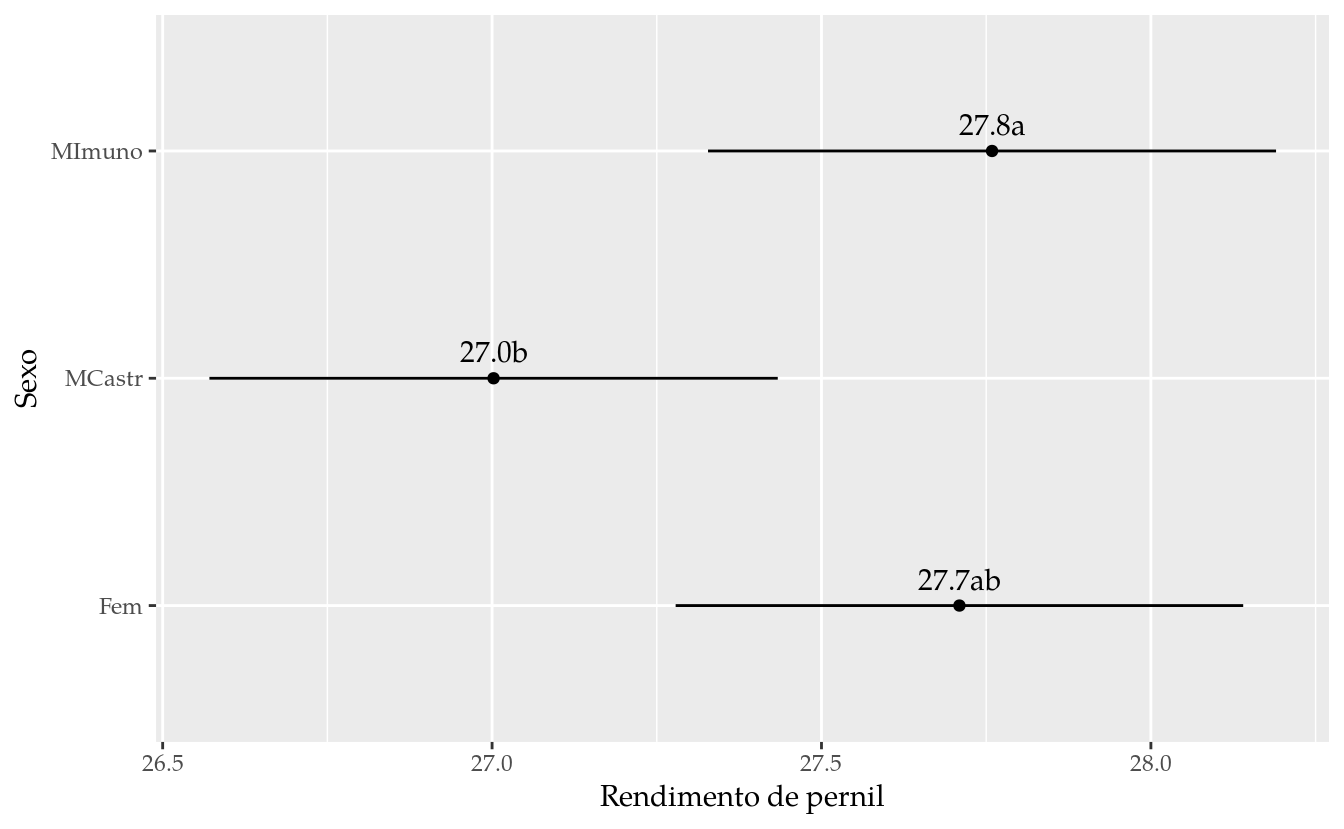
Os resultados apenas indicam diferença entre o rendimento de pernil do macho com castração física pro de castração hormonal. IMPROVE.
11.4.3 Expessura de toucinho de barriga
Começamos com a análise exploratória para a variável resposta expessura de toucinho de barriga (etbar).
ggplot(data = tb,
mapping = aes(x = pi, y = etbar)) +
facet_grid(facets = sexo ~ dieta) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")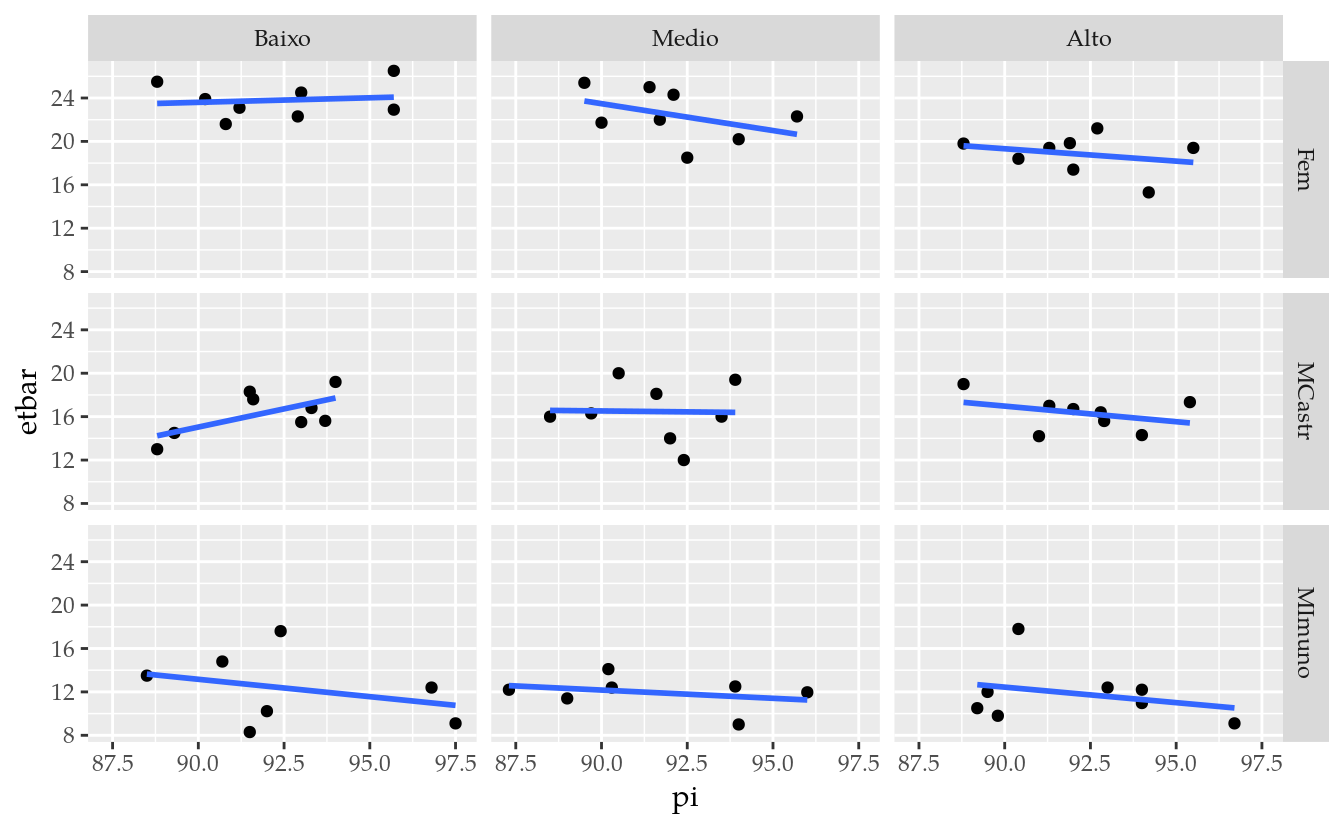
ggplot(data = tb,
mapping = aes(x = id, y = etbar)) +
facet_grid(facets = sexo ~ dieta) +
geom_point() +
geom_smooth(se = FALSE, method = "lm")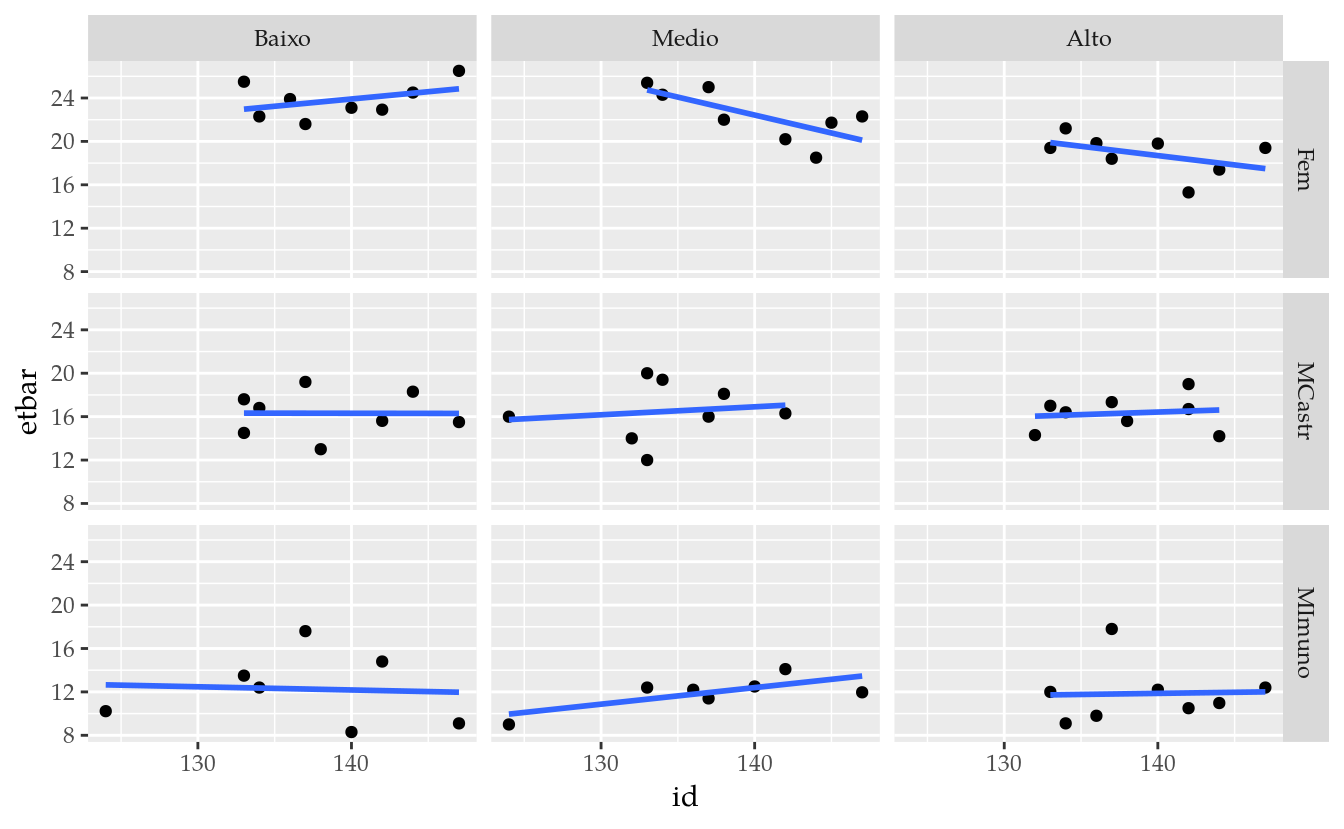
Considerando os gráficos não parece haver efeito da idade na espessura de toucinho de barriga. O efeito de peso inicial se mostra com valor divergente entre os níveis de sexo. Seria uma evidência contra a suposição de aditividade de efeitos?
O modelo estatístico assumido para esta variável é o mesmo declarado anteriormente para rendimento de pernil.
# Análise de variância (em experimentos não ortogonais a ordem dos
# termos é importante!).
m1 <- lm(etbar ~ pi + id + sexo * dieta, data = tb)
# Avaliação dos pressupostos.
par(mfrow = c(2, 2)); plot(m1); layout(1)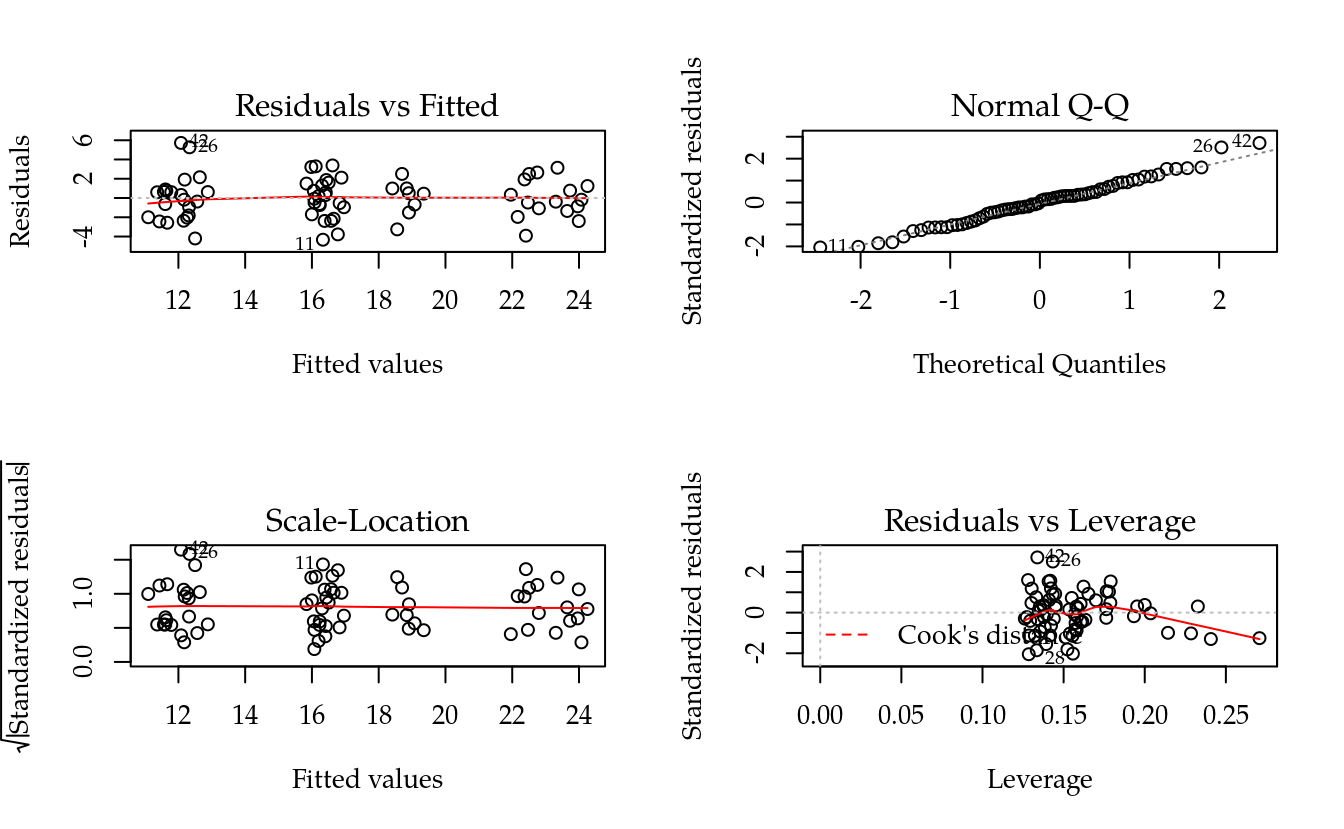
# Resíduos contra cada termo do modelo.
residualPlots(m1, layout = c(2, 3))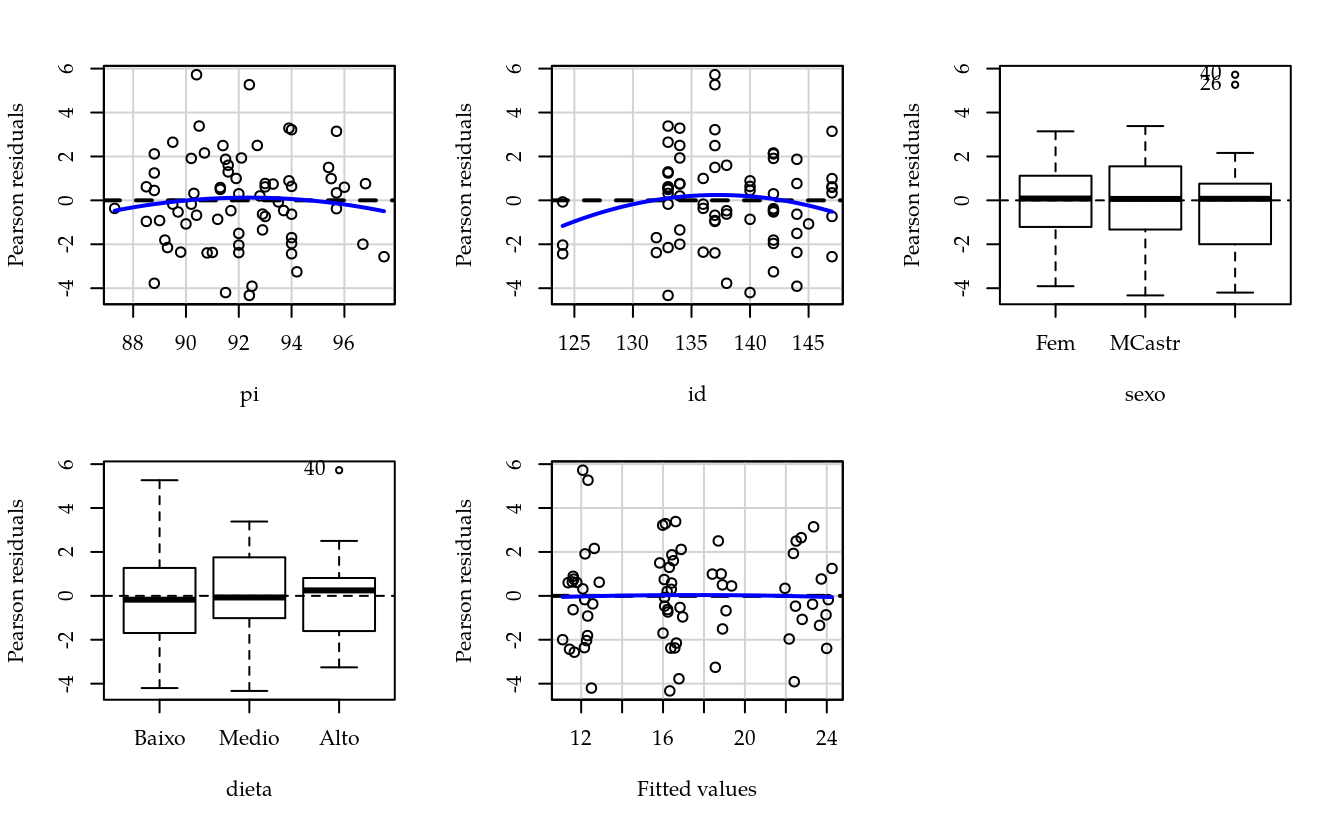
## Test stat Pr(>|Test stat|)
## pi -0.5774 0.5659
## id -1.3591 0.1794
## sexo
## dieta
## Tukey test -0.6484 0.5167# Quadro de análise de variância.
# anova(m1)
# drop1(m1, test = "F", scope = . ~ .)
Anova(m1)## Anova Table (Type II tests)
##
## Response: etbar
## Sum Sq Df F value Pr(>F)
## pi 7.81 1 1.5224 0.22215
## id 0.19 1 0.0366 0.84892
## sexo 1043.35 2 101.6996 < 2e-16 ***
## dieta 41.77 2 4.0719 0.02205 *
## sexo:dieta 65.05 4 3.1703 0.01992 *
## Residuals 302.64 59
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Ajuste do modelo reduzido.
m2 <- lm(etbar ~ sexo * dieta, data = tb)
anova(m2, m1)## Analysis of Variance Table
##
## Model 1: etbar ~ sexo * dieta
## Model 2: etbar ~ pi + id + sexo * dieta
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 61 310.48
## 2 59 302.64 2 7.8318 0.7634 0.4706Não houve efeito de peso inicial e idade na espessura de toucinho de barriga. A interação entre sexo e dieta foi significativa. Nós códigos a seguir será efeito estudo do nível de sexo em cada nível de dieta por meio de comparações múltiplas de médias.
# emm2 <- emmeans(m2, specs = ~sexo + dieta)
# emm2 %>% as.data.frame()
emm2 <- emmeans(m2, specs = ~dieta | sexo)
emm2## sexo = Fem:
## dieta emmean SE df lower.CL upper.CL
## Baixo 23.8 0.798 61 22.2 25.4
## Medio 22.4 0.798 61 20.8 24.0
## Alto 18.8 0.798 61 17.2 20.4
##
## sexo = MCastr:
## dieta emmean SE df lower.CL upper.CL
## Baixo 16.3 0.798 61 14.7 17.9
## Medio 16.5 0.798 61 14.9 18.1
## Alto 16.3 0.798 61 14.7 17.9
##
## sexo = MImuno:
## dieta emmean SE df lower.CL upper.CL
## Baixo 12.3 0.853 61 10.6 14.0
## Medio 11.9 0.853 61 10.2 13.6
## Alto 11.8 0.798 61 10.3 13.4
##
## Confidence level used: 0.95grid <- attr(emm2, "grid")
L <- attr(emm2, "linfct")
rownames(L) <- with(grid, paste(sexo, dieta, sep = ":"))
L %>% `colnames<-`(NULL)## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## Fem:Baixo 1 0 0 0 0 0 0 0 0
## Fem:Medio 1 0 0 1 0 0 0 0 0
## Fem:Alto 1 0 0 0 1 0 0 0 0
## MCastr:Baixo 1 1 0 0 0 0 0 0 0
## MCastr:Medio 1 1 0 1 0 1 0 0 0
## MCastr:Alto 1 1 0 0 1 0 0 1 0
## MImuno:Baixo 1 0 1 0 0 0 0 0 0
## MImuno:Medio 1 0 1 1 0 0 1 0 0
## MImuno:Alto 1 0 1 0 1 0 0 0 1# Estudar a diferença entre níveis de dieta em cada sexo.
grid_grp <- grid %>%
group_by(sexo)
# Matriz com termos para determinar médias ajustadas para os pontos
# experimentais.
X_grp <-
by(L,
INDICES = group_indices(grid_grp),
FUN = as.matrix,
rownames.force = TRUE) %>%
setNames(nm = group_keys(grid_grp)[[1]])
# Obtém as comparações múltiplas em cada estrato.
results <- lapply(X_grp,
FUN = wzRfun::apmc,
model = m2,
focus = "dieta") %>%
do.call(what = rbind) %>%
separate(dieta, into = c("sexo", "dieta")) %>%
wzRfun::equallevels(tb) %>%
`rownames<-`(NULL)
results## sexo dieta fit lwr upr cld
## 1 Fem Baixo 23.79063 22.19566 25.38559 a
## 2 Fem Medio 22.42813 20.83316 24.02309 a
## 3 Fem Alto 18.84225 17.24728 20.43722 b
## 4 MCastr Baixo 16.31413 14.71916 17.90909 a
## 5 MCastr Medio 16.47500 14.88003 18.06997 a
## 6 MCastr Alto 16.31725 14.72228 17.91222 a
## 7 MImuno Baixo 12.27500 10.56991 13.98009 a
## 8 MImuno Medio 11.93757 10.23248 13.64267 a
## 9 MImuno Alto 11.84688 10.25191 13.44184 a# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results,
mapping = aes(x = fit, y = dieta)) +
facet_wrap(facets = ~sexo, ncol = 1) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = "Espessura de toucinho",
y = "Energia na dieta")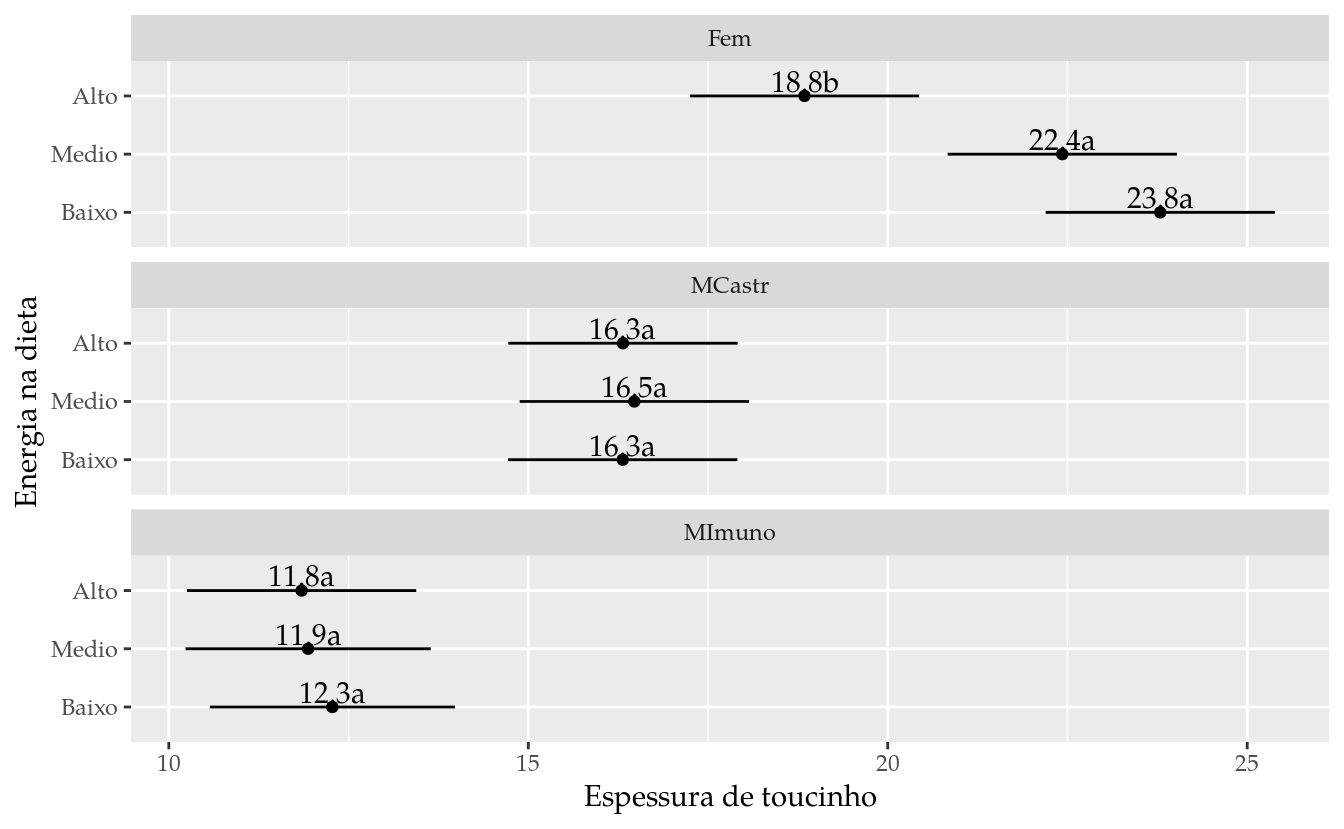
Os resultados mostram que não há efeito de dieta para os machos, apenas na fêmea, de tal forma que a espessura de toucinho de barriga é menor com alta energia na dieta.
11.5 Outro exemplo
str(labestData::RamalhoEg13.2) # tamanho amostral.## 'data.frame': 36 obs. of 5 variables:
## $ milh : Factor w/ 4 levels "BR201","C515",..: 1 1 1 4 4 4 2 2 2 3 ...
## $ feij : Factor w/ 3 levels "Carioca","E585",..: 3 2 1 3 2 1 3 2 1 3 ...
## $ bloc : Factor w/ 3 levels "1","2","3": 1 1 1 1 1 1 1 1 1 1 ...
## $ prod : int 8268 7170 10646 4211 5357 5589 6040 7006 7445 7392 ...
## $ plant: int 20 20 20 18 20 18 21 19 20 20 ...11.6 Considerações finais
FIXME TODO
11.7 Delineamento ótimo
TODO: como alocar os animais conhecendo previamente
- Os pesos iniciais
pi. - As idades
id. - Que serão estudados 9 pontos experimentais de igual tamanho.
FIXME ???
- Ordenar em
pi. - A cada 9 animais (bloco de tamanho igual ao número de pontos experimentais) em ordem, casualizar um dos pontos experimentais.
- Como fazer simultâneamente com as duas variáveis?
- Padronizar as escalas.
- Obter um k-means de 9 centróides e tamanho 9?
- Fazer sequêncial, separando 2 grupos sendo um com 9 pontos. Repetir com os demais pontos.
Referências Bibliográficas
ZIMMERMANN, F. J. Estatística aplicada à pesquisa agrícola. 1st ed. Santo Antônio de Goiás, GO: Embrapa Arroz e Feijão, 2004.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani