Capítulo 19 Análise conjunta de experimentos
Atenção: as análises nesse capítulo estão considerando apenas modelos de efeitos fixos. Posteriormente serão incluídas análises com modelos de efeitos mistos.
rm(list = objects())
library(agricolae) # HSD.test().
library(emmeans) # emmeans().
library(multcomp) # glht() e métodos.
library(tidyverse) # Manipulação e visualização.
# Funções.
source("mpaer_functions.R")19.1 Experimentos em delineamento inteiramente casualizado
Os dados em RamalhoEx8.1 são da produção de grãos (kg/ha) obtidos na avaliação de cultivares de arroz conduzidos em três locais no Estado de Minas Gerais. O experimento avaliou 10 cultivares em delineamento inteiramente casualizado com 3 repetições em cada local.
19.1.1 Análise exploratória
# github.com/pet-estatistica/labestData/blob/devel/data-raw/RamalhoEx8.1.txt
# help(RamalhoEx8.1, package = "labestData")
da <- labestData::RamalhoEx8.1
da$prod <- da$prod/1000
da$rept <- factor(da$rept)
str(da)## 'data.frame': 90 obs. of 4 variables:
## $ cult : Factor w/ 10 levels "1","2","3","4",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ local: Factor w/ 3 levels "Felixlandia",..: 2 2 2 2 2 2 2 2 2 2 ...
## $ rept : Factor w/ 3 levels "1","2","3": 1 1 1 1 1 1 1 1 1 1 ...
## $ prod : num 4.89 4.17 5.62 4.58 4.31 ...xtabs(~local + cult, data = da)## cult
## local 1 2 3 4 5 6 7 8 9 10
## Felixlandia 3 3 3 3 3 3 3 3 3 3
## Lambari 3 3 3 3 3 3 3 3 3 3
## Lavras 3 3 3 3 3 3 3 3 3 3ggplot(data = da,
mapping = aes(x = cult, y = prod)) +
facet_wrap(facets = ~local, ncol = 1) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")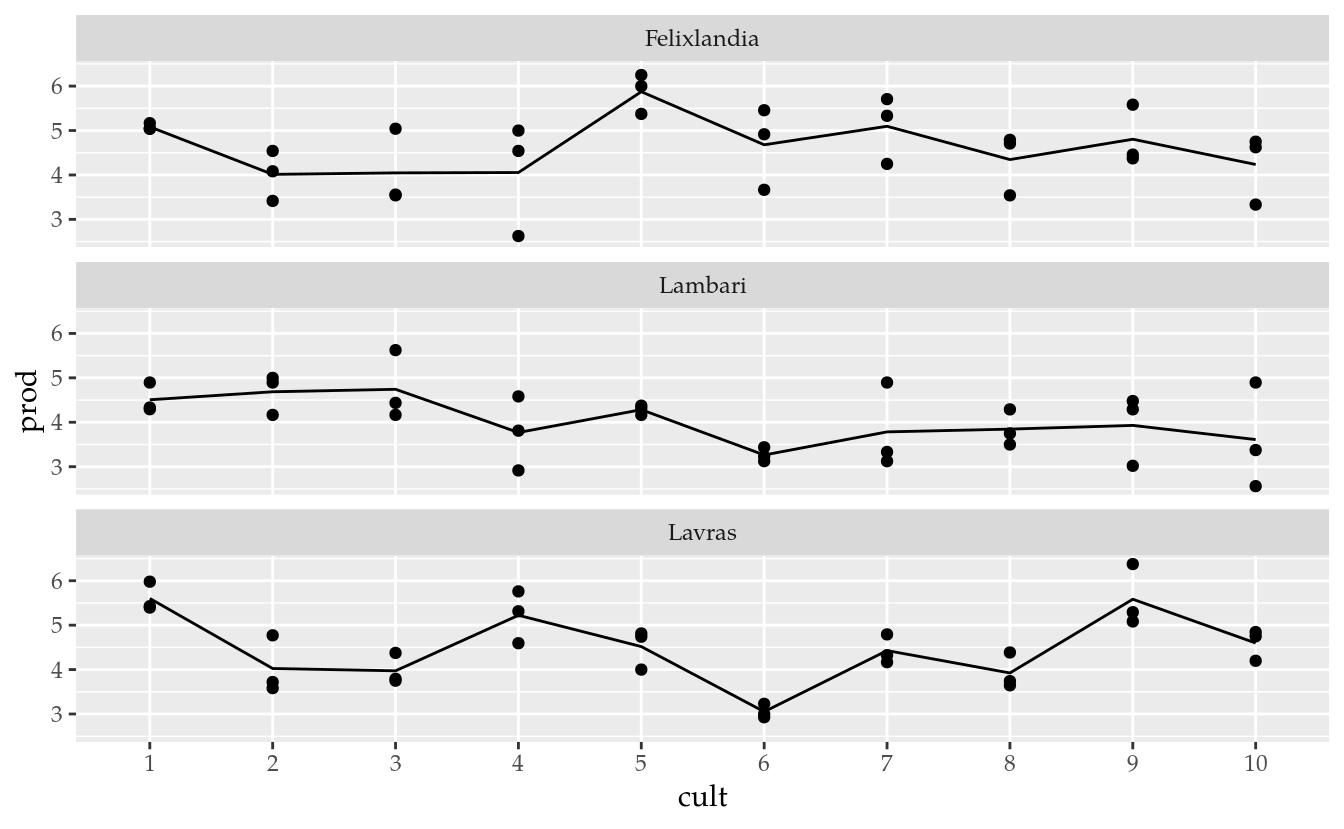
19.1.2 Análises individuais
da_local <- split(da, f = da$local)
# Análise para cada local.
m0_local <- lapply(da_local,
FUN = function(data) {
lm(prod ~ cult, data = data)
})
# Gráficos de normalidade.
par(mfrow = c(1, 3))
sapply(m0_local, FUN = plot, which = 2)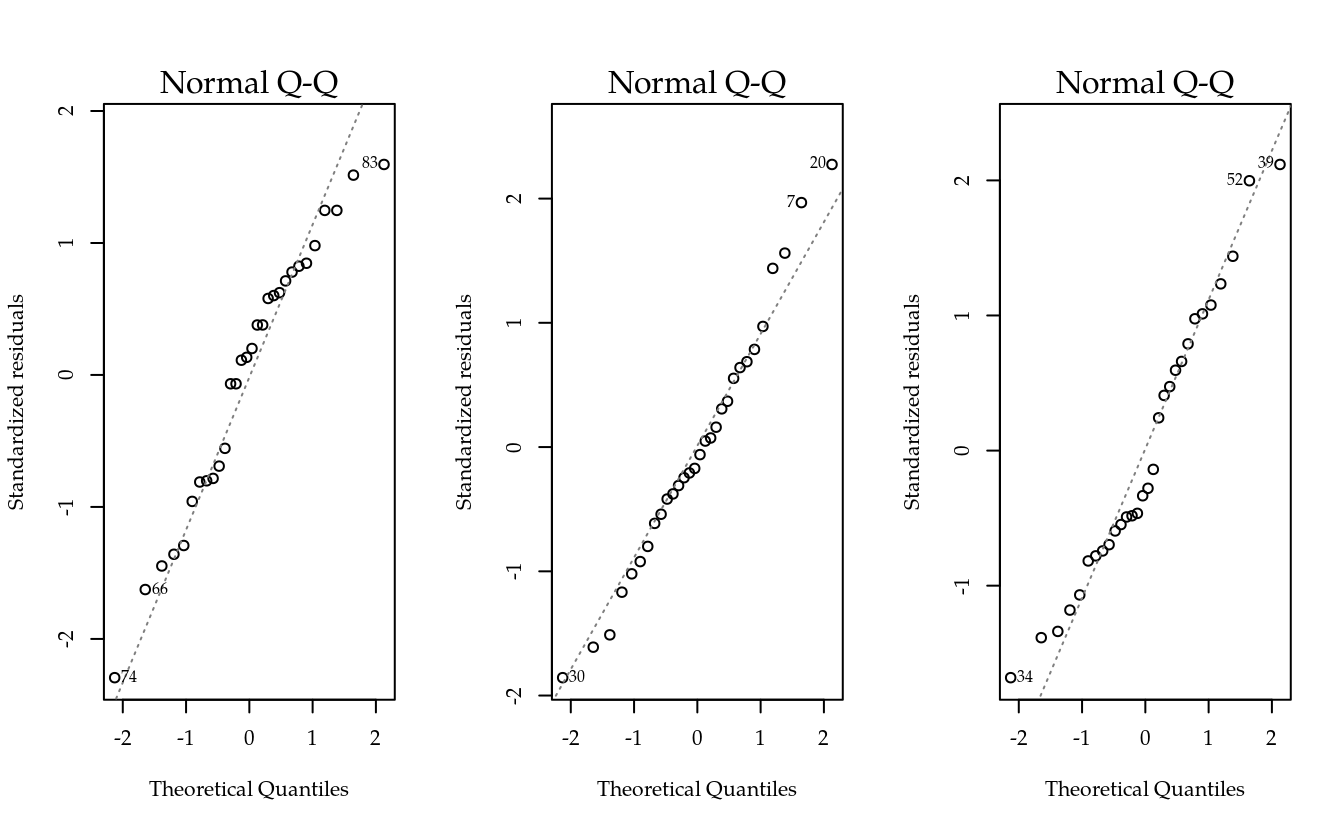
## $Felixlandia
## NULL
##
## $Lambari
## NULL
##
## $Lavras
## NULLlayout(1)
# Quadrados médios.
sapply(m0_local, FUN = function(m0) deviance(m0)/df.residual(m0))## Felixlandia Lambari Lavras
## 0.5831171 0.4785143 0.2096028# Quadro de análise de variância.
lapply(m0_local, FUN = anova)## $Felixlandia
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 9 9.8681 1.09645 1.8803 0.1149
## Residuals 20 11.6623 0.58312
##
## $Lambari
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 9 6.4959 0.72176 1.5083 0.2119
## Residuals 20 9.5703 0.47851
##
## $Lavras
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 9 17.5645 1.9516 9.311 1.97e-05 ***
## Residuals 20 4.1921 0.2096
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Teste de Tukey.
tk <- lapply(m0_local,
FUN = function(m0) {
tb <- HSD.test(m0, trt = "cult")$groups
tb$groups <- trimws(as.character(tb$groups))
cbind(cult = rownames(tb), tb)
})
tk## $Felixlandia
## cult prod groups
## 5 5 5.874000 a
## 7 7 5.096000 a
## 1 1 5.082667 a
## 9 9 4.804667 a
## 6 6 4.679667 a
## 8 8 4.346667 a
## 10 10 4.235333 a
## 4 4 4.055000 a
## 3 3 4.046667 a
## 2 2 4.013333 a
##
## $Lambari
## cult prod groups
## 3 3 4.742333 a
## 2 2 4.686667 a
## 1 1 4.507000 a
## 5 5 4.283667 a
## 9 9 3.929667 a
## 8 8 3.846000 a
## 7 7 3.783667 a
## 4 4 3.769667 a
## 10 10 3.610333 a
## 6 6 3.263333 a
##
## $Lavras
## cult prod groups
## 1 1 5.600667 a
## 9 9 5.583333 a
## 4 4 5.222333 ab
## 10 10 4.597333 ab
## 5 5 4.517667 ab
## 7 7 4.427333 ab
## 2 2 4.024333 bc
## 3 3 3.972333 bc
## 8 8 3.923667 bc
## 6 6 3.052000 c19.1.3 Análise conjunta
A análise conjunta está considerando efeito fixo para local e cultivar.
# Modelo com apenas um estrato para verificar pressupostos.
m0 <- lm(terms(prod ~ local + local:rept + cult + local:cult,
keep.order = TRUE),
data = da)
par(mfrow = c(2, 2))
plot(m0)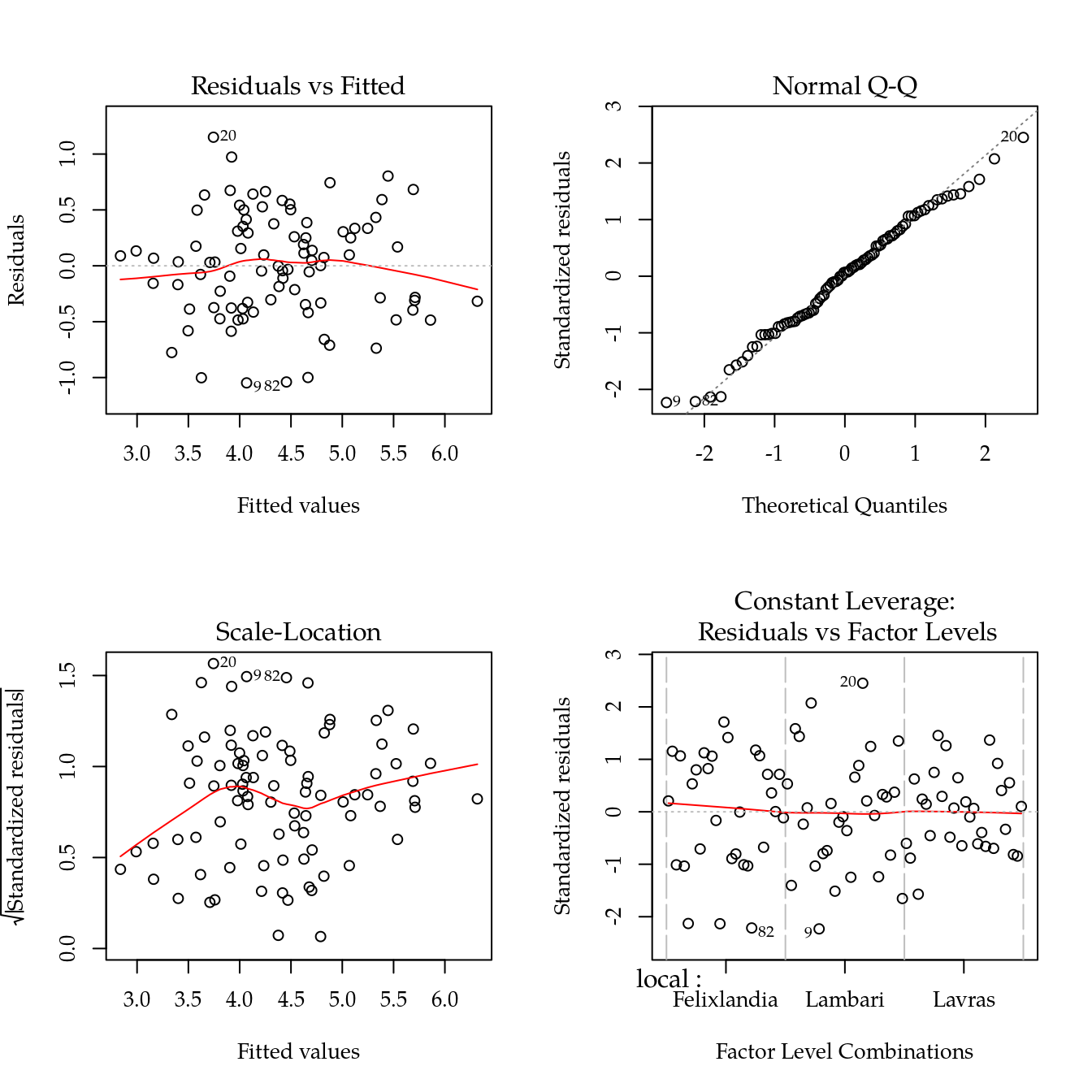
layout(1)
# Modelo com dois estratos.
m1 <- aov(prod ~ local + Error(local:rept) + cult + local:cult,
data = da)
# Quadro de anova.
summary(m1)##
## Error: local:rept
## Df Sum Sq Mean Sq F value Pr(>F)
## local 2 5.574 2.7869 2.991 0.126
## Residuals 6 5.590 0.9317
##
## Error: Within
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 9 14.43 1.6036 4.366 0.000261 ***
## local:cult 18 19.50 1.0831 2.949 0.001111 **
## Residuals 54 19.84 0.3673
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Médias marginais ajustadas por local.
emm <- emmeans(m1, specs = ~cult | local)## Note: re-fitting model with sum-to-zero contrastsemm## local = Felixlandia:
## cult emmean SE df lower.CL upper.CL
## 1 5.08 0.376 51.7 4.33 5.84
## 2 4.01 0.376 51.7 3.26 4.77
## 3 4.05 0.376 51.7 3.29 4.80
## 4 4.05 0.376 51.7 3.30 4.81
## 5 5.87 0.376 51.7 5.12 6.63
## 6 4.68 0.376 51.7 3.93 5.43
## 7 5.10 0.376 51.7 4.34 5.85
## 8 4.35 0.376 51.7 3.59 5.10
## 9 4.80 0.376 51.7 4.05 5.56
## 10 4.24 0.376 51.7 3.48 4.99
##
## local = Lambari:
## cult emmean SE df lower.CL upper.CL
## 1 4.51 0.376 51.7 3.75 5.26
## 2 4.69 0.376 51.7 3.93 5.44
## 3 4.74 0.376 51.7 3.99 5.50
## 4 3.77 0.376 51.7 3.02 4.52
## 5 4.28 0.376 51.7 3.53 5.04
## 6 3.26 0.376 51.7 2.51 4.02
## 7 3.78 0.376 51.7 3.03 4.54
## 8 3.85 0.376 51.7 3.09 4.60
## 9 3.93 0.376 51.7 3.18 4.68
## 10 3.61 0.376 51.7 2.86 4.36
##
## local = Lavras:
## cult emmean SE df lower.CL upper.CL
## 1 5.60 0.376 51.7 4.85 6.35
## 2 4.02 0.376 51.7 3.27 4.78
## 3 3.97 0.376 51.7 3.22 4.73
## 4 5.22 0.376 51.7 4.47 5.98
## 5 4.52 0.376 51.7 3.76 5.27
## 6 3.05 0.376 51.7 2.30 3.81
## 7 4.43 0.376 51.7 3.67 5.18
## 8 3.92 0.376 51.7 3.17 4.68
## 9 5.58 0.376 51.7 4.83 6.34
## 10 4.60 0.376 51.7 3.84 5.35
##
## Warning: EMMs are biased unless design is perfectly balanced
## Confidence level used: 0.95# Testes de Tukey por local da análise conjunta.
# ATTENTION: assume balanceamento.
tk <-
lapply(da_local,
FUN = function(data) {
rdf <- df.residual(m0)
rms <- deviance(m0)/rdf
tb <- HSD.test(y = data$prod,
trt = data$cult,
DFerror = rdf,
MSerror = rms)
tb <- tb$groups
tb$groups <- trimws(as.character(tb$groups))
names(tb)[1] <- "prod"
cbind(cult = rownames(tb), tb,
stringsAsFactors = FALSE)
})
# tk
# Junta para ter os intervalos de confiança.
tk <- bind_rows(tk, .id = "local")
tk <- inner_join(as.data.frame(emm), tk,
by = c("cult", "local"))
tk[, c(1, 2, 8, 6:7, 9)]## cult local prod lower.CL upper.CL groups
## 1 1 Felixlandia 5.082667 4.328418 5.836915 ab
## 2 2 Felixlandia 4.013333 3.259085 4.767582 b
## 3 3 Felixlandia 4.046667 3.292418 4.800915 b
## 4 4 Felixlandia 4.055000 3.300752 4.809248 b
## 5 5 Felixlandia 5.874000 5.119752 6.628248 a
## 6 6 Felixlandia 4.679667 3.925418 5.433915 ab
## 7 7 Felixlandia 5.096000 4.341752 5.850248 ab
## 8 8 Felixlandia 4.346667 3.592418 5.100915 ab
## 9 9 Felixlandia 4.804667 4.050418 5.558915 ab
## 10 10 Felixlandia 4.235333 3.481085 4.989582 b
## 11 1 Lambari 4.507000 3.752752 5.261248 a
## 12 2 Lambari 4.686667 3.932418 5.440915 a
## 13 3 Lambari 4.742333 3.988085 5.496582 a
## 14 4 Lambari 3.769667 3.015418 4.523915 a
## 15 5 Lambari 4.283667 3.529418 5.037915 a
## 16 6 Lambari 3.263333 2.509085 4.017582 a
## 17 7 Lambari 3.783667 3.029418 4.537915 a
## 18 8 Lambari 3.846000 3.091752 4.600248 a
## 19 9 Lambari 3.929667 3.175418 4.683915 a
## 20 10 Lambari 3.610333 2.856085 4.364582 a
## 21 1 Lavras 5.600667 4.846418 6.354915 a
## 22 2 Lavras 4.024333 3.270085 4.778582 abc
## 23 3 Lavras 3.972333 3.218085 4.726582 abc
## 24 4 Lavras 5.222333 4.468085 5.976582 ab
## 25 5 Lavras 4.517667 3.763418 5.271915 abc
## 26 6 Lavras 3.052000 2.297752 3.806248 c
## 27 7 Lavras 4.427333 3.673085 5.181582 abc
## 28 8 Lavras 3.923667 3.169418 4.677915 bc
## 29 9 Lavras 5.583333 4.829085 6.337582 a
## 30 10 Lavras 4.597333 3.843085 5.351582 abc# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = tk,
mapping = aes(x = prod, y = cult)) +
facet_wrap(facets = ~local) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lower.CL, xmax = upper.CL),
height = 0) +
geom_label(mapping = aes(label = sprintf("%0.2f%s", prod, groups)),
label.padding = unit(0.15, "lines"),
size = 3,
vjust = -0.25) +
expand_limits(y = c(NA, nlevels(da$cult) + 1)) +
labs(x = "Produção",
y = "Cultivares")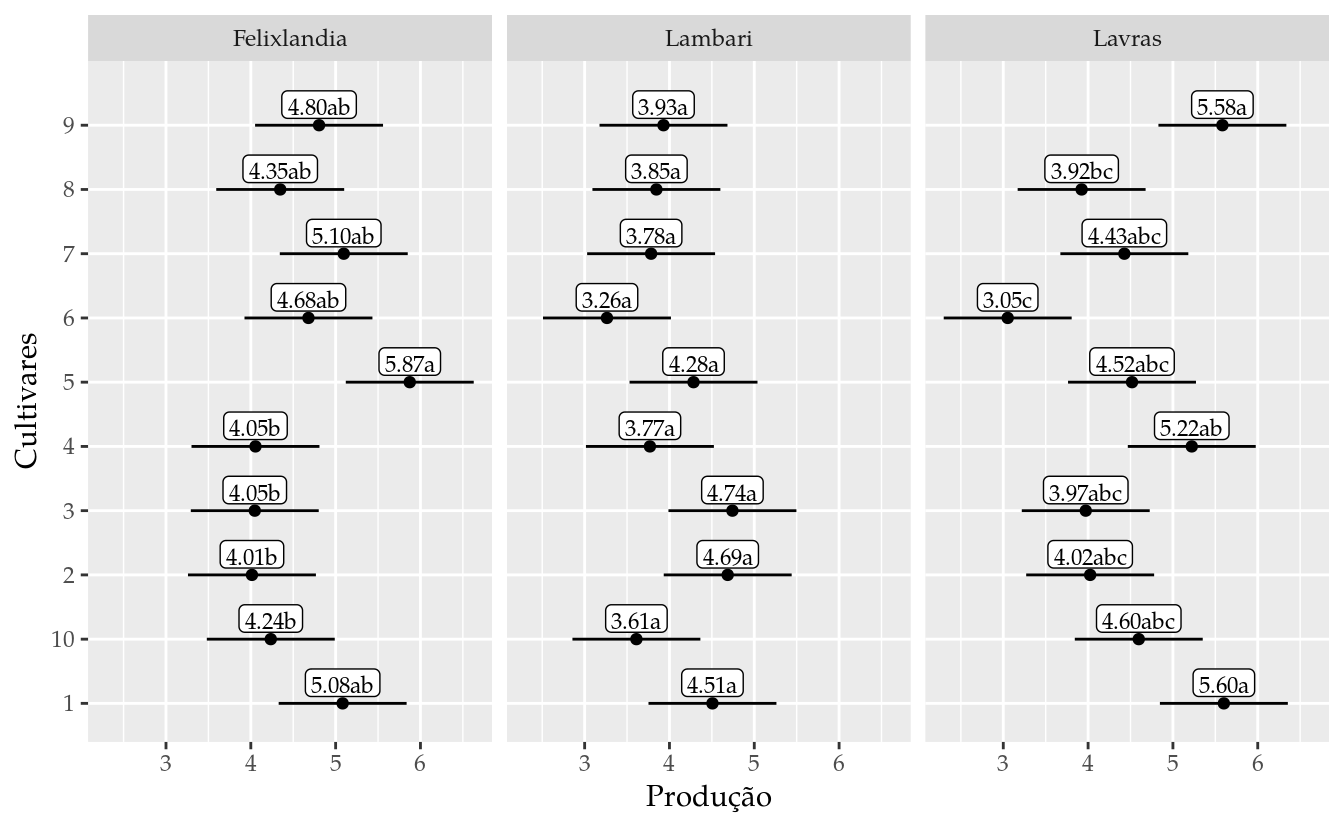
19.2 Experimentos em delineamento de blocos completos
Os dados em BanzattoQd8.2.1 são referentes a um grupo de 4 ensaios de competição de 10 cultivares de batata que foram instalados no delineamento de blocos casualizados com 4 repetições. A variável resposta é a produção de tubérculos.
19.2.1 Análise exploratória
# github.com/pet-estatistica/labestData/blob/devel/data-raw/BanzattoQd8.2.1.txt
# help(BanzattoQd8.2.1, package = "labestData")
da <- labestData::BanzattoQd8.2.1
str(da)## 'data.frame': 160 obs. of 4 variables:
## $ exper: Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 1 ...
## $ bloco: Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 1 ...
## $ cult : Factor w/ 10 levels "Achat","AN-3",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ prod : num 17.2 29.3 37.1 29 27.5 ...xtabs(~exper + cult, data = da)## cult
## exper Achat AN-3 Apuã Baronesa Bintje Carola Elipsa Matilda Ômega
## 1 4 4 4 4 4 4 4 4 4
## 2 4 4 4 4 4 4 4 4 4
## 3 4 4 4 4 4 4 4 4 4
## 4 4 4 4 4 4 4 4 4 4
## cult
## exper Radosa
## 1 4
## 2 4
## 3 4
## 4 4ggplot(data = da,
mapping = aes(x = cult, y = prod, color = bloco)) +
facet_wrap(facets = ~exper, ncol = 1) +
geom_point() +
geom_line(mapping = aes(group = bloco),
color = "gray") +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")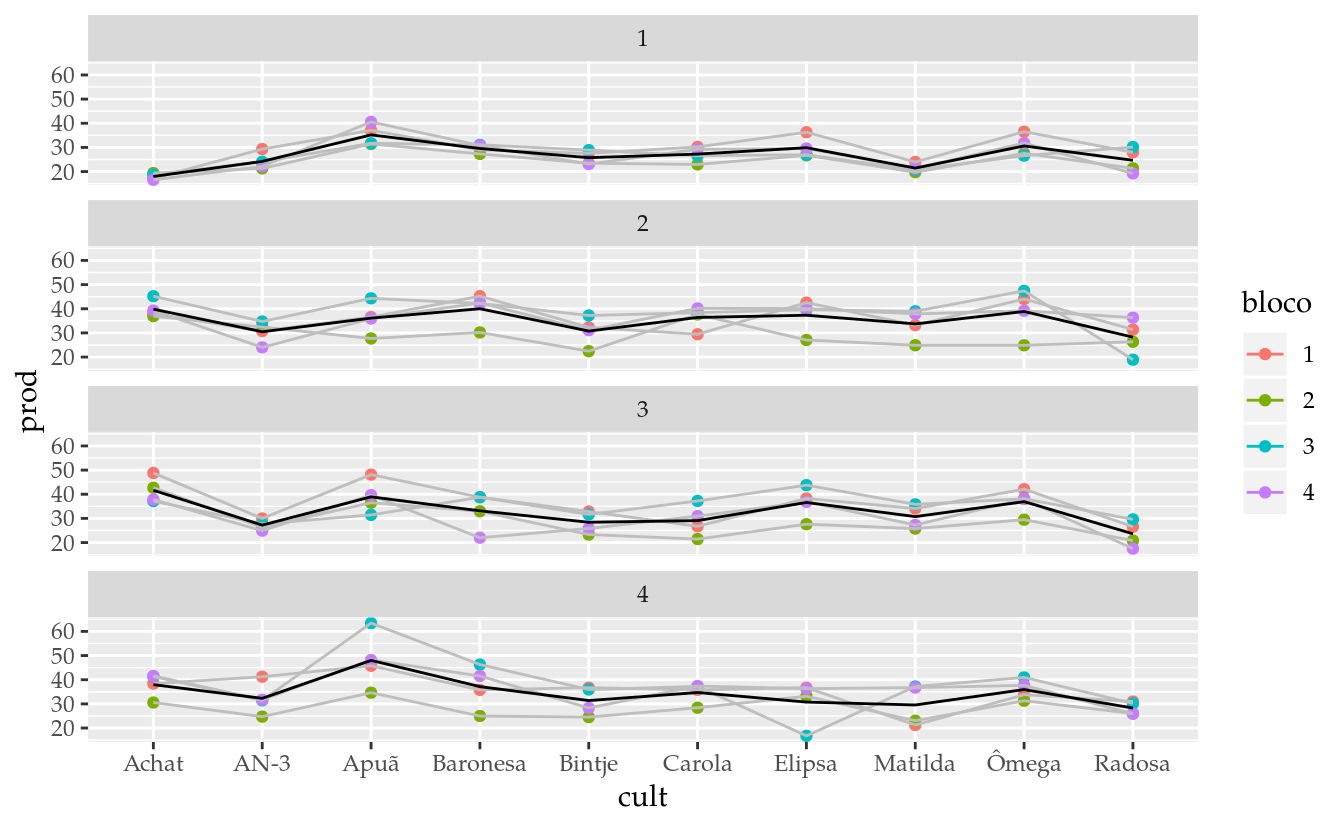
19.2.2 Análises individuais
da_exper <- split(da, f = da$exper)
# Análise para cada exper.
m0_exper <- lapply(da_exper,
FUN = function(data) {
lm(prod ~ bloco + cult, data = data)
})
# Gráficos de normalidade.
par(mfrow = c(2, 2))
sapply(m0_exper, FUN = plot, which = 2)
## $`1`
## NULL
##
## $`2`
## NULL
##
## $`3`
## NULL
##
## $`4`
## NULLlayout(1)
# Quadrados médios.
sapply(m0_exper, FUN = function(m0) deviance(m0)/df.residual(m0))## 1 2 3 4
## 8.696262 27.931909 21.219884 38.139726# Quadro de análise de variância.
lapply(m0_exper, FUN = anova)## $`1`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 146.40 48.799 5.6115 0.004007 **
## cult 9 882.84 98.094 11.2800 4.405e-07 ***
## Residuals 27 234.80 8.696
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## $`2`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 533.99 177.996 6.3725 0.002086 **
## cult 9 628.63 69.848 2.5007 0.031637 *
## Residuals 27 754.16 27.932
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## $`3`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 442.18 147.39 6.9460 0.001299 **
## cult 9 1198.69 133.19 6.2765 9.132e-05 ***
## Residuals 27 572.94 21.22
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## $`4`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 587.29 195.76 5.1328 0.006136 **
## cult 9 1181.95 131.33 3.4433 0.006051 **
## Residuals 27 1029.77 38.14
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Teste de Tukey.
tk <- lapply(m0_exper,
FUN = function(m0) {
tb <- HSD.test(m0, trt = "cult")$groups
tb$groups <- trimws(as.character(tb$groups))
tb <- cbind(cult = rownames(tb), tb)
rownames(tb) <- NULL
return(tb)
})
tk## $`1`
## cult prod groups
## 1 Apuã 35.1975 a
## 2 Ômega 30.6125 ab
## 3 Elipsa 29.8800 ab
## 4 Baronesa 29.5525 ab
## 5 Carola 27.2425 bc
## 6 Bintje 25.7525 bc
## 7 Radosa 24.6725 bcd
## 8 AN-3 24.2150 bcd
## 9 Matilda 21.4525 cd
## 10 Achat 17.9850 d
##
## $`2`
## cult prod groups
## 1 Baronesa 40.0025 a
## 2 Achat 39.8250 a
## 3 Ômega 38.8700 a
## 4 Elipsa 37.3175 a
## 5 Carola 36.4025 a
## 6 Apuã 36.1225 a
## 7 Matilda 33.7375 a
## 8 Bintje 30.7675 a
## 9 AN-3 30.4850 a
## 10 Radosa 28.2400 a
##
## $`3`
## cult prod groups
## 1 Achat 41.6250 a
## 2 Apuã 38.9250 ab
## 3 Ômega 36.9300 abc
## 4 Elipsa 36.6000 abc
## 5 Baronesa 33.1175 abcd
## 6 Matilda 30.7325 abcd
## 7 Carola 29.0800 bcd
## 8 Bintje 28.4050 bcd
## 9 AN-3 27.2275 cd
## 10 Radosa 23.6075 d
##
## $`4`
## cult prod groups
## 1 Apuã 48.0050 a
## 2 Achat 37.9600 ab
## 3 Baronesa 37.1400 ab
## 4 Ômega 35.9275 ab
## 5 Carola 34.6975 ab
## 6 AN-3 32.2825 b
## 7 Bintje 31.3750 b
## 8 Elipsa 30.7425 b
## 9 Matilda 29.5475 b
## 10 Radosa 28.2675 b19.2.3 Análise conjunta
A análise conjunta está considerando efeito fixo para experimento, bloco e cultivar.
Atenção: esteja ciente de que a maior variância residual (experimento 4) é quase 5 vezes maior que a menor variância (experimento 1).
# Modelo com apenas um estrato para verificar pressupostos.
m0 <- lm(terms(prod ~ exper + exper/bloco + cult + exper:cult,
keep.order = TRUE),
data = da)
par(mfrow = c(2, 2))
plot(m0)
layout(1)
# Modelo com dois estratos.
m1 <- aov(prod ~ exper + Error(exper:bloco) + cult + exper:cult,
data = da)
# Quadro de anova.
summary(m1)##
## Error: exper:bloco
## Df Sum Sq Mean Sq F value Pr(>F)
## exper 3 1820 606.6 4.257 0.0289 *
## Residuals 12 1710 142.5
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Error: Within
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 9 2403 267.04 11.128 3.89e-12 ***
## exper:cult 27 1489 55.14 2.298 0.00137 **
## Residuals 108 2592 24.00
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Médias marginais ajustadas por exper.
emm <- emmeans(m1, specs = ~cult | exper)## Note: re-fitting model with sum-to-zero contrastsemm## exper = 1:
## cult emmean SE df lower.CL upper.CL
## Achat 18.0 2.99 60.5 12.0 24.0
## AN-3 24.2 2.99 60.5 18.2 30.2
## Apuã 35.2 2.99 60.5 29.2 41.2
## Baronesa 29.6 2.99 60.5 23.6 35.5
## Bintje 25.8 2.99 60.5 19.8 31.7
## Carola 27.2 2.99 60.5 21.3 33.2
## Elipsa 29.9 2.99 60.5 23.9 35.9
## Matilda 21.5 2.99 60.5 15.5 27.4
## Ômega 30.6 2.99 60.5 24.6 36.6
## Radosa 24.7 2.99 60.5 18.7 30.7
##
## exper = 2:
## cult emmean SE df lower.CL upper.CL
## Achat 39.8 2.99 60.5 33.8 45.8
## AN-3 30.5 2.99 60.5 24.5 36.5
## Apuã 36.1 2.99 60.5 30.1 42.1
## Baronesa 40.0 2.99 60.5 34.0 46.0
## Bintje 30.8 2.99 60.5 24.8 36.8
## Carola 36.4 2.99 60.5 30.4 42.4
## Elipsa 37.3 2.99 60.5 31.3 43.3
## Matilda 33.7 2.99 60.5 27.8 39.7
## Ômega 38.9 2.99 60.5 32.9 44.9
## Radosa 28.2 2.99 60.5 22.3 34.2
##
## exper = 3:
## cult emmean SE df lower.CL upper.CL
## Achat 41.6 2.99 60.5 35.6 47.6
## AN-3 27.2 2.99 60.5 21.2 33.2
## Apuã 38.9 2.99 60.5 32.9 44.9
## Baronesa 33.1 2.99 60.5 27.1 39.1
## Bintje 28.4 2.99 60.5 22.4 34.4
## Carola 29.1 2.99 60.5 23.1 35.1
## Elipsa 36.6 2.99 60.5 30.6 42.6
## Matilda 30.7 2.99 60.5 24.7 36.7
## Ômega 36.9 2.99 60.5 30.9 42.9
## Radosa 23.6 2.99 60.5 17.6 29.6
##
## exper = 4:
## cult emmean SE df lower.CL upper.CL
## Achat 38.0 2.99 60.5 32.0 43.9
## AN-3 32.3 2.99 60.5 26.3 38.3
## Apuã 48.0 2.99 60.5 42.0 54.0
## Baronesa 37.1 2.99 60.5 31.2 43.1
## Bintje 31.4 2.99 60.5 25.4 37.4
## Carola 34.7 2.99 60.5 28.7 40.7
## Elipsa 30.7 2.99 60.5 24.8 36.7
## Matilda 29.5 2.99 60.5 23.6 35.5
## Ômega 35.9 2.99 60.5 29.9 41.9
## Radosa 28.3 2.99 60.5 22.3 34.3
##
## Warning: EMMs are biased unless design is perfectly balanced
## Confidence level used: 0.95# Testes de Tukey por exper da análise conjunta.
# ATTENTION: assume balanceamento.
tk <-
lapply(da_exper,
FUN = function(data) {
rdf <- df.residual(m0)
rms <- deviance(m0)/rdf
tb <- HSD.test(y = data$prod,
trt = data$cult,
DFerror = rdf,
MSerror = rms)
tb <- tb$groups
tb$groups <- trimws(as.character(tb$groups))
names(tb)[1] <- "prod"
cbind(cult = rownames(tb), tb,
stringsAsFactors = FALSE)
})
# tk
# Junta para ter os intervalos de confiança.
tk <- bind_rows(tk, .id = "exper")
tk <- inner_join(as.data.frame(emm), tk,
by = c("cult", "exper"))
tk[, c(1, 2, 8, 6:7, 9)] %>%
arrange(exper, desc(prod))## cult exper prod lower.CL upper.CL groups
## 1 Apuã 1 35.1975 29.21047 41.18453 a
## 2 Ômega 1 30.6125 24.62547 36.59953 ab
## 3 Elipsa 1 29.8800 23.89297 35.86703 ab
## 4 Baronesa 1 29.5525 23.56547 35.53953 ab
## 5 Carola 1 27.2425 21.25547 33.22953 abc
## 6 Bintje 1 25.7525 19.76547 31.73953 abc
## 7 Radosa 1 24.6725 18.68547 30.65953 abc
## 8 AN-3 1 24.2150 18.22797 30.20203 abc
## 9 Matilda 1 21.4525 15.46547 27.43953 bc
## 10 Achat 1 17.9850 11.99797 23.97203 c
## 11 Baronesa 2 40.0025 34.01547 45.98953 a
## 12 Achat 2 39.8250 33.83797 45.81203 a
## 13 Ômega 2 38.8700 32.88297 44.85703 ab
## 14 Elipsa 2 37.3175 31.33047 43.30453 ab
## 15 Carola 2 36.4025 30.41547 42.38953 ab
## 16 Apuã 2 36.1225 30.13547 42.10953 ab
## 17 Matilda 2 33.7375 27.75047 39.72453 ab
## 18 Bintje 2 30.7675 24.78047 36.75453 ab
## 19 AN-3 2 30.4850 24.49797 36.47203 ab
## 20 Radosa 2 28.2400 22.25297 34.22703 b
## 21 Achat 3 41.6250 35.63797 47.61203 a
## 22 Apuã 3 38.9250 32.93797 44.91203 ab
## 23 Ômega 3 36.9300 30.94297 42.91703 abc
## 24 Elipsa 3 36.6000 30.61297 42.58703 abc
## 25 Baronesa 3 33.1175 27.13047 39.10453 abcd
## 26 Matilda 3 30.7325 24.74547 36.71953 abcd
## 27 Carola 3 29.0800 23.09297 35.06703 bcd
## 28 Bintje 3 28.4050 22.41797 34.39203 bcd
## 29 AN-3 3 27.2275 21.24047 33.21453 cd
## 30 Radosa 3 23.6075 17.62047 29.59453 d
## 31 Apuã 4 48.0050 42.01797 53.99203 a
## 32 Achat 4 37.9600 31.97297 43.94703 ab
## 33 Baronesa 4 37.1400 31.15297 43.12703 ab
## 34 Ômega 4 35.9275 29.94047 41.91453 b
## 35 Carola 4 34.6975 28.71047 40.68453 b
## 36 AN-3 4 32.2825 26.29547 38.26953 b
## 37 Bintje 4 31.3750 25.38797 37.36203 b
## 38 Elipsa 4 30.7425 24.75547 36.72953 b
## 39 Matilda 4 29.5475 23.56047 35.53453 b
## 40 Radosa 4 28.2675 22.28047 34.25453 b# NOTE: para ordenar os níveis de cultivar dentro de cada experimento.
tk <- tk %>%
mutate(combo = {
f <- interaction(exper, cult, drop = TRUE)
reorder(f, prod)
})
# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = tk,
mapping = aes(x = combo, y = prod)) +
facet_wrap(facets = ~exper,
nrow = 1,
scales = "free_x") +
geom_point() +
geom_errorbar(mapping = aes(ymin = lower.CL, ymax = upper.CL),
width = 0) +
scale_x_discrete(breaks = levels(tk$combo),
labels = gsub("^.*\\.", "", levels(tk$combo))) +
geom_text(mapping = aes(label = sprintf("%0.1f %s", prod, groups)),
size = 3,
angle = 90,
vjust = -0.5) +
labs(y = "Produção",
x = "Cultivares") +
expand_limits(x = c(-0.5, NA)) +
theme(axis.text.x = element_text(angle = 90,
vjust = 0.5,
hjust = 1))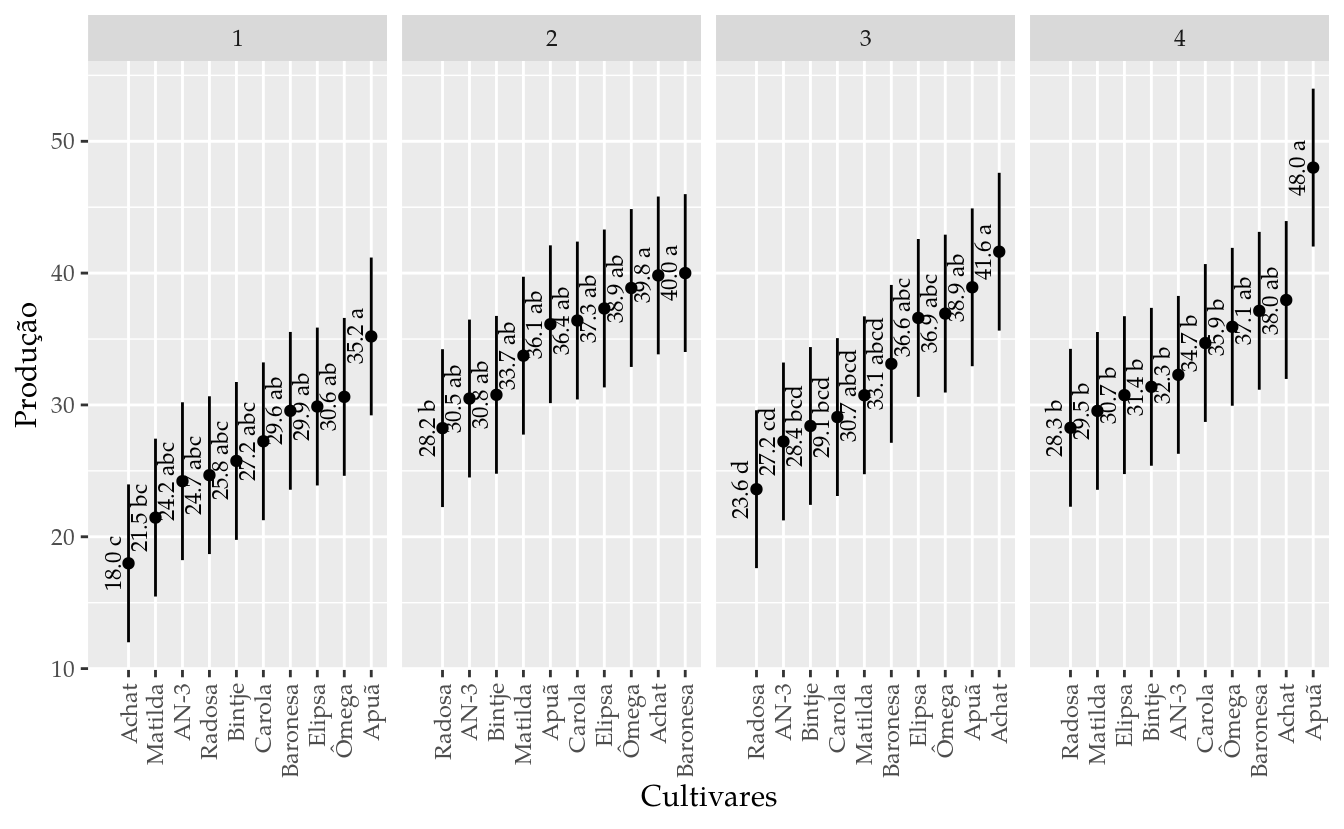
19.3 Experimentos com tratamentos comuns
# github.com/pet-estatistica/labestData/blob/devel/data-raw/ZimmermannTb12.19.txt
# help(ZimmermannTb12.19, package = "labestData")
da <- rbind(cbind(labestData::ZimmermannTb12.19, exper = 1),
cbind(labestData::ZimmermannTb12.20, exper = 2))
da$exper <- factor(da$exper)
da$prod <- da$prod/1000
str(da)## 'data.frame': 72 obs. of 4 variables:
## $ cult : Factor w/ 15 levels "CNFP 8015","CNFP 8016",..: 1 2 3 4 5 6 7 8 9 1 ...
## $ bloco: Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 2 ...
## $ prod : num 3.7 3.6 3.92 3.88 4.2 ...
## $ exper: Factor w/ 2 levels "1","2": 1 1 1 1 1 1 1 1 1 1 ...xtabs(~cult + exper, data = da)## exper
## cult 1 2
## CNFP 8015 4 0
## CNFP 8016 4 0
## CNFP 8017 4 0
## CNFP 8018 4 0
## CNFP 8019 4 0
## CNFP 8020 4 0
## Diamante Negro 4 4
## FT Nobre 4 4
## G. Brilhante 4 4
## CNFP 8021 0 4
## CNFP 8022 0 4
## CNFP 8023 0 4
## CNFP 8024 0 4
## CNFP 8025 0 4
## CNFP 8026 0 4ggplot(data = da,
mapping = aes(x = cult, y = prod, color = bloco)) +
facet_wrap(facets = ~exper, ncol = 1) +
geom_point() +
geom_line(mapping = aes(group = bloco),
color = "gray") +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean") +
theme(axis.text.x = element_text(angle = 45, hjust = 1))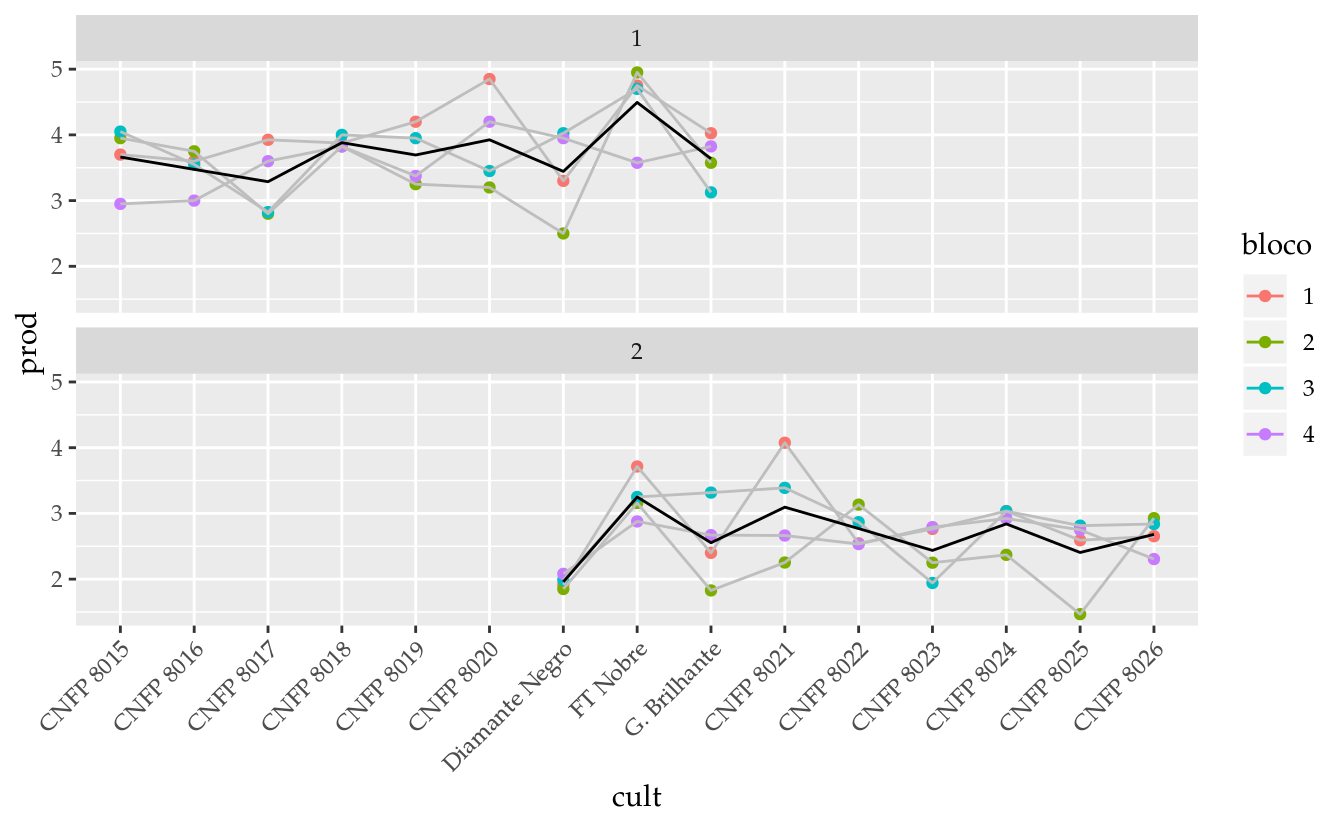
19.3.1 Análises individuais
da_exper <- split(da, f = da$exper)
# Análise para cada exper.
m0_exper <- lapply(da_exper,
FUN = function(data) {
lm(prod ~ bloco + cult, data = data)
})
# Gráficos de normalidade.
par(mfrow = c(1, 2))
sapply(m0_exper, FUN = plot, which = 2)
## $`1`
## NULL
##
## $`2`
## NULLlayout(1)
# Quadrados médios.
sapply(m0_exper, FUN = function(m0) deviance(m0)/df.residual(m0))## 1 2
## 0.2563194 0.1899621# Quadro de análise de variância.
lapply(m0_exper, FUN = anova)## $`1`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 1.3096 0.43653 1.7031 0.19305
## cult 8 4.0035 0.50043 1.9524 0.09805 .
## Residuals 24 6.1517 0.25632
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## $`2`
## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 3 1.4150 0.47168 2.4830 0.08515 .
## cult 8 4.8013 0.60017 3.1594 0.01369 *
## Residuals 24 4.5591 0.18996
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Teste de Tukey.
tk <- lapply(m0_exper,
FUN = function(m0) {
tb <- HSD.test(m0, trt = "cult")$groups
tb$groups <- trimws(as.character(tb$groups))
tb <- cbind(cult = rownames(tb), tb)
rownames(tb) <- NULL
return(tb)
})
tk## $`1`
## cult prod groups
## 1 FT Nobre 4.49375 a
## 2 CNFP 8020 3.92500 a
## 3 CNFP 8018 3.88125 a
## 4 CNFP 8019 3.69375 a
## 5 CNFP 8015 3.66250 a
## 6 G. Brilhante 3.63750 a
## 7 CNFP 8016 3.47500 a
## 8 Diamante Negro 3.44375 a
## 9 CNFP 8017 3.28750 a
##
## $`2`
## cult prod groups
## 1 FT Nobre 3.250000 a
## 2 CNFP 8021 3.095140 a
## 3 CNFP 8024 2.840275 ab
## 4 CNFP 8022 2.769443 ab
## 5 CNFP 8026 2.681253 ab
## 6 G. Brilhante 2.553473 ab
## 7 CNFP 8023 2.436808 ab
## 8 CNFP 8025 2.405558 ab
## 9 Diamante Negro 1.958335 b19.3.2 Análise conjunta
A análise conjunta está considerando efeito fixo para experimento, bloco e cultivar.
# Modelo com apenas um estrato para verificar pressupostos.
m0 <- lm(terms(prod ~ exper + exper/bloco + cult + exper:cult,
keep.order = TRUE),
data = da)
par(mfrow = c(2, 2))
plot(m0)
layout(1)
# Modelo com dois estratos.
m1 <- aov(prod ~ exper + Error(exper:bloco) + cult + exper:cult,
data = da)
# Quadro de anova.
summary(m1)##
## Error: exper:bloco
## Df Sum Sq Mean Sq F value Pr(>F)
## exper 1 20.097 20.097 44.26 0.000557 ***
## Residuals 6 2.725 0.454
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Error: Within
## Df Sum Sq Mean Sq F value Pr(>F)
## cult 14 8.641 0.6172 2.766 0.00444 **
## exper:cult 2 0.163 0.0817 0.366 0.69540
## Residuals 48 10.711 0.2231
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Abandona a interação não significativa para ter todos os efeitos
# estimados.
m1 <- aov(prod ~ exper + Error(exper:bloco) + cult,
data = da)
# Médias marginais ajustadas do modelo de 2 estratos.
# emm <- emmeans(m1, specs = ~cult)
# as.data.frame(emm)
# Volta para o modelo de um estrato.
m0 <- lm(prod ~ exper + exper:bloco + cult,
data = da)
emm <- emmeans(m0, specs = ~cult)## NOTE: A nesting structure was detected in the fitted model:
## bloco %in% experas.data.frame(emm)## cult emmean SE df lower.CL upper.CL
## 1 CNFP 8015 3.026968 0.2518577 50 2.521097 3.532839
## 2 CNFP 8016 2.839468 0.2518577 50 2.333597 3.345339
## 3 CNFP 8017 2.651968 0.2518577 50 2.146097 3.157839
## 4 CNFP 8018 3.245718 0.2518577 50 2.739847 3.751589
## 5 CNFP 8019 3.058218 0.2518577 50 2.552347 3.564089
## 6 CNFP 8020 3.289468 0.2518577 50 2.783597 3.795339
## 7 Diamante Negro 2.701042 0.1648796 50 2.369872 3.032213
## 8 FT Nobre 3.871875 0.1648796 50 3.540705 4.203045
## 9 G. Brilhante 3.095486 0.1648796 50 2.764316 3.426657
## 10 CNFP 8021 3.730672 0.2518577 50 3.224801 4.236543
## 11 CNFP 8022 3.404975 0.2518577 50 2.899104 3.910846
## 12 CNFP 8023 3.072340 0.2518577 50 2.566469 3.578211
## 13 CNFP 8024 3.475807 0.2518577 50 2.969936 3.981678
## 14 CNFP 8025 3.041090 0.2518577 50 2.535219 3.546961
## 15 CNFP 8026 3.316785 0.2518577 50 2.810914 3.822656grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid[[1]]
# glht(m0, linfct = mcp(cult = "Tukey"))
results <- wzRfun::apmc(L,
model = m0,
focus = "cult",
test = "fdr") %>%
mutate(cld = wzRfun::ordered_cld(let = cld, means = fit))
results## cult fit lwr upr cld
## 1 CNFP 8015 3.026968 2.521097 3.532839 ac
## 2 CNFP 8016 2.839468 2.333597 3.345339 bc
## 3 CNFP 8017 2.651968 2.146097 3.157839 bc
## 4 CNFP 8018 3.245718 2.739847 3.751589 ac
## 5 CNFP 8019 3.058218 2.552347 3.564089 ac
## 6 CNFP 8020 3.289468 2.783597 3.795339 ac
## 7 Diamante Negro 2.701042 2.369872 3.032213 c
## 8 FT Nobre 3.871875 3.540705 4.203045 a
## 9 G. Brilhante 3.095486 2.764316 3.426657 bc
## 10 CNFP 8021 3.730672 3.224801 4.236543 ab
## 11 CNFP 8022 3.404975 2.899104 3.910846 ac
## 12 CNFP 8023 3.072340 2.566469 3.578211 ac
## 13 CNFP 8024 3.475807 2.969936 3.981678 ac
## 14 CNFP 8025 3.041090 2.535219 3.546961 ac
## 15 CNFP 8026 3.316785 2.810914 3.822656 ac# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results,
mapping = aes(x = reorder(cult, fit), y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = -0.5) +
labs(y = "Produção",
x = "Cultivares") +
expand_limits(x = c(-0.5, NA)) +
theme(axis.text.x = element_text(angle = 90,
vjust = 0.5,
hjust = 1))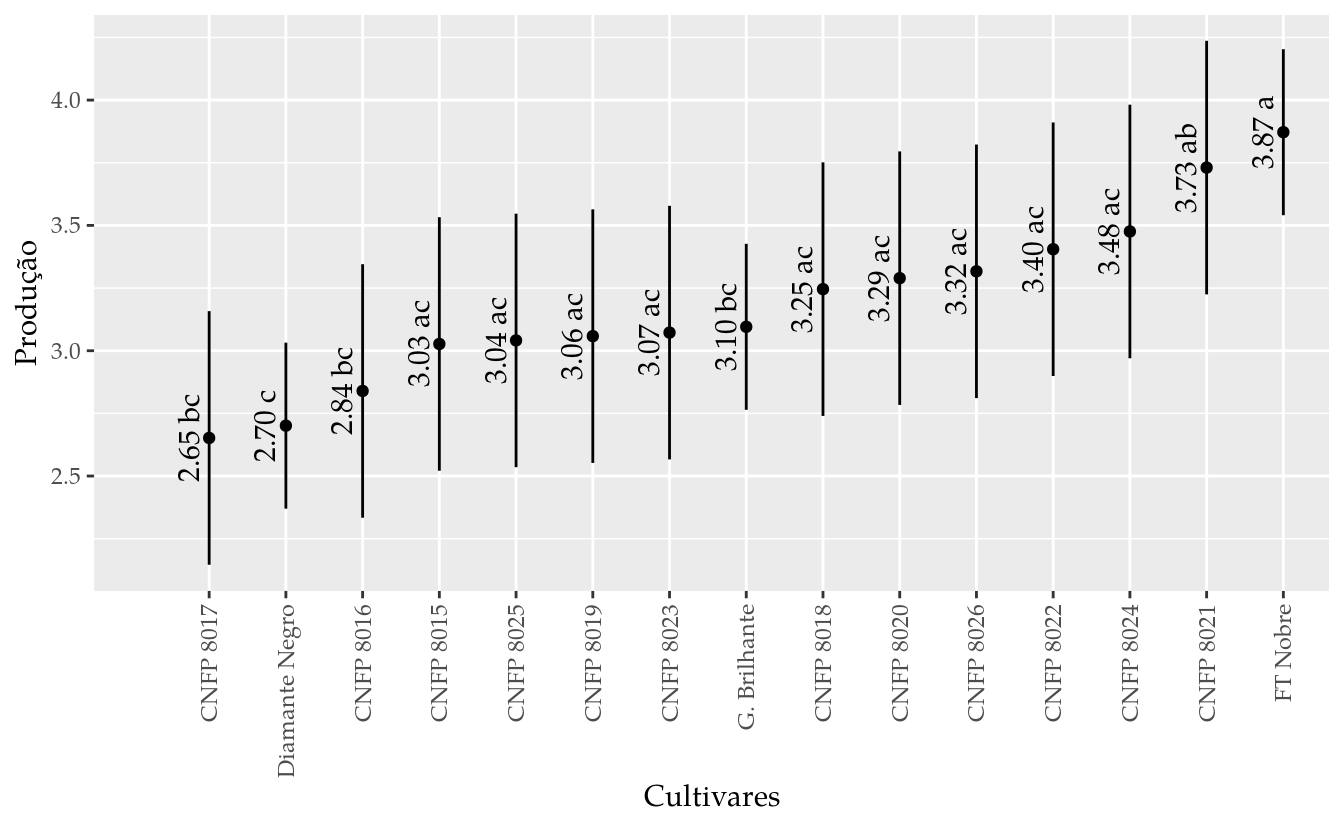
19.4 Experimentos ao longo do tempo
TODO as ser feito: experimento em várias safras. E em vários locais e safras.
ATTENTION: essa análise precisa ser refeita nos moldes de uma análise de experimento de medidas repetidas pois, visto que a cultura é perene, a mesma unidade experimental é medida ao longo do tempo. Se fosse uma cultura anual, cada safra seria um novo cultivo onde as unidades poderiam ser todas diferentes e o experimento ser em outras instalações/condições. Poderia ser considerado medida repetida apenas se as mesmas parcelas recebessem os mesmos tratamentos ao longo do tempo na mesma área experimental.
# github.com/pet-estatistica/labestData/blob/devel/data-raw/RamalhoTb8.12.txt
# help(RamalhoTb8.12, package = "labestData")
da <- labestData::RamalhoTb8.12
str(da)## 'data.frame': 120 obs. of 4 variables:
## $ prog: Factor w/ 10 levels "1","2","3","4",..: 1 1 1 2 2 2 3 3 3 4 ...
## $ ano : Ord.factor w/ 3 levels "1"<"2"<"3": 1 2 3 1 2 3 1 2 3 1 ...
## $ bloc: Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 1 1 ...
## $ prod: num 4.3 7.62 3.11 13.2 7.7 ...xtabs(~ano + prog, data = da)## prog
## ano 1 2 3 4 5 6 7 8 9 10
## 1 4 4 4 4 4 4 4 4 4 4
## 2 4 4 4 4 4 4 4 4 4 4
## 3 4 4 4 4 4 4 4 4 4 4ggplot(data = da,
mapping = aes(x = prog, y = prod, color = bloc)) +
facet_wrap(facets = ~ano, ncol = 1) +
geom_point() +
geom_line(mapping = aes(group = bloc),
color = "gray") +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")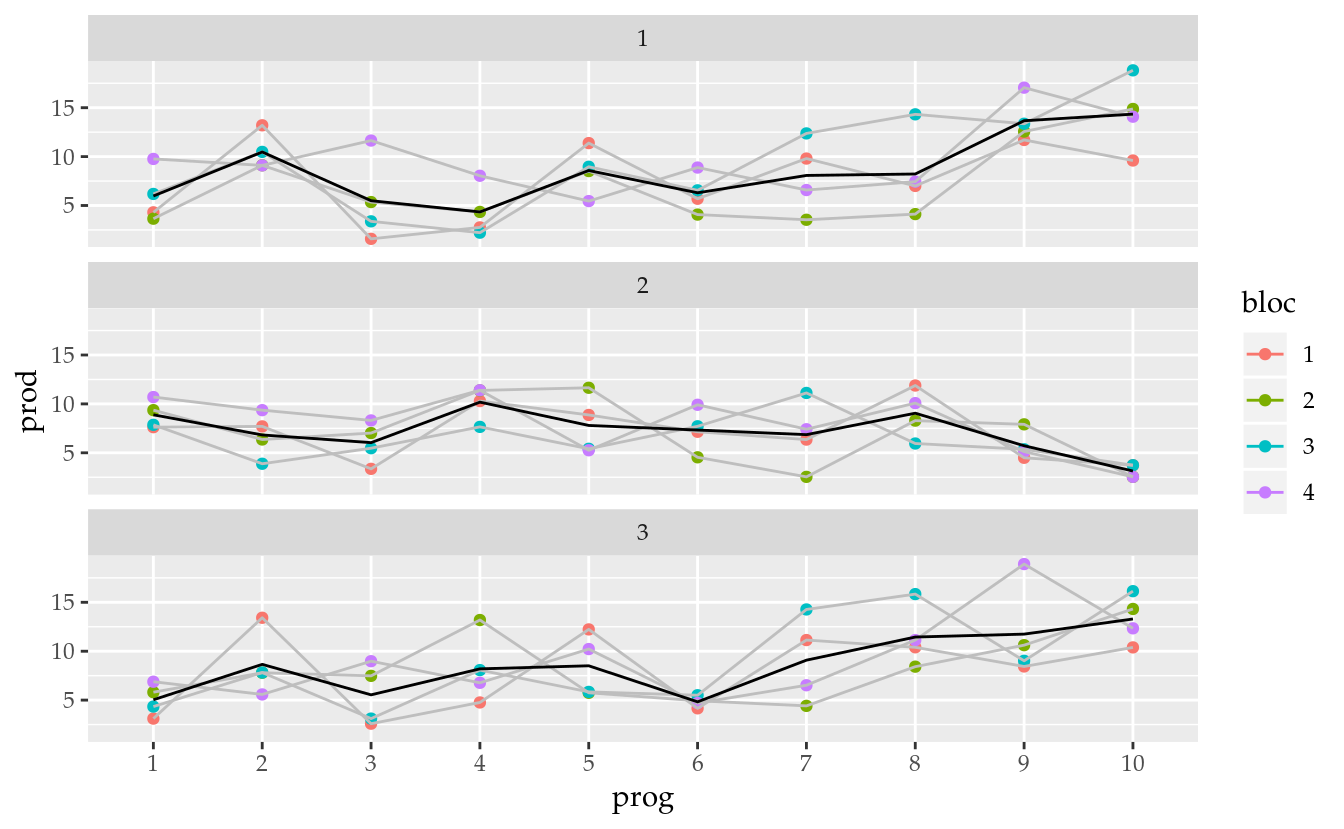
# NOTE: o comportamento bianual de produção de café.
ggplot(data = da,
mapping = aes(x = ano, y = prod)) +
facet_wrap(facets = ~prog, nrow = 1) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")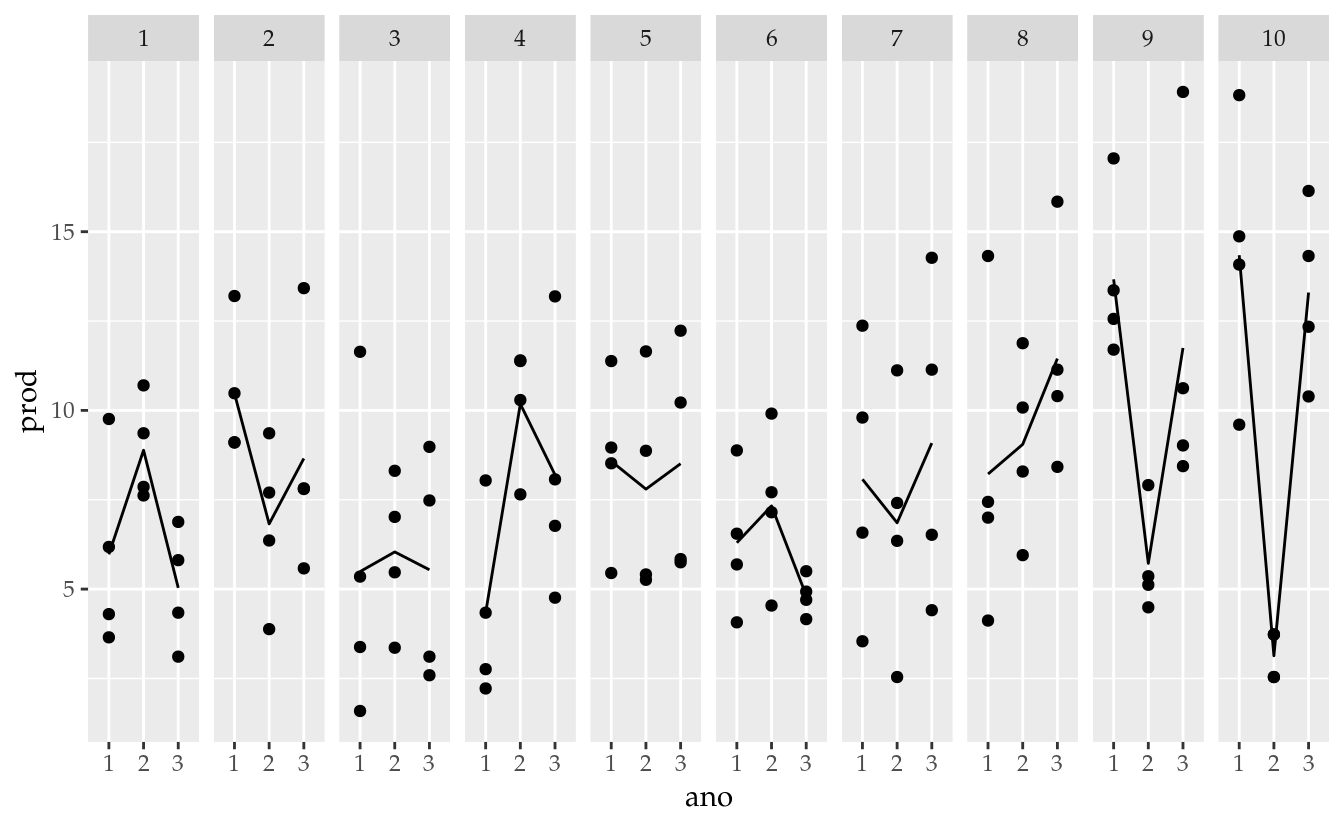
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani