Capítulo 13 Blocos incompletos balanceados
TODO:
- http://home.iitk.ac.in/~shalab/anova/chapter5-anova-incomplete-block-designs.pdf.
- https://stat.ethz.ch/education/semesters/as2015/anova/09_Incomplete_Blocks.pdf.
- http://www.maths.qmul.ac.uk/~rab/DOEbook/doeweb11.pdf.

Figura 13.1: Ilustração de um experimento em delineamento de blocos incompletos balanceados (tipo 3). As 10 pessoas são os blocos. Cada pessoa avalia 3 tratamentos. Os avatares foram extraídos de www.freepik.com. Este delineamento possui \(t = 6\) tratamentos, bloco de tamanho \(k = 3\), \(r = 5\) repetições, \(b = 10\) blocos, \(\lambda = 2\) ocorrências simultâneas de cada par e eficiência de 0,8.
13.1 Análise exploratória
library(agricolae) # BIB.test() e design.bib().
library(emmeans) # emmeans().
library(multcomp) # glht().
library(lme4) # lmer().
library(lmerTest) # anova() para classe lmerMod.
library(tidyverse) # Manipulação e visualização.
# Funções.
source("mpaer_functions.R")# Dados contidos no labestData.
da <- as_tibble(labestData::PimentelPg185)
str(da)## Classes 'tbl_df', 'tbl' and 'data.frame': 30 obs. of 3 variables:
## $ bloc: Factor w/ 10 levels "1","2","3","4",..: 1 1 1 2 2 2 3 3 3 4 ...
## $ trat: Factor w/ 5 levels "1","2","3","4",..: 1 2 3 1 2 4 1 2 5 1 ...
## $ y : int 35 28 27 30 20 22 28 16 18 36 ...# # Caso não tenha o pacote labestData, leia do txt no repositório.
# url <- paste0("https://raw.githubusercontent.com/pet-estatistica/",
# "labestData/devel/data-raw/PimentelPg185.txt")
# da <- read_tsv(url) %>%
# mutate(bloc = factor(bloc, levels = unique(sort(bloc))),
# trat = factor(trat, levels = unique(sort(trat))))
# São 6 repetições de cada tratamento.
# O tamanho do bloco é 3.
addmargins(xtabs(~trat + bloc, data = da))## bloc
## trat 1 2 3 4 5 6 7 8 9 10 Sum
## 1 1 1 1 1 1 1 0 0 0 0 6
## 2 1 1 1 0 0 0 1 1 1 0 6
## 3 1 0 0 1 1 0 1 1 0 1 6
## 4 0 1 0 1 0 1 1 0 1 1 6
## 5 0 0 1 0 1 1 0 1 1 1 6
## Sum 3 3 3 3 3 3 3 3 3 3 30# Quantas vezes cada par ocorre junto.
by(data = da$trat,
INDICES = da$bloc,
FUN = function(x) {
apply(combn(sort(x), m = 2),
MARGIN = 2,
FUN = paste0,
collapse = "_")
}) %>%
flatten_chr() %>%
table()## .
## 1_2 1_3 1_4 1_5 2_3 2_4 2_5 3_4 3_5 4_5
## 3 3 3 3 3 3 3 3 3 3# Obtém as coordenadas radiais para gráfico de conexão.
da <- da %>%
do({
k <- nlevels(.$trat)
a <- seq(0, 2 * pi, length.out = k + 1)[-(k + 1)]
cbind(.,
data.frame(coord_x = sin(a)[as.integer(.$trat)],
coord_y = cos(a)[as.integer(.$trat)]))
})
# Gráfico de conexão.
ext <- c(-1.2, 1.2)
ggplot(data = da,
mapping = aes(coord_x, coord_y)) +
facet_wrap(facets = ~bloc, nrow = 2) +
geom_point() +
geom_polygon(fill = NA, color = "orange") +
geom_label(mapping = aes(label = trat)) +
expand_limits(x = ext, y = ext) +
coord_equal()
#-----------------------------------------------------------------------
# Visualização dos dados.
ggplot(data = da,
mapping = aes(x = trat, y = y, color = bloc)) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean") +
geom_text(mapping = aes(label = bloc),
hjust = 0,
nudge_x = 0.02,
show.legend = FALSE)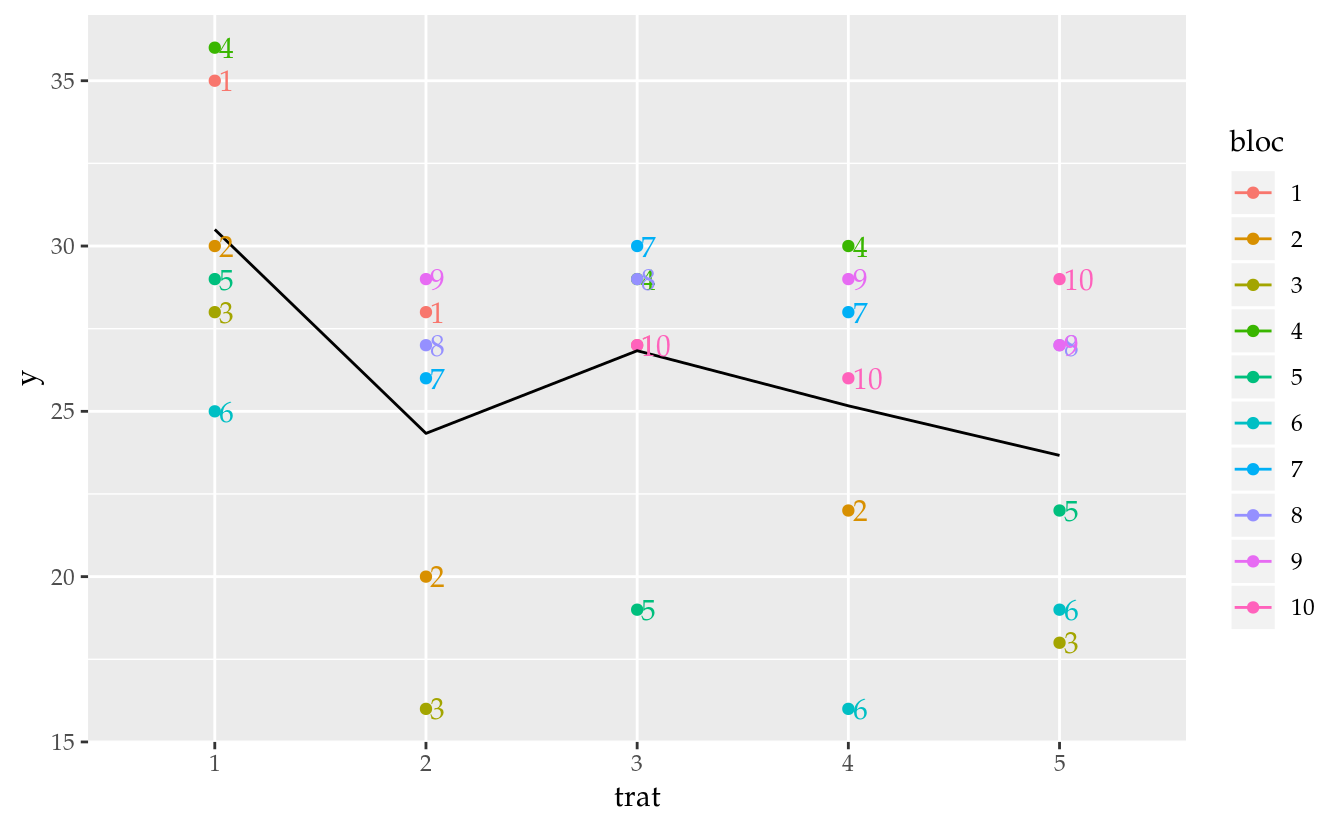
# ggplot(data = da,
# mapping = aes(x = trat, y = y)) +
# facet_wrap(facets = ~bloc) +
# geom_point()library(igraph)
edg <- by(data = da$trat,
INDICES = da$bloc,
FUN = combn,
m = 2) %>%
flatten_int()
ghp <- graph(edg, directed = FALSE)
plot(ghp,
layout = layout_in_circle,
edge.curved = FALSE)13.2 Modelo de efeito fixo de bloco
O modelo estatístico quando se considera efeito fixo para todos os termos experimentais é representado por \[ \begin{aligned} Y | i, j &\sim \text{Normal}(\mu_{ij}, \sigma^2)\\ \mu_{ij} &= \mu + \tau_i + \gamma_j \end{aligned} \] em que \(Y\) é o valor observado da variável resposta, \(\mu_{ij}\) é a média condicional o nível \(i\) de tratamento e \(j\) de bloco. \(\tau_i\) acomoda o efeito dos tratamentos e \(\gamma_j\) o efeito de blocos.
Os parâmetros desse modelo são estimados pelo método de mínimos quadrados (ordinários) por meio da função lm().
# Ajuste do modelo de efeitos fixos.
m0 <- lm(y ~ bloc + trat, data = da)
# Análise dos pressupostos.
par(mfrow = c(2, 2)); plot(m0); layout(1)
# Quadro de análise de variância (hipóteses sequenciais).
anova(m0)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 9 412.70 45.856 21.383 2.746e-07 ***
## trat 4 283.69 70.922 33.072 1.495e-07 ***
## Residuals 16 34.31 2.144
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Troca a ordem dos termos na fórmula para ver que as SQ mudam, ou seja,
# os efeitos não são ortogonais.
anova(lm(y ~ trat + bloc, data = da))## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## trat 4 178.87 44.717 20.852 3.473e-06 ***
## bloc 9 517.52 57.502 26.815 5.342e-08 ***
## Residuals 16 34.31 2.144
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Quadro de análise de variância (hipóteses marginais).
# drop1(m0, test = "F")
car::Anova(m0)## Anova Table (Type II tests)
##
## Response: y
## Sum Sq Df F value Pr(>F)
## bloc 517.52 9 26.815 5.342e-08 ***
## trat 283.69 4 33.072 1.495e-07 ***
## Residuals 34.31 16
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos efeitos e medidas de ajuste.
summary(m0)##
## Call:
## lm(formula = y ~ bloc + trat, data = da)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.0222 -0.9556 -0.1333 1.1167 1.6889
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 35.7556 1.0004 35.742 < 2e-16 ***
## bloc2 -5.9111 1.2349 -4.787 0.000202 ***
## bloc3 -9.4444 1.2349 -7.648 9.89e-07 ***
## bloc4 1.3778 1.2349 1.116 0.281022
## bloc5 -7.1556 1.2349 -5.795 2.74e-05 ***
## bloc6 -10.4000 1.2729 -8.170 4.21e-07 ***
## bloc7 0.7778 1.2349 0.630 0.537691
## bloc8 0.2444 1.2349 0.198 0.845578
## bloc9 1.0000 1.2729 0.786 0.443573
## bloc10 -0.3778 1.2729 -0.297 0.770447
## trat2 -9.2000 0.9262 -9.933 3.01e-08 ***
## trat3 -8.0667 0.9262 -8.710 1.81e-07 ***
## trat4 -8.3333 0.9262 -8.998 1.17e-07 ***
## trat5 -7.7333 0.9262 -8.350 3.17e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.464 on 16 degrees of freedom
## Multiple R-squared: 0.953, Adjusted R-squared: 0.9149
## F-statistic: 24.98 on 13 and 16 DF, p-value: 3.72e-08Como visto pelo quadro de análise de variância que as somas de quadrados sequenciais se alteram com a ordem dos termos, os efeitos de bloco e tratamento não são ortogonais no delineamento de blocos incompletos balanceados. Podemos comprovar isso por meio das matrizes de projeção no espaço de cada termo do modelo.
# Matrizes de delineamento para cada termo.
X_mu <- model.matrix(~1, data = da)
X_bl <- model.matrix(~1 + bloc, data = da)
X_vr <- model.matrix(~1 + trat, data = da)
# Matrizes de projeção para cada termo. `proj()` está definida em
# `mpaer-functions.R`.
H_mu <- proj(X_mu)
H_bl <- proj(X_bl)
H_vr <- proj(X_vr)
# Serão ortogonais se a matriz for n x n de 0.
P <- (H_bl - H_mu) %*% (H_vr - H_mu)
all(round(P, digits = 3) == 0)## [1] FALSE# image(P, asp = 1, x = 1:nrow(P), y = 1:ncol(P))A matrix P de dimensão \(n \times n\) não é uma matriz de zeros. Dessa forma, tem-se a comprovação de que os efeitos mencionados não são ortogonais. No caso de serem, os quadros de análise de variância sequencial e marginal seriam idênticos.
# Médias ajustadas marginais.
emm <- emmeans(m0, specs = ~trat)
emm## trat emmean SE df lower.CL upper.CL
## 1 32.8 0.644 16 31.4 34.1
## 2 23.6 0.644 16 22.2 24.9
## 3 24.7 0.644 16 23.3 26.1
## 4 24.4 0.644 16 23.1 25.8
## 5 25.0 0.644 16 23.7 26.4
##
## Results are averaged over the levels of: bloc
## Confidence level used: 0.95# Extração da matriz de funções lineares.
L <- attr(emm, "linfct")
grid <- attr(emm, "grid")
rownames(L) <- grid[[1]]
# Entenda como são obtidas as médias marginais.
MASS::fractions(t(L))## 1 2 3 4 5
## (Intercept) 1 1 1 1 1
## bloc2 1/10 1/10 1/10 1/10 1/10
## bloc3 1/10 1/10 1/10 1/10 1/10
## bloc4 1/10 1/10 1/10 1/10 1/10
## bloc5 1/10 1/10 1/10 1/10 1/10
## bloc6 1/10 1/10 1/10 1/10 1/10
## bloc7 1/10 1/10 1/10 1/10 1/10
## bloc8 1/10 1/10 1/10 1/10 1/10
## bloc9 1/10 1/10 1/10 1/10 1/10
## bloc10 1/10 1/10 1/10 1/10 1/10
## trat2 0 1 0 0 0
## trat3 0 0 1 0 0
## trat4 0 0 0 1 0
## trat5 0 0 0 0 1# Médias ajustadas marginais.
L %*% coef(m0)## [,1]
## 1 32.76667
## 2 23.56667
## 3 24.70000
## 4 24.43333
## 5 25.03333# Matriz de covariância para as médias marginais.
vcov_m0 <- L %*% vcov(m0) %*% t(L)
vcov_m0## 1 2 3 4 5
## 1 0.4145926 -0.0142963 -0.0142963 -0.0142963 -0.0142963
## 2 -0.0142963 0.4145926 -0.0142963 -0.0142963 -0.0142963
## 3 -0.0142963 -0.0142963 0.4145926 -0.0142963 -0.0142963
## 4 -0.0142963 -0.0142963 -0.0142963 0.4145926 -0.0142963
## 5 -0.0142963 -0.0142963 -0.0142963 -0.0142963 0.4145926# Erro padrão de um contraste entre médias ajustadas marginais.
sqrt(vcov_m0[1, 1] + vcov_m0[2, 2] - 2 * vcov_m0[1, 2])## [1] 0.9261629IMPORTANT: a matriz de covariância das médias ajustadas marginais não é diagonal. Ou seja, não são estimativas independentes. Dessa forma, o erro padrão do contraste irá considerar a covariância entre tais estimativas. A covariância entre médias vem da não ortogonalidade entre os efeitos de bloco e tratamento. Como a covariância foi negativa, ela irá aumentar a variância do contraste. Lembre-se que a variância da diferença entre médias é \[ \text{Var}(\hat{\mu}_1 - \hat{\mu}_2) = \text{Var}(\hat{\mu}_1) + \text{Var}(\hat{\mu}_2) - 2 \cdot \text{Cov}(\hat{\mu}_1, \hat{\mu}_2). \]
# Constrastes par a par, ou seja, contrastes de Tukey.
contrast(emm, method = "tukey")## contrast estimate SE df t.ratio p.value
## 1 - 2 9.200 0.926 16 9.933 <.0001
## 1 - 3 8.067 0.926 16 8.710 <.0001
## 1 - 4 8.333 0.926 16 8.998 <.0001
## 1 - 5 7.733 0.926 16 8.350 <.0001
## 2 - 3 -1.133 0.926 16 -1.224 0.7383
## 2 - 4 -0.867 0.926 16 -0.936 0.8788
## 2 - 5 -1.467 0.926 16 -1.584 0.5276
## 3 - 4 0.267 0.926 16 0.288 0.9983
## 3 - 5 -0.333 0.926 16 -0.360 0.9960
## 4 - 5 -0.600 0.926 16 -0.648 0.9646
##
## Results are averaged over the levels of: bloc
## P value adjustment: tukey method for comparing a family of 5 estimates# Contrastes par a par usando a `mcp(. = "Tukey")`. O ajuste para a
# multiplicidade é feito com pelo método FDR (false discovery rate).
summary(glht(m0, linfct = mcp(trat = "Tukey")),
test = adjusted(type = "fdr"))##
## Simultaneous Tests for General Linear Hypotheses
##
## Multiple Comparisons of Means: Tukey Contrasts
##
##
## Fit: lm(formula = y ~ bloc + trat, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 2 - 1 == 0 -9.2000 0.9262 -9.933 3.01e-07 ***
## 3 - 1 == 0 -8.0667 0.9262 -8.710 6.03e-07 ***
## 4 - 1 == 0 -8.3333 0.9262 -8.998 5.85e-07 ***
## 5 - 1 == 0 -7.7333 0.9262 -8.350 7.92e-07 ***
## 3 - 2 == 0 1.1333 0.9262 1.224 0.398
## 4 - 2 == 0 0.8667 0.9262 0.936 0.519
## 5 - 2 == 0 1.4667 0.9262 1.584 0.266
## 4 - 3 == 0 -0.2667 0.9262 -0.288 0.777
## 5 - 3 == 0 0.3333 0.9262 0.360 0.777
## 5 - 4 == 0 0.6000 0.9262 0.648 0.658
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- fdr method)Com contrast(., method = "tukey") são obtidos os contrastes par a par, ou seja, contrastes de Tukey. A documentação do pacote emmeans recomenda usar a multcomp::glht() e seus métodos associados para resultados mais confiáveis já que na multcomp dispõe de maior controle de opções e flexibilidade para uso em diferentes classes de modelos.
O método de Tukey usando pela contrast(., method = "tukey") é aproximado quando os erros padrões diferem. Isso é mencionado na seção Details documentação ?tukey.emmc: The default multiplicity adjustment method is “tukey”, which is only approximate when the standard errors differ. Por essa razão, recomenda-se priorizar o uso da multcomp::glht().
# As mesmas comparações múltiplas de antes.
results_m0 <- wzRfun::apmc(X = L,
model = m0,
focus = "trat",
test = "fdr")
results_m0## trat fit lwr upr cld
## 1 1 32.76667 31.40168 34.13165 a
## 2 2 23.56667 22.20168 24.93165 b
## 3 3 24.70000 23.33502 26.06498 b
## 4 4 24.43333 23.06835 25.79832 b
## 5 5 25.03333 23.66835 26.39832 b# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results_m0,
mapping = aes(x = fit, y = reorder(trat, fit))) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lwr, xmax = upr),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
vjust = 0,
nudge_y = 0.07) +
labs(x = "Produção",
y = "Tratamentos")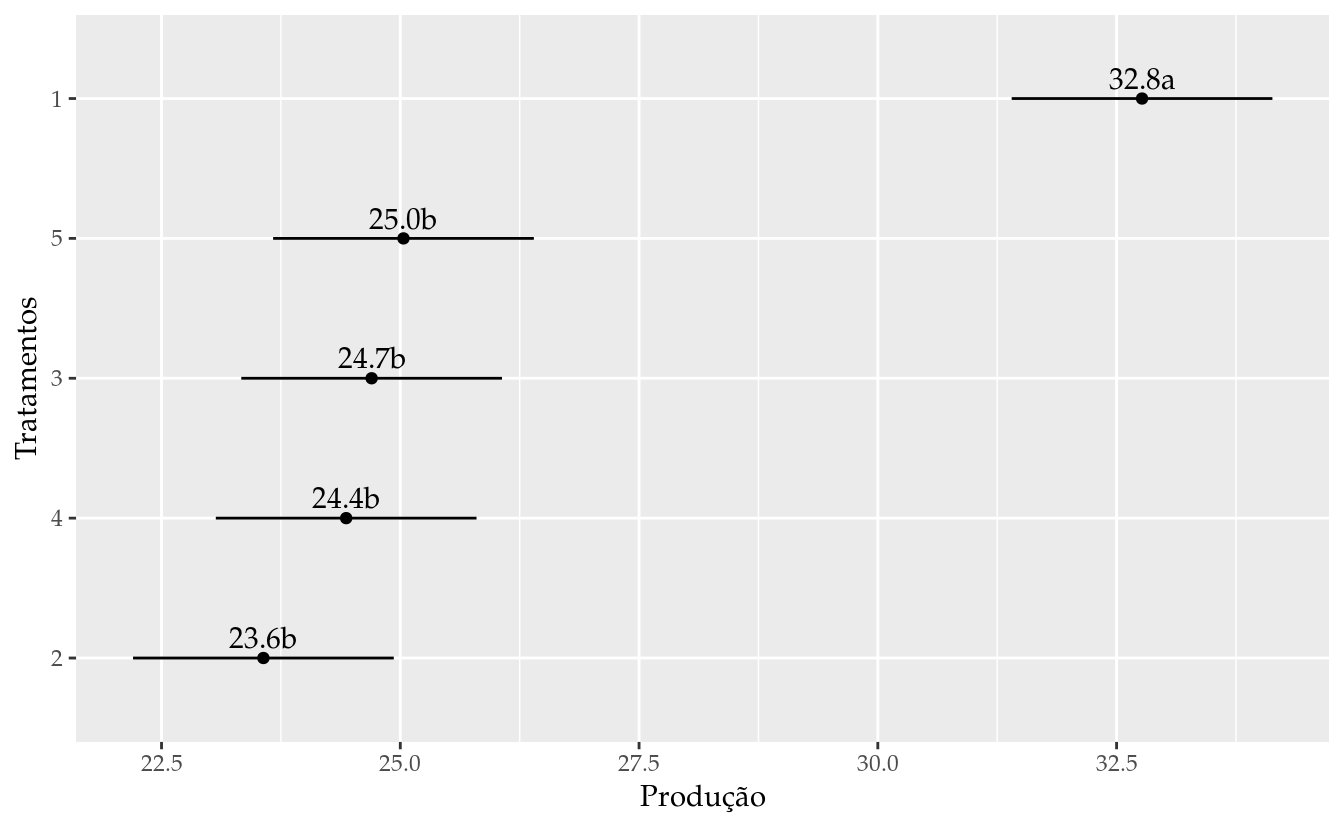
A função agricolae::BIB.test() faz a análise considerando o modelo de efeitos fixos utilizado até aqui. A função aplica o teste de Tukey HSD.
# Usando função do `agricolae`.
with(da,
BIB.test(block = bloc,
trt = trat,
y = y,
test = "tukey",
console = TRUE))##
## ANALYSIS BIB: y
## Class level information
##
## Block: 1 2 3 4 5 6 7 8 9 10
## Trt : 1 2 3 4 5
##
## Number of observations: 30
##
## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## block.unadj 9 412.70 45.856 21.383 2.746e-07 ***
## trt.adj 4 283.69 70.922 33.072 1.495e-07 ***
## Residuals 16 34.31 2.144
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## coefficient of variation: 5.6 %
## y Means: 26.1
##
## trat, statistics
##
## y mean.adj SE r std Min Max
## 1 30.50000 32.76667 0.6438886 6 4.230839 25 36
## 2 24.33333 23.56667 0.6438886 6 5.163978 16 29
## 3 26.83333 24.70000 0.6438886 6 4.020779 19 30
## 4 25.16667 24.43333 0.6438886 6 5.307228 16 30
## 5 23.66667 25.03333 0.6438886 6 4.633213 18 29
##
## Tukey
## Alpha : 0.05
## Std.err : 0.6548961
## HSD : 2.83746
## Parameters BIB
## Lambda : 3
## treatmeans : 5
## Block size : 3
## Blocks : 10
## Replication: 6
##
## Efficiency factor 0.8333333
##
## <<< Book >>>
##
## Comparison between treatments means
## Difference pvalue sig.
## 1 - 2 9.2000000 0.0000 ***
## 1 - 3 8.0666667 0.0000 ***
## 1 - 4 8.3333333 0.0000 ***
## 1 - 5 7.7333333 0.0000 ***
## 2 - 3 -1.1333333 0.7383
## 2 - 4 -0.8666667 0.8788
## 2 - 5 -1.4666667 0.5276
## 3 - 4 0.2666667 0.9983
## 3 - 5 -0.3333333 0.9960
## 4 - 5 -0.6000000 0.9646
##
## Treatments with the same letter are not significantly different.
##
## y groups
## 1 32.76667 a
## 5 25.03333 b
## 3 24.70000 b
## 4 24.43333 b
## 2 23.56667 bOs testes de hipóteses para os contrastes de Tukey são iguais entre a contrast(., method = "tukey") e a BIB.test(). O recomendável é usar tais funções apenas em situação de balanceamento. Ou seja, caso ocorra perda de unidade experimental, tenha covariável, etc, o uso não é mais recomendado.
13.3 Análise com recupação de informação interblocos
A análise com recuperação da informação interblocos nada mais é que o procedimento para obtenção das médias ajustadas marginais dos tratamentos conforme um modelo de efeito aleatório de bloco mas a partir do ajuste do modelo de efeito fixo de bloco. Simplificando, é obter o resultado de um modedelo de efeito aleatório a partir de um modelo e efeito fixo de blocos.
Na análise interbloco assumimos que os efeitos de bloco são aleatórios e usamos a informação a respeito das diferenças entre blocos para fazer a estimação dos efeitos dos tratamentos. Para a estimação das médias marginais ajustadas dos tratamentos precisamos da estimativa de componente de variância de blocos ajustados para tratamentos (\(\hat{\sigma}^2_b\)) e da estimativa da variância residual (\(\hat{\sigma}^2\)). Ambas podem ser obtidas do quadro de análise de variância. Com essas estimativas aplicamos a seguinte fórmula \[ \tau_i^{*} = \left\{\begin{matrix} \displaystyle \bar{y}_{..} + \frac{ky_{i.} - \sum_{j = 1}^{b} n_{ij} y_{.j} + h\sum_{j = 1}^{b} n_{ij}y_{.j} - hy_{..}}{ \lambda a + (r - \lambda) h}, & & \text{se } \hat\sigma_b^2 > 0 \\ & & \\ \displaystyle \frac{y_{i.} - (1/a) y_{..}}{r}, & & \text{se } \hat\sigma_b^2 = 0 \end{matrix}\right. \] em que \(i\) é o índice para os níveis de tratamento, \(j\) é o índice para os níveis de bloco, \(h = \hat\sigma^2/(\hat\sigma^2 + k\hat\sigma_{b}^{2})\). Os valores \(n_{ij}\) são ocorrências e \(y_{.j}\), \(y_{i.}\) e \(y_{..}\) são totais marginais de blocos, de tratamentos e global. Interessados nas expressões, recomenda-se consultar o capítulo 6 de ZIMMERMANN (2004).
A função que aplicamos abaixo faz esse computo ao serem fornecidas as variáveis necessárias. A função está definida no arquivo mpaer-functions.R.
# Obtém as médias ajustadas com recuperação da informação interbloco.
# Função está definida em `./mpaer-functions.R`.
res <- emmeans_interbloco(trt = "trat",
blc = "bloc",
resp = "y",
data = da)
res## trat emmeans
## 1 1 32.67313
## 2 2 23.59830
## 3 3 24.78803
## 4 4 24.46359
## 5 5 24.97694# Componentes de variância e mais.
attributes(res)$information## $n_trat
## [1] 5
##
## $n_block
## [1] 10
##
## $block_variance
## [1] 19.92889
##
## $residual_variance
## [1] 2.144444
##
## $df_residual
## [1] 16Na seção a seguir será ajustado o modelo que considera o efeito aleatório de blocos. No final serão apresentadas as médias marginais ajustadas que podem ser comparadas com as previamente obtidas pela análise interbloco. As médias podem ser ligeiramente diferentes por conta da diferença entre o método ANOVA e o método REML na estimativa do componente de variância de efeito de bloco.
TODO e as expressões para os erros padrões? Como que fica?
13.4 Modelo de efeito aleatório de bloco
A análise com modelo de efeitos mistos considera que o termo bloco tem efeito aleatório. Dessa forma, o modelo estatístico é representado por \[ \begin{aligned} Y | i, j &\sim \text{Normal}(\mu_{ij}, \sigma^2)\\ \mu_{ij} &= \mu + \tau_i + b_j\\ b_{j} &\sim \text{Normal}(0, \sigma^2_b) \end{aligned} \] em que \(\sigma^2_b\) é a variância do efeito de bloco.
Esse modelo é ajustado aos dados por máxima verossimilhança restrita (REML) usando a lme4::lmer(). A partir desse modelo serão obtidos os mesmo artefatos, a saber, análise dos pressupostos, estimativas dos parâmetros, médias marginais ajustadas, comparações múltiplas e apresentação gráfica.
# Ajuste do modelo de efeito aleatório de bloc.
mm0 <- lmer(y ~ (1 | bloc) + trat, data = da)
par(mfrow = c(1, 3))
# Resíduos contra os valores preditos.
# plot(mm0)
plot(residuals(mm0) ~ fitted(mm0))
# Normalidade dos resíduos.
qqnorm(residuals(mm0, type = "pearson"))
# Normalidade dos efeitos de bloc.
qqnorm(ranef(mm0)$bloc[, 1])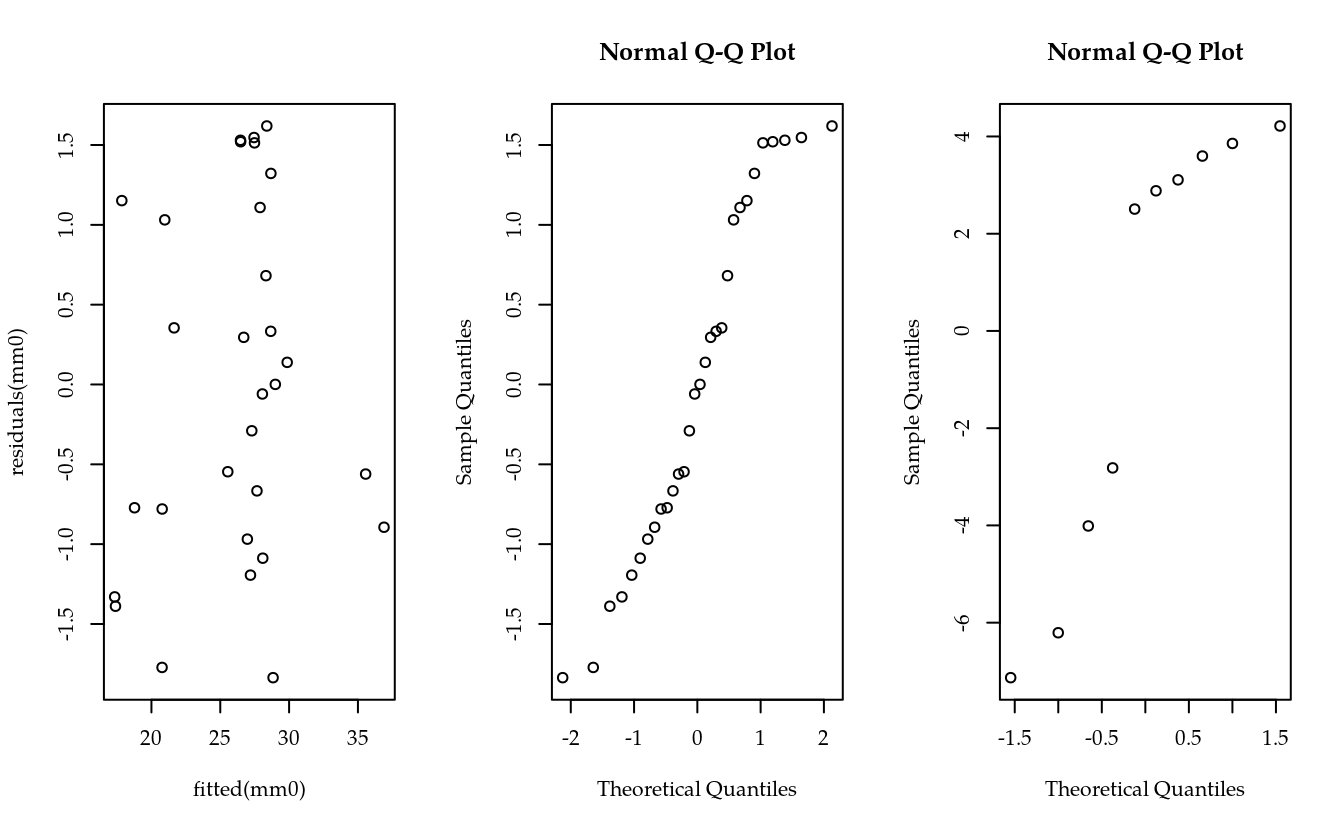
layout(1)
# Quadro de teste de Wald.
anova(mm0)## Type III Analysis of Variance Table with Satterthwaite's method
## Sum Sq Mean Sq NumDF DenDF F value Pr(>F)
## trat 277.86 69.464 4 16.172 32.354 1.571e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros.
summary(mm0, correlation = FALSE)## Linear mixed model fit by REML. t-tests use Satterthwaite's method
## [lmerModLmerTest]
## Formula: y ~ (1 | bloc) + trat
## Data: da
##
## REML criterion at convergence: 129.1
##
## Scaled residuals:
## Min 1Q Median 3Q Max
## -1.25326 -0.59067 -0.02004 0.74339 1.10507
##
## Random effects:
## Groups Name Variance Std.Dev.
## bloc (Intercept) 21.114 4.595
## Residual 2.147 1.465
## Number of obs: 30, groups: bloc, 10
##
## Fixed effects:
## Estimate Std. Error df t value Pr(>|t|)
## (Intercept) 32.6781 1.5888 11.7462 20.568 1.42e-10 ***
## trat2 -9.0814 0.9237 16.1721 -9.832 3.15e-08 ***
## trat3 -7.8947 0.9237 16.1721 -8.547 2.15e-07 ***
## trat4 -8.2161 0.9237 16.1721 -8.895 1.25e-07 ***
## trat5 -7.6982 0.9237 16.1721 -8.334 3.00e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos parâmetros dos termos de efeito aleatório.
# VarCorr(mm0)
# sqrt(attr(res, "information")[["block_variance"]])
# Extração das estimativas dos parâmetros de efeito fixo.
fixef(mm0)## (Intercept) trat2 trat3 trat4 trat5
## 32.678073 -9.081441 -7.894692 -8.216078 -7.698157# Extração das predições dos coeficientes de efeito aleatório.
ranef(mm0)## $bloc
## (Intercept)
## 1 2.882919
## 2 -2.816757
## 3 -6.207789
## 4 4.215946
## 5 -4.011162
## 6 -7.131597
## 7 3.597393
## 8 3.108008
## 9 3.856435
## 10 2.506605
##
## with conditional variances for "bloc"# Médias marginais ajustadas.
emm <- emmeans(mm0, specs = ~trat)
emm## trat emmean SE df lower.CL upper.CL
## 1 32.7 1.59 11.8 29.2 36.1
## 2 23.6 1.59 11.8 20.1 27.1
## 3 24.8 1.59 11.8 21.3 28.3
## 4 24.5 1.59 11.8 21.0 27.9
## 5 25.0 1.59 11.8 21.5 28.4
##
## Degrees-of-freedom method: kenward-roger
## Confidence level used: 0.95# Extração da matriz de funções lineares.
L <- attr(emm, "linfct")
grid <- attr(emm, "grid")
rownames(L) <- grid[[1]]
# Constrastes par a par, ou seja, contrastes de Tukey.
contrast(emm, method = "pairwise")## contrast estimate SE df t.ratio p.value
## 1 - 2 9.081 0.926 16.2 9.810 <.0001
## 1 - 3 7.895 0.926 16.2 8.528 <.0001
## 1 - 4 8.216 0.926 16.2 8.875 <.0001
## 1 - 5 7.698 0.926 16.2 8.315 <.0001
## 2 - 3 -1.187 0.926 16.2 -1.282 0.7054
## 2 - 4 -0.865 0.926 16.2 -0.935 0.8792
## 2 - 5 -1.383 0.926 16.2 -1.494 0.5802
## 3 - 4 0.321 0.926 16.2 0.347 0.9966
## 3 - 5 -0.197 0.926 16.2 -0.212 0.9995
## 4 - 5 -0.518 0.926 16.2 -0.559 0.9791
##
## P value adjustment: tukey method for comparing a family of 5 estimates# Contrastes par a par usando a `mcp(. = "Tukey")`.
summary(glht(mm0, linfct = mcp(trat = "Tukey")))##
## Simultaneous Tests for General Linear Hypotheses
##
## Multiple Comparisons of Means: Tukey Contrasts
##
##
## Fit: lmer(formula = y ~ (1 | bloc) + trat, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error z value Pr(>|z|)
## 2 - 1 == 0 -9.0814 0.9237 -9.832 <1e-04 ***
## 3 - 1 == 0 -7.8947 0.9237 -8.547 <1e-04 ***
## 4 - 1 == 0 -8.2161 0.9237 -8.895 <1e-04 ***
## 5 - 1 == 0 -7.6982 0.9237 -8.334 <1e-04 ***
## 3 - 2 == 0 1.1867 0.9237 1.285 0.701
## 4 - 2 == 0 0.8654 0.9237 0.937 0.883
## 5 - 2 == 0 1.3833 0.9237 1.498 0.564
## 4 - 3 == 0 -0.3214 0.9237 -0.348 0.997
## 5 - 3 == 0 0.1965 0.9237 0.213 1.000
## 5 - 4 == 0 0.5179 0.9237 0.561 0.981
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)# As mesmas comparações múltiplas.
results_mm0 <- wzRfun::apmc(L, model = mm0, focus = "trat")
results_mm0## trat fit lwr upr cld
## 1 1 32.67807 29.56412 35.79202 a
## 2 2 23.59663 20.48268 26.71058 b
## 3 3 24.78338 21.66943 27.89733 b
## 4 4 24.46200 21.34805 27.57595 b
## 5 5 24.97992 21.86597 28.09387 b# Matriz de covariância para as médias marginais.
vcov_mm0 <- L %*% vcov(mm0) %*% t(L)Comparações entre os modelos.
# Gráfico de segmentos para as estimativas intervalares.
results <- bind_rows(list(fixo = results_m0,
aleatorio = results_mm0),
.id = "modelo")
s <- 0.35
pd <- position_dodge(0.25)
ggplot(data = results,
mapping = aes(x = trat,
y = fit,
group = modelo,
color = modelo)) +
geom_point(position = pd) +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0,
position = pd) +
geom_text(mapping = aes(x = as.integer(trat) +
ifelse(modelo == "fixo", s, -s),
label = sprintf("%0.2f%s", fit, cld)),
position = pd,
hjust = 0.5,
show.legend = FALSE) +
labs(y = "Produção",
x = "Tratamentos")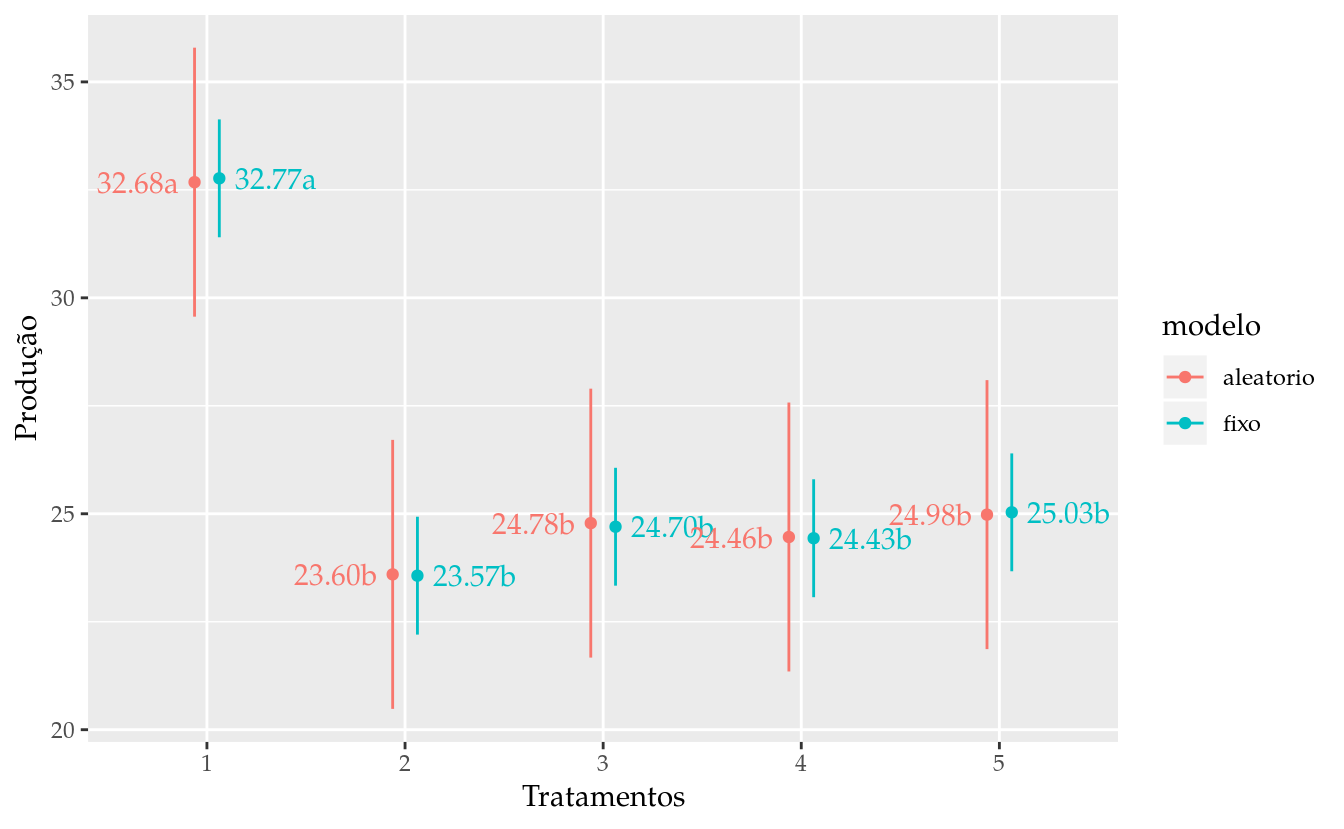
# Matriz de correlação da matriz de covariância das médias marginais.
v <- 1:3
cov2cor(vcov_m0)[v, v]## 1 2 3
## 1 1.00000000 -0.03448276 -0.03448276
## 2 -0.03448276 1.00000000 -0.03448276
## 3 -0.03448276 -0.03448276 1.00000000cov2cor(vcov_mm0)[v, v]## 3 x 3 Matrix of class "dsyMatrix"
## 1 2 3
## 1 1.000000 0.830996 0.830996
## 2 0.830996 1.000000 0.830996
## 3 0.830996 0.830996 1.000000# Erro padrão do contraste.
sqrt(vcov_m0[1, 1] + vcov_m0[2, 2] - 2 * vcov_m0[1, 2])## [1] 0.9261629sqrt(vcov_mm0[1, 1] + vcov_mm0[2, 2] - 2 * vcov_mm0[1, 2])## [1] 0.9236918# Erro padrão do contraste.
v <- c("coefficients", "sigma")
summary(glht(m0, linfct = mcp(trat = "Tukey")))$test[v] %>%
as.data.frame()## coefficients sigma
## 2 - 1 -9.2000000 0.9261629
## 3 - 1 -8.0666667 0.9261629
## 4 - 1 -8.3333333 0.9261629
## 5 - 1 -7.7333333 0.9261629
## 3 - 2 1.1333333 0.9261629
## 4 - 2 0.8666667 0.9261629
## 5 - 2 1.4666667 0.9261629
## 4 - 3 -0.2666667 0.9261629
## 5 - 3 0.3333333 0.9261629
## 5 - 4 0.6000000 0.9261629summary(glht(mm0, linfct = mcp(trat = "Tukey")))$test[v] %>%
as.data.frame()## coefficients sigma
## 2 - 1 -9.0814414 0.9236918
## 3 - 1 -7.8946916 0.9236918
## 4 - 1 -8.2160776 0.9236918
## 5 - 1 -7.6981566 0.9236918
## 3 - 2 1.1867498 0.9236918
## 4 - 2 0.8653638 0.9236918
## 5 - 2 1.3832848 0.9236918
## 4 - 3 -0.3213860 0.9236918
## 5 - 3 0.1965350 0.9236918
## 5 - 4 0.5179210 0.923691813.5 Mais exemplos
13.5.1 Exemplo 1
Dados para o exemplo estão em PimentelTb10.6.1. Número de tratamentos TODO, tamanho do bloco TODO, repetições TODO, lambda TODO.
da <- labestData::PimentelTb10.6.1
str(da)
# São 4 repetições de cada tratamento no experimento e blocos de tamanho
# 4 unidades experimentais.
addmargins(xtabs(~trat + bloc, data = da))
# Número de ocorrência dos pares de tratamentos.
r <- 4; k <- 4; a <- 13
r * (k - 1)/(a - 1)
# Quantas vezes cada par ocorre junto.
# by(data = da$trat,
# INDICES = da$bloc,
# FUN = function(x) {
# apply(combn(sort(x), m = 2),
# MARGIN = 2,
# FUN = paste0,
# collapse = "_")
# }) %>%
# flatten_chr() %>%
# table()
# Obtém as coordenadas radiais para gráfico de conexão.
da <- da %>%
do({
k <- nlevels(.$trat)
a <- seq(0, 2 * pi, length.out = k + 1)[-(k + 1)]
cbind(.,
data.frame(coord_x = sin(a)[as.integer(.$trat)],
coord_y = cos(a)[as.integer(.$trat)]))
})
# Gráfico de conexão.
ext <- c(-1.2, 1.2)
ggplot(data = da,
mapping = aes(coord_x, coord_y)) +
facet_wrap(facets = ~bloc) +
geom_point() +
geom_polygon(fill = NA, color = "orange") +
geom_label(mapping = aes(label = trat)) +
expand_limits(x = ext, y = ext) +
coord_equal()
# Ajuste do modelo de efeitos fixos.
m0 <- lm(z ~ bloc + trat, data = da)
# Análise dos pressupostos.
par(mfrow = c(2, 2)); plot(m0); layout(1)
# Quadro de análise de variância com hipóteses marginais.
# drop1(m0, test = "F")
car::Anova(m0)
# Médias marginais.
emm <- emmeans(m0, specs = ~trat)
emm
# Extração da matriz de funções lineares.
grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid[[1]]
MASS::fractions(t(L))
# As mesmas comparações múltiplas.
results_m0 <- wzRfun::apmc(L,
model = m0,
focus = "trat",
test = "fdr")
results_m0
# Ajuste do modelo de efeito aleatório de bloc.
mm0 <- lmer(z ~ (1 | bloc) + trat, data = da)
par(mfrow = c(1, 3))
# Resíduos contra os valores preditos.
# plot(mm0)
plot(residuals(mm0) ~ fitted(mm0))
# Normalidade dos resíduos.
qqnorm(residuals(mm0, type = "pearson"))
# Normalidade dos efeitos de bloc.
qqnorm(ranef(mm0)$bloc[, 1])
layout(1)
# Quadro de teste de Wald.
anova(mm0)
# Estimativas dos parâmetros dos termos de efeito.
summary(mm0)
# Médias marginais.
emm <- emmeans(mm0, specs = ~trat)
emm
# Extração da matriz de funções lineares.
grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid[[1]]
# As mesmas comparações múltiplas.
results_mm0 <- wzRfun::apmc(L,
model = mm0,
focus = "trat",
test = "fdr")
results_mm0
# Gráfico de segmentos para as estimativas intervalares.
results <- bind_rows(list(fixo = results_m0,
aleatorio = results_mm0),
.id = "modelo")
s <- 0.35
pd <- position_dodge(0.25)
ggplot(data = results,
mapping = aes(x = trat,
y = fit,
group = modelo,
color = modelo)) +
geom_point(position = pd) +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0,
position = pd) +
geom_text(mapping = aes(x = as.integer(trat) +
ifelse(modelo == "fixo", s, -s),
label = sprintf("%0.2f%s", fit, cld)),
position = pd,
hjust = 0.5,
show.legend = FALSE) +
labs(y = "Produção",
x = "Tratamentos")
# Erro padrão do contraste.
vcov_m0[1, 1] + vcov_m0[2, 2] - 2 * vcov_m0[1, 2]
vcov_mm0[1, 1] + vcov_mm0[2, 2] - 2 * vcov_mm0[1, 2]
# Matriz de correlação da matriz de covariância das médias marginais.
cov2cor(vcov_m0)[1:5, 1:5]
cov2cor(vcov_mm0)[1:5, 1:5]13.6 Obtenção de delineamentos em BIB
Diferente dos delineamentos em blocos completos, os blocos incompletos balanceados não são determináveis para qualquer escolha de número de tratamentos e tamanho de bloco. Esses delineamentos são obtidos para certas combinações dessas quantidades. Existe uma relação com números primos e potências de números bem curiosa.
Os livros de estatística experimental, como ZIMMERMANN (2004) e TODO, trazem tabelas com esses delineamentos já determinados. O pacote agricolae possui a função design.bib() que constrói delineamentos desse tipo.
TODO: como é calculada a eficiência.
# 5 tratamentos e bloco de tamanho 3.
des <- design.bib(trt = gl(3, 1),
k = 2,
randomization = FALSE)## [1] "No improvement over initial random design."
##
## Parameters BIB
## ==============
## Lambda : 1
## treatmeans : 3
## Block size : 2
## Blocks : 3
## Replication: 2
##
## Efficiency factor 0.75
##
## <<< Book >>># Alocação de 3 tratamentos em blocos de tamanho 2.
des$sketch## [,1] [,2]
## [1,] "1" "2"
## [2,] "1" "3"
## [3,] "2" "3"# Tabela (não randomizada).
des$book## plots block gl(3, 1)
## 1 101 1 1
## 2 102 1 2
## 3 201 2 1
## 4 202 2 3
## 5 301 3 2
## 6 302 3 3# 5 tratamentos e bloco de tamanho 3.
des <- design.bib(trt = gl(5, 1), k = 3, randomization = FALSE)
# 6 tratamentos e bloco de tamanho 3.
des <- design.bib(trt = gl(6, 1), k = 3, randomization = FALSE)
# 9 tratamentos e bloco de tamanho 4.
des <- design.bib(trt = gl(9, 1), k = 4, randomization = FALSE)
# 10 tratamentos e bloco de tamanho 3.
des <- design.bib(trt = gl(10, 1), k = 3, randomization = FALSE)O pacote AlgDesign possui a função optBlock() para a construção de delineamentos com blocagem. Essa função permite controle de diversos aspectos da construção do delineamento e não limita-se a construção de delineamentos incompletos. Um detalhe é que, por esta ser uma função genérica, o delineamento retornado pode não ser exatamente um delineamento de blocos incompletos balanceados.
Para que o delineamento seja de blocos incompletos balanceados, as duas quantidades a seguir devem ser números naturais positivos \[ \frac{bk}{t} \in \mathbb{N}^{+} \text{ e } \frac{bk(k - 1)}{t (t - 1)} \in \mathbb{N}^{+}, \] em que \(b\) é o número de blocos, \(k\) o tamanho do bloco e \(t\) o número de tratamentos.
O pacote crossdes, apesar de especializado na construção de delineamentos crossover, possui a função find.BIB() para a construção do delineamento de blocos incompletos balanceados. Essa função faz chamada a optBlock() mas com a verificação de condições mencionadas para que o delineamento retornado seja mesmo de blocos incompletos balanceados.
t <- 5 # Número de tratamentos.
b <- 10 # Número de blocos.
k <- 3 # Tamanho dos blocos.
# Condições de existência.
(b * k)/t # Número de repetições.
(b * k * (k - 1))/(t * (t - 1)) # Número de pares.
# 5 tratamentos em 10 blocos de tamanho 3.
des <- AlgDesign::optBlock(~.,
withinData = gl(t, 1),
blocksizes = rep(k, b))
do.call(cbind, des$Blocks) %>%
t() %>%
as.numeric() %>%
`dim<-`(c(10, 3)) %>%
as.data.frame() %>%
arrange(V1, V2, V3)
# 5 tratamentos em 10 blocos de tamanho 3.
crossdes::find.BIB(trt = t, b = b, k = k) %>%
as.data.frame() %>%
arrange(V1, V2, V3)13.7 Considerações finais
TODO
Referências Bibliográficas
ZIMMERMANN, F. J. Estatística aplicada à pesquisa agrícola. 1st ed. Santo Antônio de Goiás, GO: Embrapa Arroz e Feijão, 2004.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani