Capítulo 3 Delineamento de Blocos Casualizados
3.1 Motivação

Figura 3.1: Ilustração das etapas para construção de um delineamento em blocos completos casualizados em experimento com galinhas.
3.2 Ensaio de batatinha em Pimentel
# ATTENTION: Limpa espaço de trabalho.
rm(list = objects())
# Carrega pacotes.
library(agricolae) # HSD.test(), SNK.test().
library(ScottKnott) # SK().
library(car) # linearHypothesis().
library(multcomp) # glht().
library(emmeans) # emmeans().
library(tidyverse)
# Carrega funções.
source("mpaer_functions.R")# Url de um arquivo com dados.
da <- labestData::PimentelEg5.2
str(da)## 'data.frame': 32 obs. of 3 variables:
## $ bloco : Factor w/ 4 levels "I","II","III",..: 1 1 1 1 1 1 1 1 2 2 ...
## $ variedade: Factor w/ 8 levels "B 116-51","B 1-52",..: 1 2 3 4 5 6 7 8 1 2 ...
## $ producao : num 23.1 20 12.7 18 15.4 21.1 9.2 22.6 24.2 21.1 ...# Tabela de frequência.
xtabs(~variedade + bloco, data = da)## bloco
## variedade I II III IV
## B 116-51 1 1 1 1
## B 1-52 1 1 1 1
## B 25-50 E 1 1 1 1
## B 72-53 A 1 1 1 1
## Buena Vista 1 1 1 1
## Huinkul 1 1 1 1
## Kennebec 1 1 1 1
## S. Rafalela 1 1 1 1ggplot(data = da,
mapping = aes(x = reorder(variedade, producao),
y = producao,
color = bloco,
group = bloco)) +
geom_point() +
geom_line() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")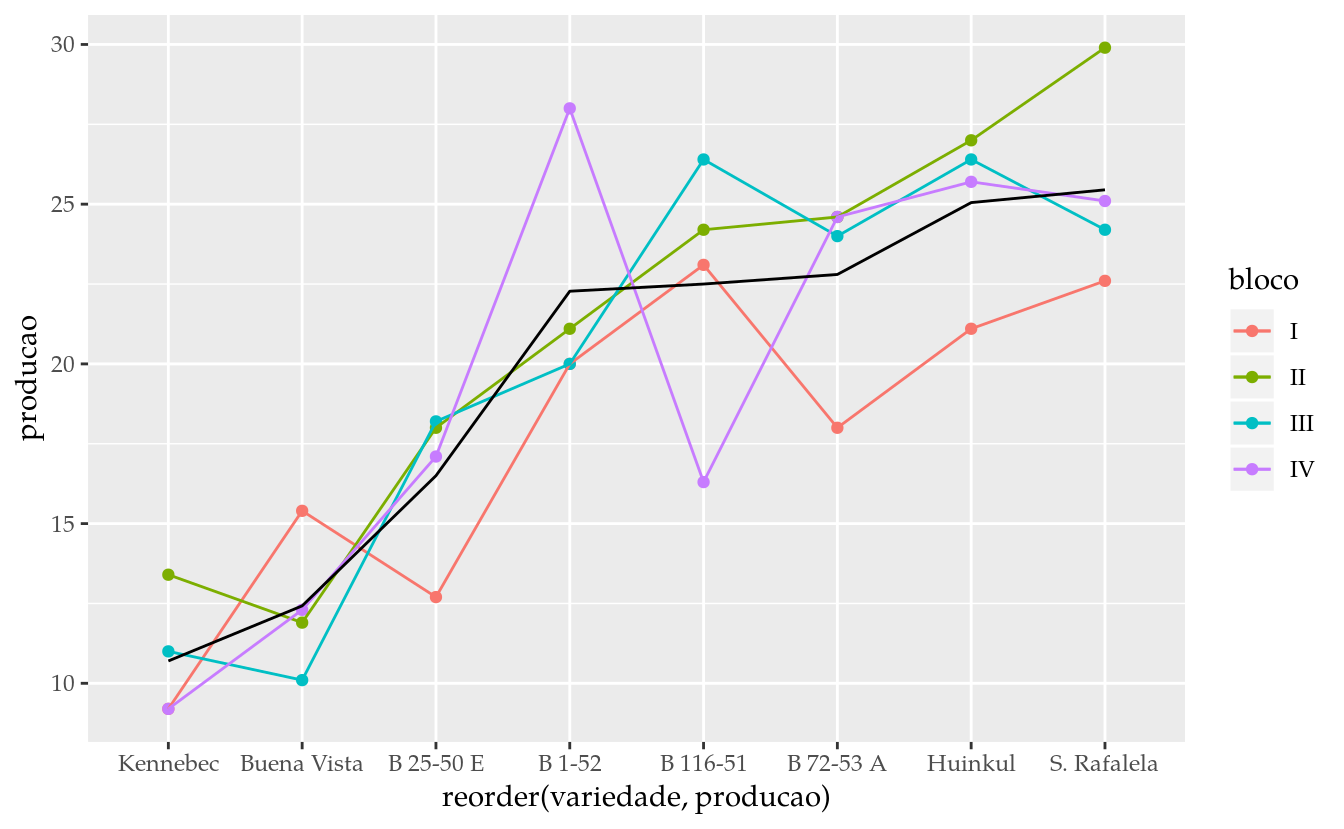
O modelo estatístico correspondente ao experimento é \[ \begin{aligned} Y | i, j &\sim \text{Normal}(\mu_{ij}, \sigma^2) \\ \mu_{ij} &= \mu + \alpha_i + \tau_j \end{aligned} \] em que \(\alpha_i\) é o efeito do bloco \(i\) e \(\tau_j\) o efeito da variedade \(j\) sobre a variável resposta \(Y\), \(\mu\) é a média de \(Y\) na ausência do efeito de bloco e variedade, \(\mu_{ij}\) é a média para as observações em cada combinação de níveis de bloco e variedade e \(\sigma^2\) é a variância das observações ao redor desse valor médio.
m0 <- lm(producao ~ bloco + variedade, data = da)
# As inferências acima só passam ser valiadas após confirmação de não
# haver afastamento dos pressupostos.
par(mfrow = c(2, 2))
plot(m0)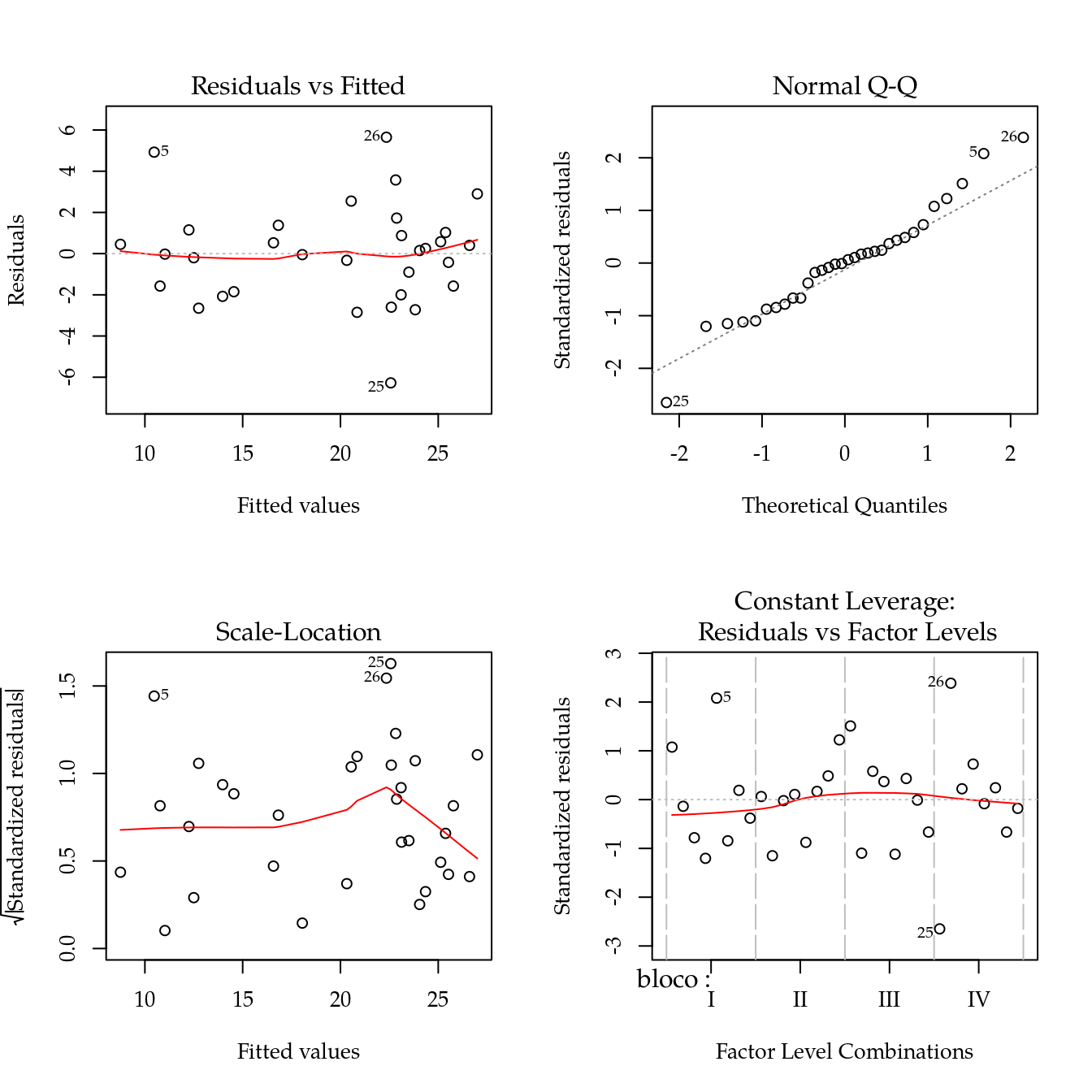
layout(1)
MASS::boxcox(m0)
# Existe efeito das fontes de variação? Ou seja, todos os \alpha_i podem
# ser considerados iguais à zero? Todos os \tau_j podem ser considerados
# à zero?
# anova(m0)
Anova(m0)## Anova Table (Type II tests)
##
## Response: producao
## Sum Sq Df F value Pr(>F)
## bloco 50.53 3 1.9709 0.1493
## variedade 919.72 7 15.3744 5.723e-07 ***
## Residuals 179.47 21
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1summary(m0)##
## Call:
## lm(formula = producao ~ bloco + variedade, data = da)
##
## Residuals:
## Min 1Q Median 3Q Max
## -6.2750 -1.6437 0.0625 1.0563 5.6500
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 20.550 1.714 11.990 7.40e-11 ***
## blocoII 3.500 1.462 2.395 0.02605 *
## blocoIII 2.275 1.462 1.556 0.13455
## blocoIV 2.025 1.462 1.385 0.18047
## variedadeB 1-52 -0.225 2.067 -0.109 0.91436
## variedadeB 25-50 E -6.000 2.067 -2.903 0.00851 **
## variedadeB 72-53 A 0.300 2.067 0.145 0.88599
## variedadeBuena Vista -10.075 2.067 -4.874 8.08e-05 ***
## variedadeHuinkul 2.550 2.067 1.234 0.23098
## variedadeKennebec -11.800 2.067 -5.708 1.15e-05 ***
## variedadeS. Rafalela 2.950 2.067 1.427 0.16825
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.923 on 21 degrees of freedom
## Multiple R-squared: 0.8439, Adjusted R-squared: 0.7696
## F-statistic: 11.35 on 10 and 21 DF, p-value: 2.153e-06# Interpretações sob a restrição paramétrica de zerar o primeiro nível.
contrasts(da$bloc)## II III IV
## I 0 0 0
## II 1 0 0
## III 0 1 0
## IV 0 0 1contrasts(da$vari)## B 1-52 B 25-50 E B 72-53 A Buena Vista Huinkul Kennebec
## B 116-51 0 0 0 0 0 0
## B 1-52 1 0 0 0 0 0
## B 25-50 E 0 1 0 0 0 0
## B 72-53 A 0 0 1 0 0 0
## Buena Vista 0 0 0 1 0 0
## Huinkul 0 0 0 0 1 0
## Kennebec 0 0 0 0 0 1
## S. Rafalela 0 0 0 0 0 0
## S. Rafalela
## B 116-51 0
## B 1-52 0
## B 25-50 E 0
## B 72-53 A 0
## Buena Vista 0
## Huinkul 0
## Kennebec 0
## S. Rafalela 1# Comparações múltiplas.
# Médias marginais ajustdas.
emm <- emmeans(m0, specs = ~variedade)
emm## variedade emmean SE df lower.CL upper.CL
## B 116-51 22.5 1.46 21 19.46 25.5
## B 1-52 22.3 1.46 21 19.24 25.3
## B 25-50 E 16.5 1.46 21 13.46 19.5
## B 72-53 A 22.8 1.46 21 19.76 25.8
## Buena Vista 12.4 1.46 21 9.39 15.5
## Huinkul 25.1 1.46 21 22.01 28.1
## Kennebec 10.7 1.46 21 7.66 13.7
## S. Rafalela 25.4 1.46 21 22.41 28.5
##
## Results are averaged over the levels of: bloco
## Confidence level used: 0.95# Comparações múltiplas, contrastes de Tukey.
# Método de correção de p-valor: false discovey rate.
tk_fdr <- summary(glht(m0, linfct = mcp(variedade = "Tukey")),
test = adjusted(type = "fdr"))
# Resumo compacto com letras.
cld(tk_fdr, decreasing = TRUE)## B 116-51 B 1-52 B 25-50 E B 72-53 A Buena Vista
## "a" "a" "b" "a" "bc"
## Huinkul Kennebec S. Rafalela
## "a" "c" "a"# Teste HSD de Tukey.
tk_hsd <- HSD.test(m0, trt = "variedade")
tk_hsd$statistics## MSerror Df Mean CV MSD
## 8.545952 21 19.7125 14.82991 6.933413tk_hsd$parameters## test name.t ntr StudentizedRange alpha
## Tukey variedade 8 4.743477 0.05results <- tk_hsd$groups %>%
rownames_to_column(var = "variedade") %>%
mutate(groups = str_trim(as.character(groups)))
results <- inner_join(results, as.data.frame(emm))## Joining, by = "variedade"results## variedade producao groups emmean SE df lower.CL upper.CL
## 1 S. Rafalela 25.450 a 25.450 1.461673 21 22.410284 28.48972
## 2 Huinkul 25.050 a 25.050 1.461673 21 22.010284 28.08972
## 3 B 72-53 A 22.800 ab 22.800 1.461673 21 19.760284 25.83972
## 4 B 116-51 22.500 ab 22.500 1.461673 21 19.460284 25.53972
## 5 B 1-52 22.275 ab 22.275 1.461673 21 19.235284 25.31472
## 6 B 25-50 E 16.500 bc 16.500 1.461673 21 13.460284 19.53972
## 7 Buena Vista 12.425 c 12.425 1.461673 21 9.385284 15.46472
## 8 Kennebec 10.700 c 10.700 1.461673 21 7.660284 13.73972# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results,
mapping = aes(x = producao, y = reorder(variedade, producao))) +
geom_point() +
geom_errorbarh(mapping = aes(xmin = lower.CL, xmax = upper.CL),
height = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", producao, groups)),
vjust = 0,
nudge_y = 0.1) +
labs(x = "Produção",
y = "Variedade")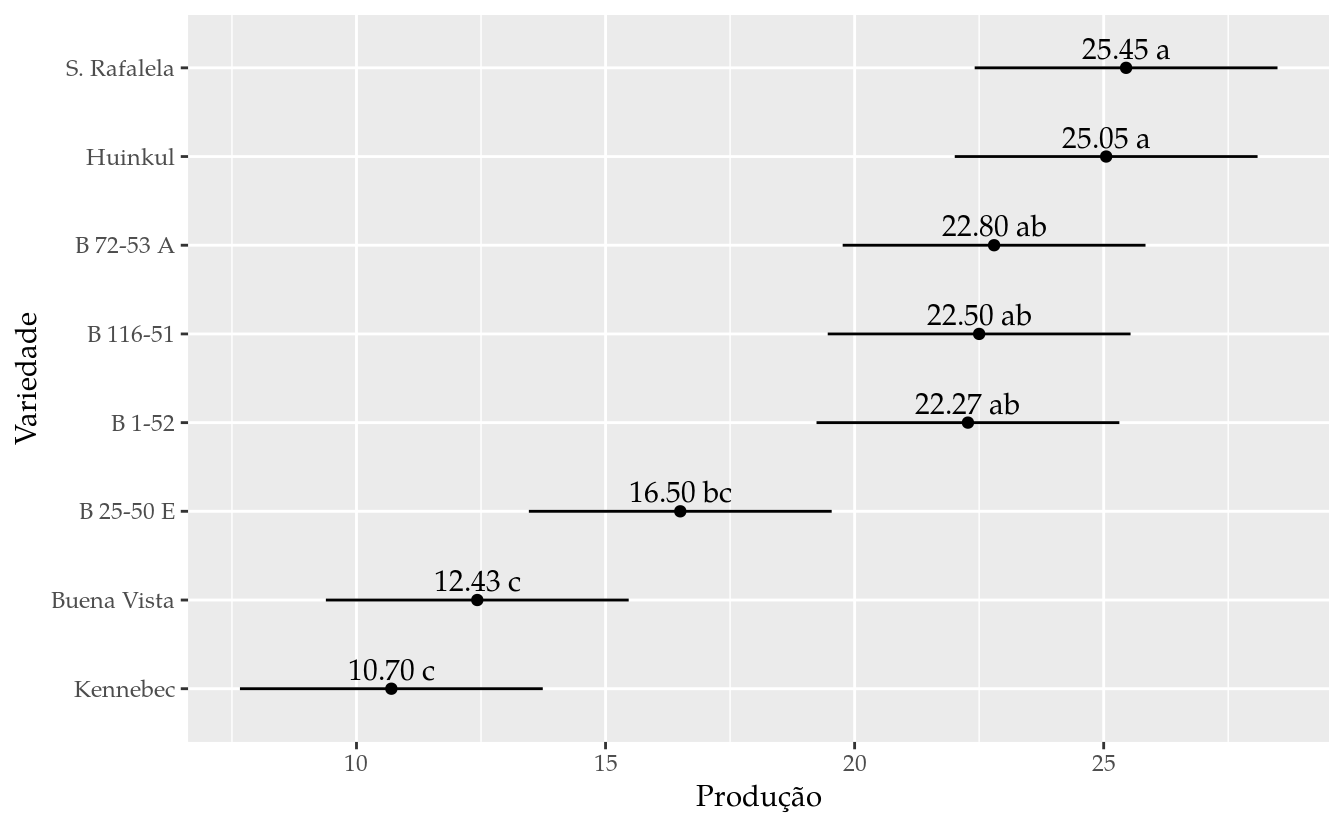
# Teste de ScottKnott.
sk <- SK(x = da,
model = "producao ~ bloco + variedade",
which = "variedade")
# summary(sk)
sk_cls <- data.frame(variedade = rownames(sk$m.inf),
mean = sk$m.inf[, "mean"],
cluster = letters[sk$groups])
inner_join(results, sk_cls) %>%
select(variedade, producao)## Joining, by = "variedade"## variedade producao
## 1 S. Rafalela 25.450
## 2 Huinkul 25.050
## 3 B 72-53 A 22.800
## 4 B 116-51 22.500
## 5 B 1-52 22.275
## 6 B 25-50 E 16.500
## 7 Buena Vista 12.425
## 8 Kennebec 10.7003.3 Peso de frutos
Dados disponíveis em BANZATTO; KRONKA (2013) que são resultados de um experimento instalado na Fazenda Chapadão, no município de Angatuba - SP. O delineamento experimental foi o de blocos casualizados completos, sendo as parcelas constituídas de 4 plantas espaçadas de 6 x 7 metros, com 12 anos de idade na época da instalação do experimento.
O experimento avaliou o efeito do promalim sobre frutos de macieiras. Os tratamentos foram: {0, 12,5; 25,0; 50,0, 12,5 + 12,5} ppm de promalim em plena floração. O último nível teve o complemento de 12,5 no início da frutificação e o primeiro é a testemunha.
# https://github.com/pet-estatistica/labestData/blob/devel/data-raw/BanzattoQd4.5.2.txt
da <- labestData::BanzattoQd4.5.2
summary(da)## promalin bloco peso
## 12.5 :4 I :5 Min. :130.6
## 25.0 :4 II :5 1st Qu.:136.8
## 50.0 :4 III:5 Median :141.6
## 12.5+12.5 :4 IV :5 Mean :143.0
## Testemunha:4 3rd Qu.:146.4
## Max. :165.0# Passa testemunha para primeiro nível para ordem mais lógica.
da <- da %>%
mutate(promalin = fct_relevel(promalin, "Testemunha", after = 0))
# Tabela de frequência.
xtabs(~promalin + bloco, data = da)## bloco
## promalin I II III IV
## Testemunha 1 1 1 1
## 12.5 1 1 1 1
## 25.0 1 1 1 1
## 50.0 1 1 1 1
## 12.5+12.5 1 1 1 1# reshape2::dcast(da, promalin ~ bloco)
da$peso[2] <- 139.3 # Corrige valor registrado errado no pacote.
ggplot(data = da,
mapping = aes(x = promalin,
y = peso,
color = bloco,
group = bloco)) +
geom_point() +
geom_line() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")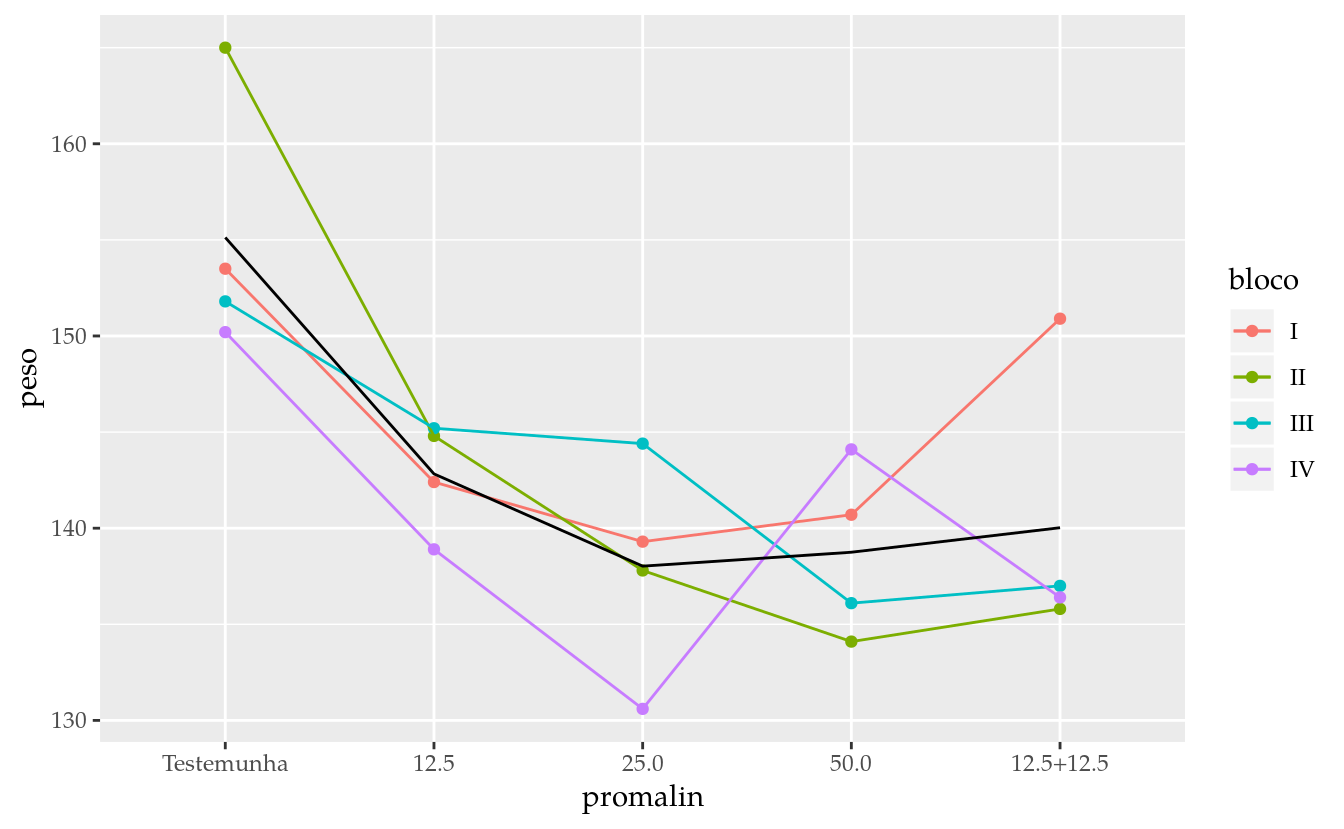
m0 <- lm(peso ~ bloco + promalin, data = da)
# As inferências acima só passam ser valiadas após confirmação de não
# haver afastamento dos pressupostos.
par(mfrow = c(2, 2))
plot(m0)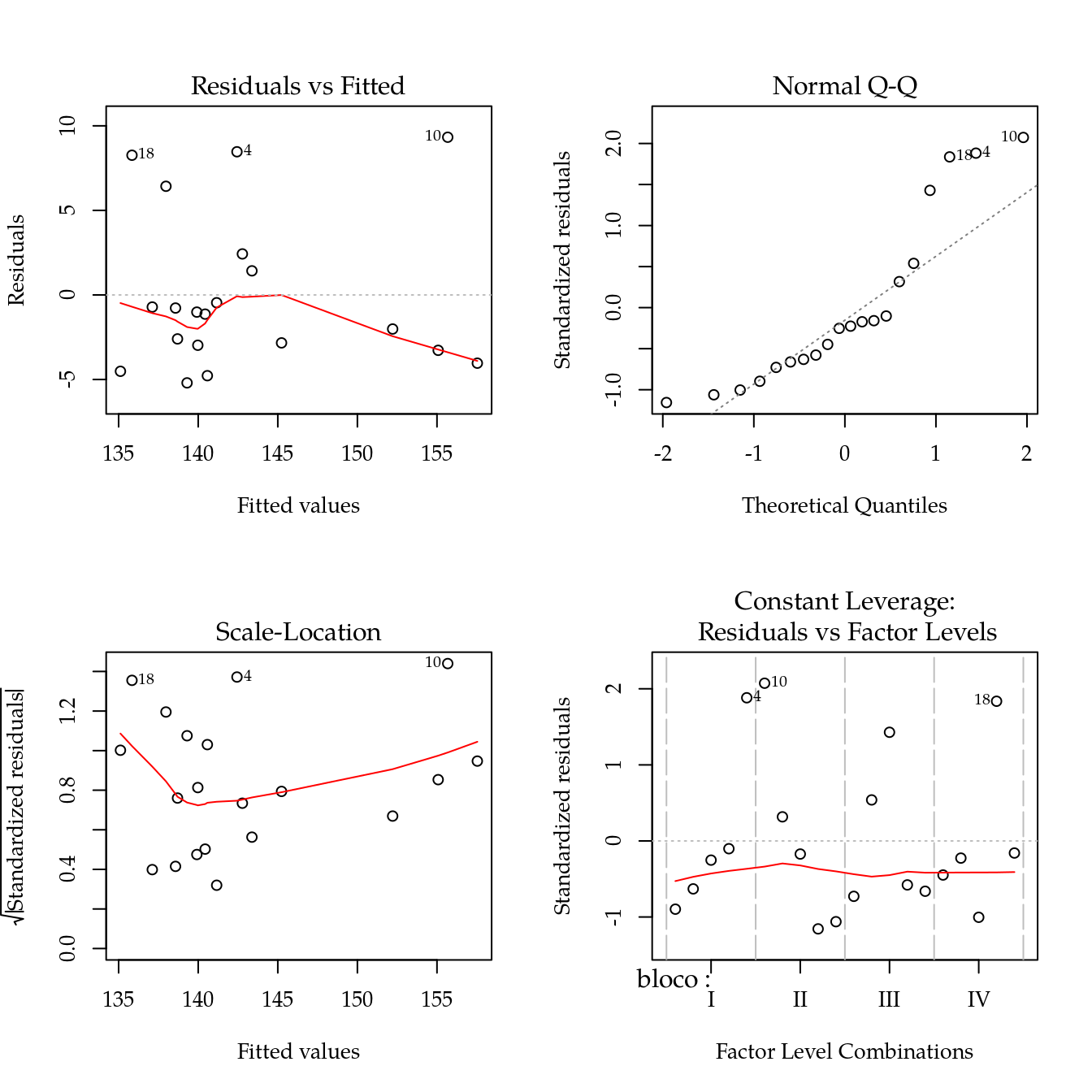
layout(1)
# anova(m0)
Anova(m0)## Anova Table (Type II tests)
##
## Response: peso
## Sum Sq Df F value Pr(>F)
## bloco 72.91 3 0.7207 0.558616
## promalin 794.79 4 5.8929 0.007334 **
## Residuals 404.61 12
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1summary(m0)##
## Call:
## lm(formula = peso ~ bloco + promalin, data = da)
##
## Residuals:
## Min 1Q Median 3Q Max
## -5.200 -3.050 -1.075 1.675 9.325
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 157.535 3.672 42.896 1.67e-14 ***
## blocoII -1.860 3.672 -0.506 0.62170
## blocoIII -2.460 3.672 -0.670 0.51564
## blocoIV -5.320 3.672 -1.449 0.17307
## promalin12.5 -12.300 4.106 -2.996 0.01116 *
## promalin25.0 -17.100 4.106 -4.165 0.00131 **
## promalin50.0 -16.375 4.106 -3.988 0.00180 **
## promalin12.5+12.5 -15.100 4.106 -3.678 0.00316 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.807 on 12 degrees of freedom
## Multiple R-squared: 0.682, Adjusted R-squared: 0.4965
## F-statistic: 3.676 on 7 and 12 DF, p-value: 0.0234# Comparações múltiplas.
# Médias marginais ajustdas.
emm <- emmeans(m0, specs = ~promalin)
emm## promalin emmean SE df lower.CL upper.CL
## Testemunha 155 2.9 12 149 161
## 12.5 143 2.9 12 136 149
## 25.0 138 2.9 12 132 144
## 50.0 139 2.9 12 132 145
## 12.5+12.5 140 2.9 12 134 146
##
## Results are averaged over the levels of: bloco
## Confidence level used: 0.95# Comparações múltiplas, contrastes de Tukey.
# Método de correção de p-valor: false discovey rate.
tk_fdr <- summary(glht(m0, linfct = mcp(promalin = "Tukey")),
test = adjusted(type = "fdr"))
# Resumo compacto com letras.
cld(tk_fdr, decreasing = TRUE)## Testemunha 12.5 25.0 50.0 12.5+12.5
## "a" "b" "b" "b" "b"# Teste HSD de Tukey.
tk_hsd <- HSD.test(m0, trt = "promalin")
tk_hsd$statistics## MSerror Df Mean CV MSD
## 33.71783 12 142.95 4.062054 13.08747tk_hsd$parameters## test name.t ntr StudentizedRange alpha
## Tukey promalin 5 4.50771 0.05results <- tk_hsd$groups %>%
rownames_to_column(var = "promalin") %>%
mutate(groups = str_trim(as.character(groups)))
results <- inner_join(results, as.data.frame(emm)) %>%
wzRfun::equallevels(da)## Joining, by = "promalin"# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results,
mapping = aes(x = promalin, y = peso)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lower.CL, ymax = upper.CL),
width = 0) +
geom_label(mapping = aes(label = sprintf("%0.2f %s",
peso, groups)),
nudge_y = 1.25)+
labs(y = "Peso de frutos de macieira (g)",
x = "Promalin")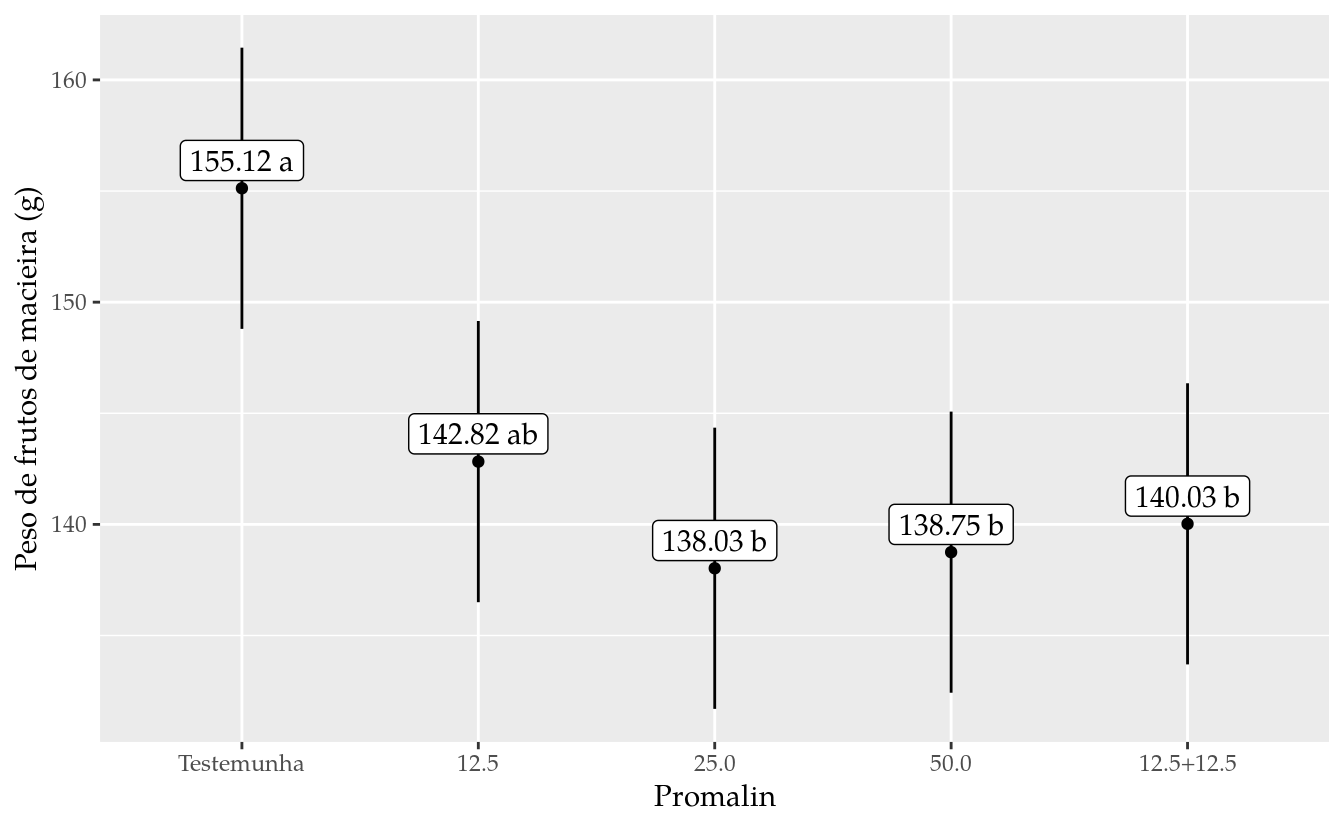
3.4 Repetições dentro da parcela
Essas duas formas não só tem implicações experimentais práticas diferentes como também definem modelos estatísticos próprios.
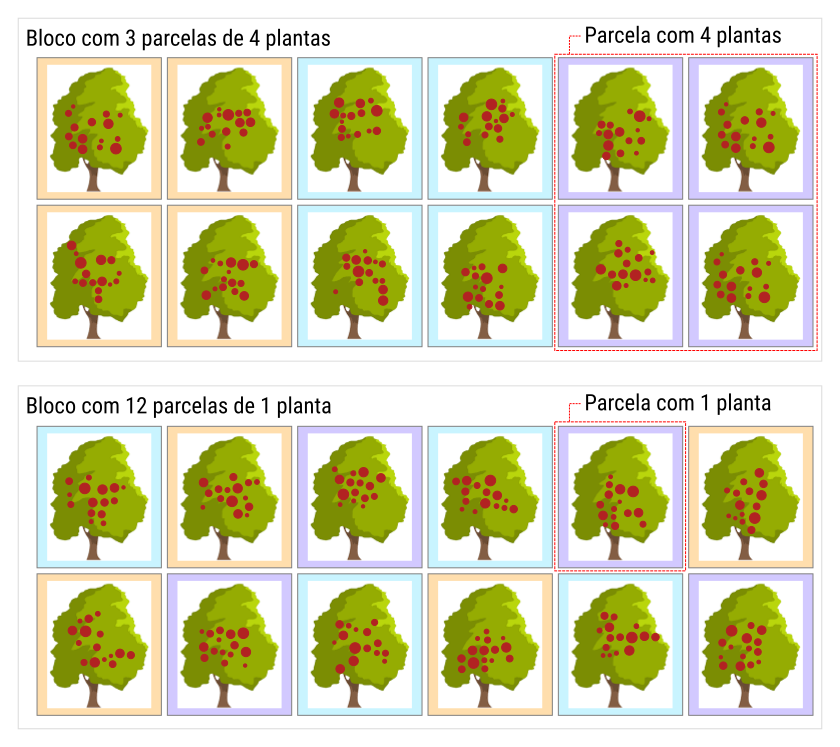
Figura 3.2: Ilustração de formas de fazer repetições dentro do bloco. No esquema de cima, o bloco contém 3 parcelas de 4 plantas cada onde os tratamentos são casualizados a cada parcela. No esquema de baixo o bloco contém 12 parcelas de 1 planta cada onde os tratamentos são casualizados de forma livre.
# https://github.com/pet-estatistica/labestData/blob/devel/data-raw/RamalhoTb7.1.txt
# help(RamalhoTb7.1, help_type = "html")
da <- labestData::RamalhoTb7.1
summary(da)## prog rept plant vol
## 1 :18 Min. :1 Min. :1.0 Min. : 20.0
## 2 :18 1st Qu.:1 1st Qu.:2.0 1st Qu.: 91.0
## 3 :18 Median :2 Median :3.5 Median :133.5
## 4 :18 Mean :2 Mean :3.5 Mean :147.6
## 5 :18 3rd Qu.:3 3rd Qu.:5.0 3rd Qu.:196.5
## 6 :18 Max. :3 Max. :6.0 Max. :388.0
## (Other):72# Renomeia para bloco para caracterizar corretamente o delineamento.
da <- da %>%
rename("bloco" = "rept") %>%
mutate_at(c("bloco", "plant"), "factor") %>%
mutate(parcela = interaction(bloco, prog))
xtabs(~bloco + prog, data = da)## prog
## bloco 1 2 3 4 5 6 7 8 9 10
## 1 6 6 6 6 6 6 6 6 6 6
## 2 6 6 6 6 6 6 6 6 6 6
## 3 6 6 6 6 6 6 6 6 6 6ggplot(data = da,
mapping = aes(x = prog,
y = vol,
color = bloco,
group = bloco)) +
geom_point() +
stat_summary(mapping = aes(group = 1),
geom = "line",
fun.y = "mean")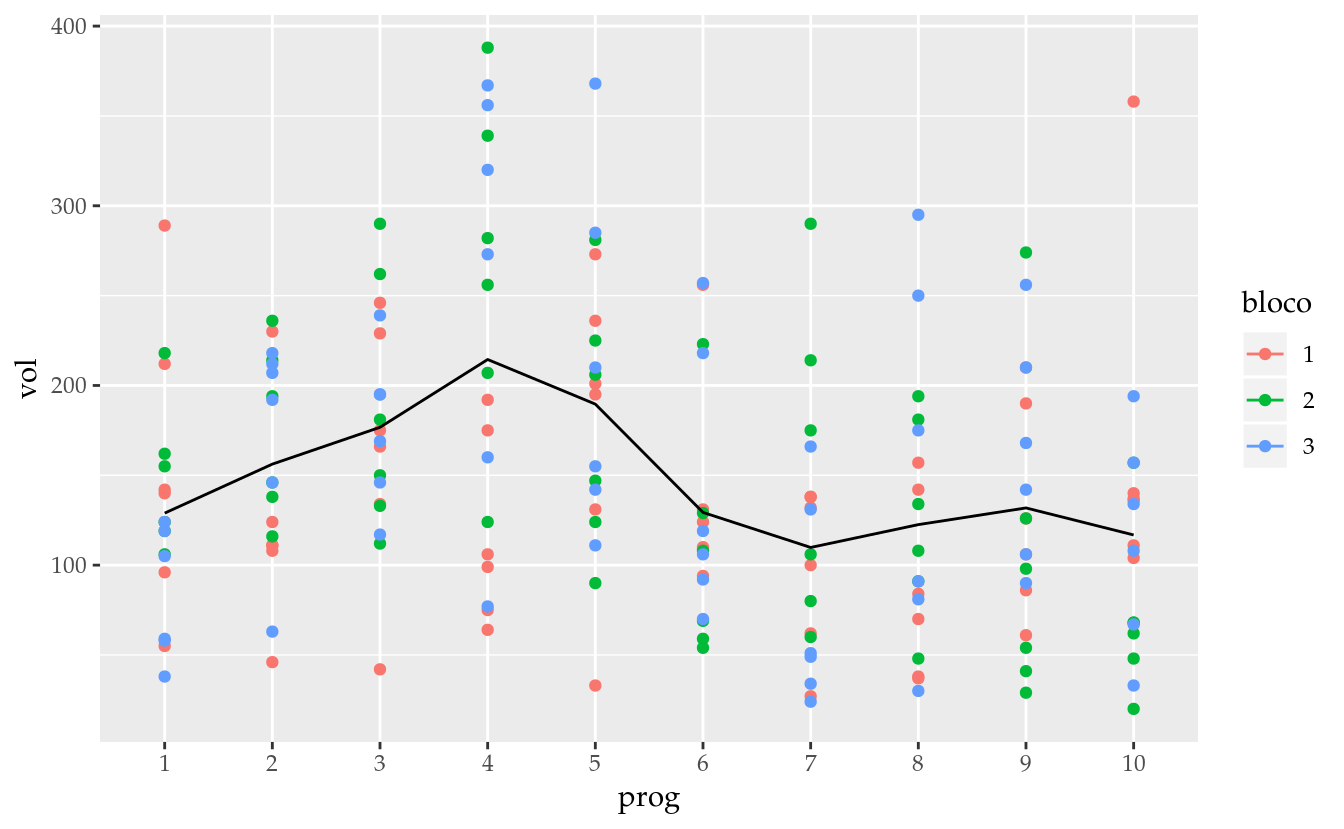
# Para fazer a análise com médias das repetições e passar o número de
# plantas como peso.
db <- da %>%
group_by(bloco, prog) %>%
summarise(n_plantas = sum(is.finite(vol)),
vol_medio = mean(vol, na.rm = TRUE))
head(db)## # A tibble: 6 x 4
## # Groups: bloco [1]
## bloco prog n_plantas vol_medio
## <fct> <fct> <int> <dbl>
## 1 1 1 6 156.
## 2 1 2 6 122.
## 3 1 3 6 165.
## 4 1 4 6 118.
## 5 1 5 6 178.
## 6 1 6 6 137.# Modelo de dois extratos: parcela e planta dentro de parcela.
# O termo `bloco:prog` representa cada parcela.
m0 <- aov(vol ~ bloco + prog + Error(parcela/plant), data = da)
summary(m0)##
## Error: parcela
## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 2 12988 6494 0.653 0.5323
## prog 9 199609 22179 2.231 0.0705 .
## Residuals 18 178970 9943
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Error: parcela:plant
## Df Sum Sq Mean Sq F value Pr(>F)
## Residuals 150 764157 5094# Modelo de extrato de parcela mas com totais e tamanho de amostra.
m1 <- aov(vol_medio ~ bloco + prog, data = db, weights = n_plantas)
summary(m1)## Df Sum Sq Mean Sq F value Pr(>F)
## bloco 2 12988 6494 0.653 0.5323
## prog 9 199609 22179 2.231 0.0705 .
## Residuals 18 178970 9943
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# ATTENTION: o modelo com 2 extratos é interessante para entender os
# componentes de variância. Essa informação é útil para planejar o
# experimento: definir tamanho de unidade experimental, número de
# observações por parcela, etc.
# As inferências acima só passam ser valiadas após confirmação de não
# haver afastamento dos pressupostos.
par(mfrow = c(2, 2))
plot(m1)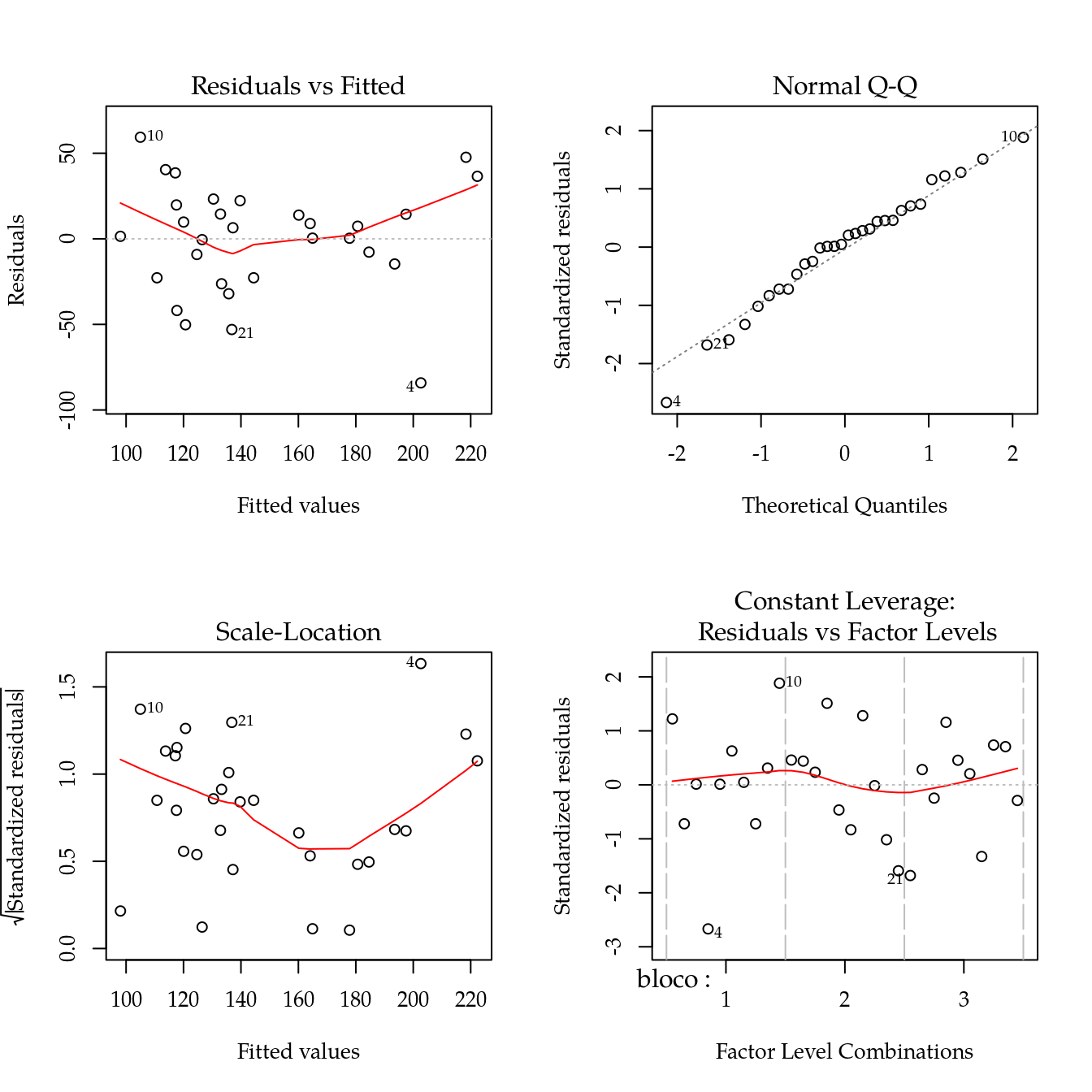
layout(1)
# anova(m0)
Anova(m1)## Anova Table (Type II tests)
##
## Response: vol_medio
## Sum Sq Df F value Pr(>F)
## bloco 12988 2 0.6531 0.53231
## prog 199609 9 2.2306 0.07046 .
## Residuals 178970 18
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1summary.lm(m1)##
## Call:
## aov(formula = vol_medio ~ bloco + prog, data = db, weights = n_plantas)
##
## Residuals:
## Min 1Q Median 3Q Max
## -206.125 -50.817 9.703 45.224 145.377
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 117.1500 25.7459 4.550 0.000248 ***
## bloco2 15.7167 18.2051 0.863 0.399324
## bloco3 19.6667 18.2051 1.080 0.294278
## prog2 27.2778 33.2378 0.821 0.422561
## prog3 47.7778 33.2378 1.437 0.167746
## prog4 85.5000 33.2378 2.572 0.019180 *
## prog5 60.6667 33.2378 1.825 0.084606 .
## prog6 0.3889 33.2378 0.012 0.990794
## prog7 -19.1111 33.2378 -0.575 0.572421
## prog8 -6.3889 33.2378 -0.192 0.849724
## prog9 2.8889 33.2378 0.087 0.931698
## prog10 -12.1667 33.2378 -0.366 0.718595
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 99.71 on 18 degrees of freedom
## Multiple R-squared: 0.5429, Adjusted R-squared: 0.2636
## F-statistic: 1.944 on 11 and 18 DF, p-value: 0.1016# Médias marginais ajustdas.
emm <- emmeans(m1, specs = ~prog)
emm## prog emmean SE df lower.CL upper.CL
## 1 129 23.5 18 79.6 178
## 2 156 23.5 18 106.8 206
## 3 177 23.5 18 127.3 226
## 4 214 23.5 18 165.1 264
## 5 190 23.5 18 140.2 239
## 6 129 23.5 18 80.0 179
## 7 110 23.5 18 60.5 159
## 8 123 23.5 18 73.2 172
## 9 132 23.5 18 82.5 181
## 10 117 23.5 18 67.4 166
##
## Results are averaged over the levels of: bloco
## Confidence level used: 0.95# Comparações múltiplas, contrastes de Tukey.
# Método de correção de p-valor: false discovey rate.
tk_fdr <- summary(glht(m1, linfct = mcp(prog = "Tukey")),
test = adjusted(type = "fdr"))
# Resumo compacto com letras.
cld(tk_fdr, level = 0.1, decreasing = TRUE)## 1 2 3 4 5 6 7 8 9 10
## "a" "a" "a" "a" "a" "a" "a" "a" "a" "a"# Teste HSD de Tukey.
tk_hsd <- HSD.test(m1, trt = "prog", alpha = 0.1)
tk_hsd$statistics## MSerror Df Mean CV MSD
## 9942.754 18 147.6278 67.54376 262.1242tk_hsd$parameters## test name.t ntr StudentizedRange alpha
## Tukey prog 10 4.553176 0.1results <- tk_hsd$groups %>%
rownames_to_column(var = "prog") %>%
mutate(groups = str_trim(as.character(groups)))
results <- inner_join(results, as.data.frame(emm))## Joining, by = "prog"# Gráfico de segmentos para as estimativas intervalares.
ggplot(data = results,
mapping = aes(x = reorder(prog, vol_medio), y = vol_medio)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lower.CL, ymax = upper.CL),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s",
vol_medio, groups)),
angle = 90,
vjust = 0,
nudge_x = -0.1)+
labs(y = expression("Volume de madeira por árvore" ~ (m^3 ~ 10^4)),
x = "Progênie")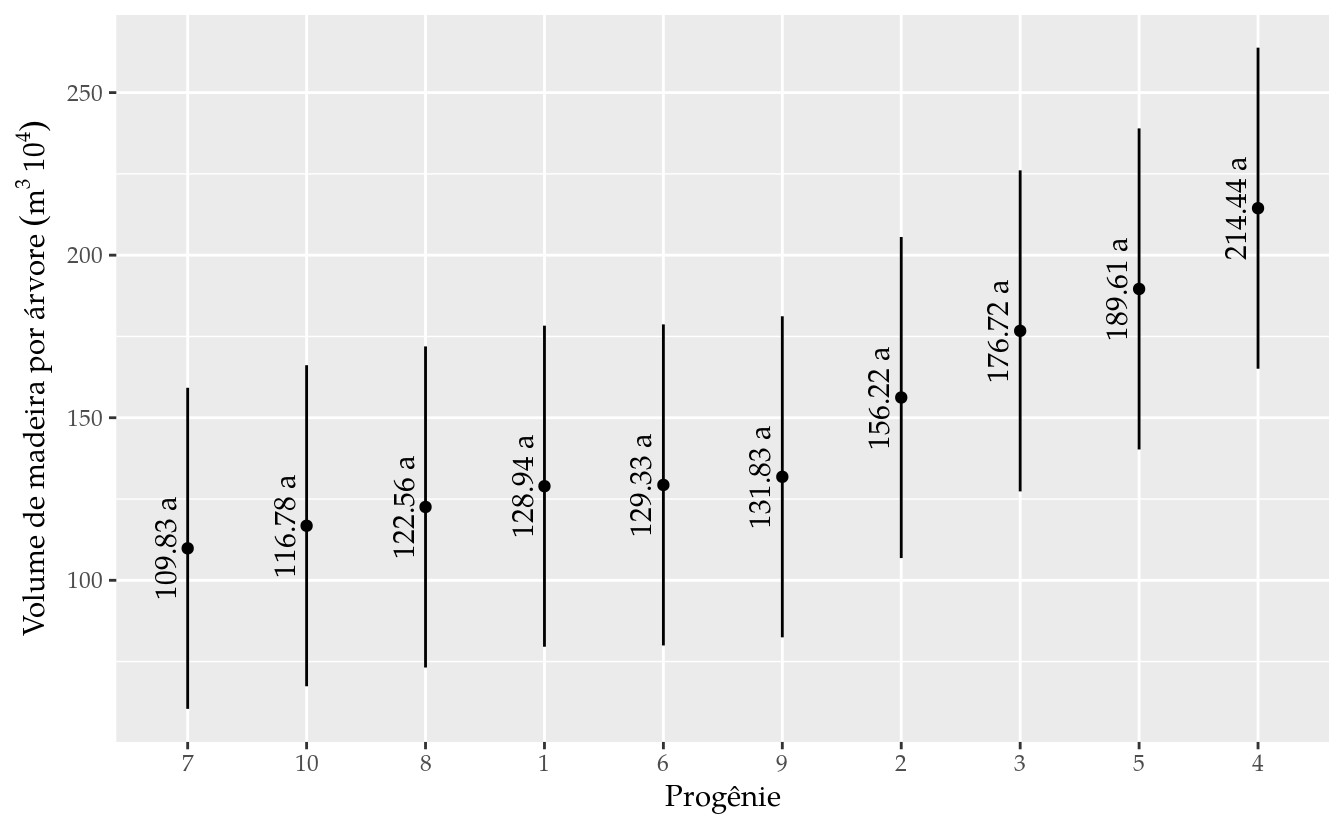
Referências Bibliográficas
BANZATTO, D. A.; KRONKA, S. D. Experimentação agrícola. 4th ed. Jaboticabal, SP: Funep, 2013.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani