Capítulo 10 Fatorial duplo com dois tratamentos adicionais
10.1 Construção com dados simulados
Serão usados os pacotes car, multcomp e tidyverse pelas mesmas razões descritas no capítulo anterior a esse no qual a análise de um experimento fatorial com um tratamento adicional foi ilustrada. Recomenda-se, para melhor entendimento do presente capítulo, que o anterior tenha sido lido.
# ATTENTION: Limpa espaço de trabalho.
rm(list = objects())
# Carrega pacotes.
library(car) # linearHypothesis()
library(multcomp) # glht()
library(tidyverse)Para demonstrar o procedimento dessa análise, será usado um conjunto de dados artificial construido por simulação. Uma parte importantíssima da prepração dos dados é fazer com que os pontos adicionais sejam os últimos níveis dos fatores em todos os fatores. Como variável resposta colocaremos apenas um ruído branco gaussiano. Conforme a tabela de ocorrência cruzada dos pontos experimentais, tem-se com esses dados simulados, um experimento com estrutura fatorial de 2 \(\times\) 4 mais 2 tratamentos adicionais.
# Criando um conjunto de dados artificial.
da <- rbind(
# Parte fatorial.
crossing(A = as.character(1:2),
B = as.character(1:4),
r = 1:3),
# Parte adicional. P: positivo, N: negativo.
tibble(A = "P", B = "P", r = 1:3),
tibble(A = "N", B = "N", r = 1:3))
# Converte fatores experimentais qualitativos para classe
# correspondente. IMPORTANT: para o procedimento que será adotado, é
# preciso que o tratamento adicional seja o último nível.
da <- mutate(da,
A = fct_relevel(factor(A), c("N", "P"), after = Inf),
B = fct_relevel(factor(B), c("N", "P"), after = Inf))
# Adiciona uma resposta que é apenas ruído branco.
set.seed(18900217) # Nascimento de Fisher.
da$y <- rnorm(nrow(da))
str(da)## Classes 'tbl_df', 'tbl' and 'data.frame': 30 obs. of 4 variables:
## $ A: Factor w/ 4 levels "1","2","N","P": 1 1 1 1 1 1 1 1 1 1 ...
## $ B: Factor w/ 6 levels "1","2","3","4",..: 1 1 1 2 2 2 3 3 3 4 ...
## $ r: int 1 2 3 1 2 3 1 2 3 1 ...
## $ y: num -0.6174 -0.5028 -0.7863 0.6474 -0.0393 ...# Estrutura de um fatorial 2 x 4 + 1 com 3 repetições.
xtabs(~A + B, data = da)## B
## A 1 2 3 4 N P
## 1 3 3 3 3 0 0
## 2 3 3 3 3 0 0
## N 0 0 0 0 3 0
## P 0 0 0 0 0 3Obtemos efeitos não estimados quando ajustamos um modelo utilizando a função lm(), uma vez que não existem todos os pontos experimentais do experimento fatorial completo. Isso porque a chamada da model.matrix() cria a matriz de delineamento com todos os termos de interação (abordagem maximal) e eles só serão estimados se houverem observações para todos os pontos experimentais do fatorial completo.
É exatamente este aspecto da análise que a dificulta: ser um fatorial incompleto. O que será mostrado aqui são formas de contornar esta complicação para que a análise possa ser feita da forma mais reproduzível possível.
# ATTENTION: alguns parâmetros não serão estimados porque são baseados
# em pontos experimentais que não existem.
m0 <- lm(y ~ A * B, data = da)
cbind(coef(m0))## [,1]
## (Intercept) -0.6354965
## A2 0.3253692
## AN 0.4800888
## AP 0.6139738
## B2 1.4495451
## B3 -0.1583176
## B4 0.4258809
## BN NA
## BP NA
## A2:B2 -1.6920751
## AN:B2 NA
## AP:B2 NA
## A2:B3 -0.8209788
## AN:B3 NA
## AP:B3 NA
## A2:B4 -0.1108463
## AN:B4 NA
## AP:B4 NA
## A2:BN NA
## AN:BN NA
## AP:BN NA
## A2:BP NA
## AN:BP NA
## AP:BP NAO uso dos contrastes de Helmert foi um facilitador para a análise com um ponto adicional. Existem mais opções de contrastes/codificação quando se tem dois ou mais pontos adicionais. A mais adequada depende dos objetivos do experimento. Para a situação em mãos, podemos usar contrastes pra cada uma das seguintes hipóteses:
- Positivo vs (Fatorial + Negativo), Negativo vs Fatorial, entre pontos fatoriais.
- Negativo vs (Fatorial + Positivo), Positivo vs Fatorial, entre pontos fatoriais.
- Positivo vs Negativo, Fatorial vs (Positivo + Negativo), entre pontos fatoriais.
- Positivo vs Fatorial, Negativo vs Fatorial, entre pontos fatoriais.
Nas 4 opções existe as hipóteses chamadas de “entre pontos fatoriais” que são relacionadas ao efeito de A, de B e da interação A:B, com a A e B sendo os fatores experimentais. As cláusulas 1 e 2 são simétricas, trocando apenas Positivo e Negativo de lugar. A cláusula 3 estabelece o contraste entre os pontos adicionais e eles juntos contra os pontos fatoriais. O último caso contrasta cada ponto adicional com os pontos fatoriais.
É importante mencionar que destes 4 casos, apenas o último não produz um conjunto de contrastes ortogonal. Uma vez não sendo ortogonal, a estatística F do quadro de análise de variância (hipóteses sequenciais) poderá ser diferente da estatística F do teste de Wald (hipóteses marginais).
A escolha do conjunto de contrastes depende das hipóteses do experimento. Considere como exemplo que o experimento avalie dois herbicidas (fator A) e combinados com 4 formas de aplicação no cultivo de soja estudando a produtividade. O ponto adicional positivo ou testemunha positiva é a capina mecânica que não provoca intereferencias fisiológicos na planta. Portanto, espera-se que seja o tratamento mais produtivo. O ponto adicional negativo ou testemunha negativa é não fazer a capina, portanto, haverá competição das plantas daninhas com a cultura. Com isso, espera-se que seja o tratamento menos produtivo. Os pontos fatoriais, que são aplicações de herbicida de alguma forma, devem estabeler-se, em produtividade, entre as duas testemunhas.
Suponha que não existe diferença entre pontos fatoriais, ou seja, todas as combinações de herbicida com a forma de aplicação apresentaram a mesma produtividade (hipótese nula não rejeitada). Então o pesquisador deseja saber na sequência: i) o uso de herbicida difere de não fazer o controle (Negativo)? e ii) a capina manual (Positivo) é tão boa quando o uso dos herbicidas? Para isso, as hipóteses da cláusula 4 são as adequadas.
Agora que foi escolhido o conjunto de hipóteses, deve-se usar os contrastes correspondentes. Faremos modificação do constraste de Helmert para adequá-lo a cláusula 4 tanto para o fator A quanto para B.
# Atribuindo os constrastes via modificação do Helmert.
ctr_A <- C(da$A, contr = contr.helmert)
attr(ctr_A, "contrasts")[, nlevels(da$A) - 1] <- c(-1, -1, 0, 2)
ctr_B <- C(da$B, contr = contr.helmert)
attr(ctr_B, "contrasts")[, nlevels(da$B) - 1] <- c(-1, -1, -1, -1, 0, 4)
# Usa contrastes definidos pelo usuário para A e B.
contrasts(da$A) <- attr(ctr_A, "contrasts")
contrasts(da$B) <- attr(ctr_B, "contrasts")
# Criação da matriz de delineamento.
X_full <- model.matrix(~A * B, data = da)
attr(X_full, "contrasts")## $A
## [,1] [,2] [,3]
## 1 -1 -1 -1
## 2 1 -1 -1
## N 0 2 0
## P 0 0 2
##
## $B
## [,1] [,2] [,3] [,4] [,5]
## 1 -1 -1 -1 -1 -1
## 2 1 -1 -1 -1 -1
## 3 0 2 -1 -1 -1
## 4 0 0 3 -1 -1
## N 0 0 0 4 0
## P 0 0 0 0 4# Ajuste do modelo com aov() para usar `split =`.
m0 <- aov(y ~ 0 + X_full, data = da)
cbind(coef(m0))## [,1]
## X_full(Intercept) -0.22999374
## X_fullA1 -0.16530291
## X_fullA2 0.03729303
## X_fullA3 0.10423551
## X_fullB1 0.30175379
## X_fullB2 -0.29018693
## X_fullB3 0.08972273
## X_fullA1:B1 -0.42301878
## X_fullA1:B2 0.00417646
## X_fullA1:B3 0.09085479O modelo m0 foi ajustado usando-se os contrastes definidos pelo usuário motivados pela cláusula 4. Para particionar as somas de quadrados, usamos novamente o método summary() com o parâmetro split. A lista indicando os termos é passada.
# Coeficientes e seus efeitos.
ef <- list("fat_vs_N" = 3,
"fat_vs_P" = 4,
"A" = 2,
"B" = 5:7,
"A:B" = 8:10)
summary(m0, split = list(X_full = ef))## Df Sum Sq Mean Sq F value Pr(>F)
## X_full 10 11.488 1.1488 1.325 0.283
## X_full: fat_vs_N 1 0.213 0.2128 0.245 0.626
## X_full: fat_vs_P 1 0.199 0.1987 0.229 0.637
## X_full: A 1 0.656 0.6558 0.757 0.395
## X_full: B 3 4.704 1.5679 1.809 0.178
## X_full: A:B 3 2.742 0.9141 1.055 0.390
## Residuals 20 17.336 0.8668NOTE: é importante enfatizar que a escolha que fizemos de hipóteses (cláusula 4) não corresponde a um conjunto de hipóteses lineares ortogonais. Portanto, a estatística F da partição do quadro de análise de variância não será igual a estatística F do teste de Wald para alguma das hipóteses.
# Produto interno nulo entre colunas -> hipóteses lineares ortogonais.
C(da$A, contr = contr.helmert)## [1] 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 P P P N N N
## attr(,"contrasts")
## [,1] [,2] [,3]
## 1 -1 -1 -1
## 2 1 -1 -1
## N 0 2 -1
## P 0 0 3
## Levels: 1 2 N PC(da$A, contr = contr.treatment)## [1] 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 P P P N N N
## attr(,"contrasts")
## 2 3 4
## 1 0 0 0
## 2 1 0 0
## N 0 1 0
## P 0 0 1
## Levels: 1 2 N PC(da$A, contr = contr.SAS)## [1] 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 P P P N N N
## attr(,"contrasts")
## 1 2 3
## 1 1 0 0
## 2 0 1 0
## N 0 0 1
## P 0 0 0
## Levels: 1 2 N PC(da$A, contr = contr.poly)## [1] 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 P P P N N N
## attr(,"contrasts")
## .L .Q .C
## 1 -0.6708204 0.5 -0.2236068
## 2 -0.2236068 -0.5 0.6708204
## N 0.2236068 -0.5 -0.6708204
## P 0.6708204 0.5 0.2236068
## Levels: 1 2 N P# Produto interno não nulo entre colunas.
contrasts(da$A)## [,1] [,2] [,3]
## 1 -1 -1 -1
## 2 1 -1 -1
## N 0 2 0
## P 0 0 2Conforme descrito no capítulo anterior, para modelos mais gerais não tem-se uma função que se comporta como a aov(). Então reproduziremos os resultados usando o objeto de classe lm() empregado na multcomp::glht() e na car::linearHypothesis(). O primeiro passo é ajustar o modelo com a lm(). Lembre-se que os contrastes já foram definidos para os fatores A e B.
# A `aov()` já abandona parâmetros não estimados do vetor. Mas para
# modelos de outras classes, terá que ser feito manualmente.
X_ok <- X_full[, str_replace(names(coef(m0)), "X_full", "")]
# Modelo linear com matriz onde todos efeitos são estimáveis.
m1 <- lm(y ~ 0 + X_ok, data = da)
cbind(lm = coef(m1), aov = coef(m0))## lm aov
## X_ok(Intercept) -0.22999374 -0.22999374
## X_okA1 -0.16530291 -0.16530291
## X_okA2 0.03729303 0.03729303
## X_okA3 0.10423551 0.10423551
## X_okB1 0.30175379 0.30175379
## X_okB2 -0.29018693 -0.29018693
## X_okB3 0.08972273 0.08972273
## X_okA1:B1 -0.42301878 -0.42301878
## X_okA1:B2 0.00417646 0.00417646
## X_okA1:B3 0.09085479 0.09085479Uma forma equivalente de exibir as mesmas partições obtidas com a summary(..., split) é passar a matriz de delineamento dividida nas colunas mantendo juntos os parâmetros de um mesmo termo do modelo. Com isso, reproduz-se os resultados da chamada da aov(). O importante é saber como criar a matriz (conjunto de contrastes corretos e eliminação das colunas de efeitos não estimáveis) e como particioná-la corretamente.
anova(lm(y ~
X_ok[, 3, drop = FALSE] + # fat_vs_N
X_ok[, 4, drop = FALSE] + # fat_vs_P
X_ok[, 2, drop = FALSE] + # A
X_ok[, 5:7, drop = FALSE] + # B
X_ok[, 8:10, drop = FALSE], # A:B
data = da))## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## X_ok[, 3, drop = FALSE] 1 0.2128 0.21280 0.2455 0.6257
## X_ok[, 4, drop = FALSE] 1 0.1987 0.19868 0.2292 0.6373
## X_ok[, 2, drop = FALSE] 1 0.6558 0.65580 0.7566 0.3947
## X_ok[, 5:7, drop = FALSE] 3 4.7038 1.56793 1.8088 0.1780
## X_ok[, 8:10, drop = FALSE] 3 2.7423 0.91410 1.0545 0.3904
## Residuals 20 17.3364 0.86682A seguir, a estatística F do teste de Wald será obtida com a multcomp::cftest() e com a car::linearHypothesis().
A multcomp::cftest() precisa que os efeitos sob hipótese sejam informados pela posição que ocupam dentro do vetor ou pelo nome. Com isso, ela é capaz de testar hipóteses sob os efeitos estimados mas não sob combinações lineares gerais com eles.
# `multcomp::cftest()` é aplicável a uma ampla classe de modelos.
cftest(m1, parm = ef[[1]], test = Ftest()) # Fat. vs Neg.##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## X_okA2 == 0 0.03729
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 0.02934 1 20 0.8657cftest(m1, parm = ef[[2]], test = Ftest()) # Fat. vs Pos.##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## X_okA3 == 0 0.1042
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 0.2292 1 20 0.6373cftest(m1, parm = ef[[3]], test = Ftest()) # A.##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## X_okA1 == 0 -0.1653
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 0.7566 1 20 0.3947cftest(m1, parm = ef[[4]], test = Ftest()) # B.##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## X_okB1 == 0 0.30175
## X_okB2 == 0 -0.29019
## X_okB3 == 0 0.08972
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 1.809 3 20 0.178cftest(m1, parm = ef[[5]], test = Ftest()) # A:B.##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## X_okA1:B1 == 0 -0.423019
## X_okA1:B2 == 0 0.004176
## X_okA1:B3 == 0 0.090855
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 1.055 3 20 0.3904A car::linearHypothesis() é mais genérica pois recebe uma matriz com coeficientes que especificam a hipótese em questão. Se por um lado a criação da matriz demanda mais atenção, com uma matriz pode-se testar hipóteses mais gerais.
# Matriz de hipóteses lineares (linear hypothesis matrix).
m <- diag(length(coef(m1)))
lhm <- ef
for (i in 1:length(ef)) {
lhm[[i]] <- m[ef[[i]], , drop = FALSE]
}
lhm## $fat_vs_N
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
## [1,] 0 0 1 0 0 0 0 0 0 0
##
## $fat_vs_P
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
## [1,] 0 0 0 1 0 0 0 0 0 0
##
## $A
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
## [1,] 0 1 0 0 0 0 0 0 0 0
##
## $B
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
## [1,] 0 0 0 0 1 0 0 0 0 0
## [2,] 0 0 0 0 0 1 0 0 0 0
## [3,] 0 0 0 0 0 0 1 0 0 0
##
## $`A:B`
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
## [1,] 0 0 0 0 0 0 0 1 0 0
## [2,] 0 0 0 0 0 0 0 0 1 0
## [3,] 0 0 0 0 0 0 0 0 0 1# `car::linearHypothesis()` é aplicável a uma ampla classe de modelos.
linearHypothesis(m1, hypothesis.matrix = lhm[[1]], test = "F")## Linear hypothesis test
##
## Hypothesis:
## X_okA2 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X_ok
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 21 17.362
## 2 20 17.336 1 0.025431 0.0293 0.8657linearHypothesis(m1, hypothesis.matrix = lhm[[2]], test = "F")## Linear hypothesis test
##
## Hypothesis:
## X_okA3 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X_ok
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 21 17.535
## 2 20 17.336 1 0.19867 0.2292 0.6373linearHypothesis(m1, hypothesis.matrix = lhm[[3]], test = "F")## Linear hypothesis test
##
## Hypothesis:
## X_okA1 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X_ok
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 21 17.992
## 2 20 17.336 1 0.6558 0.7566 0.3947linearHypothesis(m1, hypothesis.matrix = lhm[[4]], test = "F")## Linear hypothesis test
##
## Hypothesis:
## X_okB1 = 0
## X_okB2 = 0
## X_okB3 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X_ok
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 23 22.040
## 2 20 17.336 3 4.7038 1.8088 0.178linearHypothesis(m1, hypothesis.matrix = lhm[[5]], test = "F")## Linear hypothesis test
##
## Hypothesis:
## X_okA1:B1 = 0
## X_okA1:B2 = 0
## X_okA1:B3 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X_ok
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 23 20.079
## 2 20 17.336 3 2.7423 1.0545 0.3904As comparações múltiplas de médias dentro de cada fator são feitas com os códigos a seguir.
# Pontos experimentais únicos de A * B.
i <- da %>% select(A, B) %>% duplicated()
X_un <- X_ok[!i, ] # Estimativas.
pts <- da[!i, c("A", "B")] # Pontos experimentais.
cbind(pts, "|" = "|", X_un)## A B | (Intercept) A1 A2 A3 B1 B2 B3 A1:B1 A1:B2 A1:B3
## 1 1 1 | 1 -1 -1 -1 -1 -1 -1 1 1 1
## 4 1 2 | 1 -1 -1 -1 1 -1 -1 -1 1 1
## 7 1 3 | 1 -1 -1 -1 0 2 -1 0 -2 1
## 10 1 4 | 1 -1 -1 -1 0 0 3 0 0 -3
## 13 2 1 | 1 1 -1 -1 -1 -1 -1 -1 -1 -1
## 16 2 2 | 1 1 -1 -1 1 -1 -1 1 -1 -1
## 19 2 3 | 1 1 -1 -1 0 2 -1 0 2 -1
## 22 2 4 | 1 1 -1 -1 0 0 3 0 0 3
## 25 P P | 1 0 0 2 0 0 0 0 0 0
## 28 N N | 1 0 2 0 0 0 0 0 0 0all_pairwise <- function(L, collapse = "vs", row_names = rownames(L)) {
ij <- combn(x = 1:nrow(L), m = 2)
K <- apply(X = ij,
MARGIN = 2,
FUN = function(i) { L[i[1], ] - L[i[2], ] })
K <- t(K)
if (is.null(row_names)) {
nm <- 1:nrow(L)
} else {
nm <- row_names
}
rownames(K) <- apply(ij,
MARGIN = 2,
FUN = function(i) {
paste(nm[i], collapse = collapse)
})
return(K)
}
# Contrastes dois a dois (pairwise) entre níveis de A.
L_A <- do.call(rbind, by(X_un, pts$A, colMeans))
K_A <- all_pairwise(head(L_A, n = -2))
summary(glht(m1, linfct = K_A))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.3306 0.3801 0.87 0.395
## (Adjusted p values reported -- single-step method)# Contrastes dois a dois (pairwise) entre níveis de B.
L_B <- do.call(rbind, by(X_un, pts$B, colMeans))
K_B <- all_pairwise(head(L_B, n = -2))
summary(glht(m1, linfct = K_B))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.6035 0.5375 -1.123 0.680
## 1vs3 == 0 0.5688 0.5375 1.058 0.718
## 1vs4 == 0 -0.3705 0.5375 -0.689 0.900
## 2vs3 == 0 1.1723 0.5375 2.181 0.163
## 2vs4 == 0 0.2330 0.5375 0.434 0.972
## 3vs4 == 0 -0.9393 0.5375 -1.747 0.327
## (Adjusted p values reported -- single-step method)# Estudo da interação: entre níveis de B dentro de A.
L_BinA <- by(X_un, pts$A, FUN = as.matrix)
L_BinA <- lapply(L_BinA, "rownames<-", NULL)
lapply(head(L_BinA, n = -2),
FUN = function(s) {
summary(glht(m1, linfct = all_pairwise(s)))
})## $`1`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -1.4495 0.7602 -1.907 0.257
## 1vs3 == 0 0.1583 0.7602 0.208 0.997
## 1vs4 == 0 -0.4259 0.7602 -0.560 0.943
## 2vs3 == 0 1.6079 0.7602 2.115 0.182
## 2vs4 == 0 1.0237 0.7602 1.347 0.545
## 3vs4 == 0 -0.5842 0.7602 -0.768 0.868
## (Adjusted p values reported -- single-step method)
##
##
## $`2`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.2425 0.7602 0.319 0.988
## 1vs3 == 0 0.9793 0.7602 1.288 0.581
## 1vs4 == 0 -0.3150 0.7602 -0.414 0.975
## 2vs3 == 0 0.7368 0.7602 0.969 0.768
## 2vs4 == 0 -0.5576 0.7602 -0.733 0.882
## 3vs4 == 0 -1.2943 0.7602 -1.703 0.348
## (Adjusted p values reported -- single-step method)# Estudo da interação: entre níveis de A dentro de B.
L_AinB <- by(X_un, pts$B, FUN = as.matrix)
L_AinB <- lapply(L_AinB, "rownames<-", NULL)
lapply(head(L_AinB, n = -2),
FUN = function(s) {
summary(glht(m1, linfct = all_pairwise(s)))
})## $`1`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.3254 0.7602 -0.428 0.673
## (Adjusted p values reported -- single-step method)
##
##
## $`2`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 1.3667 0.7602 1.798 0.0873 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)
##
##
## $`3`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.4956 0.7602 0.652 0.522
## (Adjusted p values reported -- single-step method)
##
##
## $`4`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.2145 0.7602 -0.282 0.781
## (Adjusted p values reported -- single-step method)# Contraste do ponto adicional Negativo contra os pontos fatoriais.
K <- rbind(colMeans(L_A[c("1", "2"), ]) - L_A["N", ])
summary(glht(m1, linfct = K))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1 == 0 -0.2161 0.5701 -0.379 0.709
## (Adjusted p values reported -- single-step method)# Contraste do ponto adicional Positivo contra os pontos fatoriais.
K <- rbind(colMeans(L_A[c("1", "2"), ]) - L_A["P", ])
summary(glht(m1, linfct = K))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1 == 0 -0.3500 0.5701 -0.614 0.546
## (Adjusted p values reported -- single-step method)Apenas para verificação, vamos obter as médias amostrais. O experimento que foi simulado é balanceado e com efeitos ortogonais (NOTE: experimento com efeitos ortogonais é diferente de hipóteses lineares ortogonais), as médias amostrais serão estimativas pontuais iguais às médias ajustadas.
# Diferenças entre médias amostrais apenas para verificação.
# Diferenças entre médias marginais dos níveis de A.
da %>% group_by(A) %>% summarise(m = -mean(y)) %>%
pull(m) %>% head(n = -2) %>% outer(., ., "-") %>% as.dist()## 1
## 2 0.3306058# Diferenças entre médias marginais dos níveis de B.
da %>% group_by(B) %>% summarise(m = -mean(y)) %>%
pull(m) %>% head(n = -2) %>% outer(., ., "-") %>% as.dist()## 1 2 3
## 2 -0.6035076
## 3 0.5688070 1.1723146
## 4 -0.3704578 0.2330498 -0.9392648# Diferença entre média dos pontos fatoriais vs cada ponto adicional.
x <- ifelse(da$A %in% head(levels(da$A), n = -2),
"fat",
as.character(da$A))
da %>% group_by(!!x) %>%
summarise(m = -mean(y)) %>%
pull(m) %>% outer(., ., "-") %>% as.dist()## 1 2
## 2 -0.2161146
## 3 -0.3499996 -0.133885010.2 Análise do experimento com fusarium em Zimmermann
Os dados que usaremos para aplicar o procedimento acima estão em ZIMMERMANN (2004). Trata-se de um experimento fatorial \(3 \times 3 + 2\), ou seja, um fatorial duplo com mais dois pontos adicionais. O experimento foi realizado no delineamento inteiramente casualizado.
A variável resposta é o número de sementes infectadas com fusarium de um total de 40 sementes expostas (nsi). Portanto, a resposta pode ser analisada considerando distribuição binomial se forem razoáveis as suposições para tal. Digo razoáveis porque nem todo experimento que produza uma resposta assim – um número inteiro no numerador tendo outro número inteiro no denominador – terá distribuição binomial. Existem suposições, como independência entre os ensaios de Bernoulli e probabilidade de sucesso homogênea em cada ponto experimental. Geralmente as sementes apresentam tamanho variável, estão em condições fisiológicas diferentes e são colocadas para germinar todas juntas em um ambiente compartilhado (gerbox, faixa de canteiro, bandeja de germinação). A probabilidade de sucesso pode mudar com o estado/tamanho/idade da semente dentro de um mesma unidade experimental, o que faz com que a variância observada na resposta seja maior que aquela esperada pela distribuição binomial. Além do mais, estarem germinando em um ambiente compartilhado pode provocar não independência das germinações individuais das sementes, ou seja, uma semente infectada pode produzir inóculo que aumenta a chance de infecção das demais sementes da mesma repetição. Independência entre os ensaios de Bernoulli e taxa de sucesso constante são suposições para a distribuição binomial.
É bastante comum de ver em livros de Estatística Experimental o uso da transformação arco seno da raíz quadrada para dados de proporção como estes. Essa transformação é uma transformação estabilizadora da variância para dados de distribuição binomial2. Lembre-se que uma variável aleatória com distribuição binomial de parâmetros \(n\) e \(p\) tem variância \(p(1 - p)\). A transformação \(f(x) = \arcsin(\sqrt(x))\) é empregada para levar os dados para uma escala com maior homogeneidade de variâncias e assim realizar análise assumindo-se distribuição gaussiana. Hoje tem-se os modelos lineares generalizados (GLM) disponíveis para a análise de dados de proporção como este, o que dimunui a justificativa para uso de transformações estabilizadoras da variância. De qualquer forma, com propósito didático, será feita a análise com a variável transformada e depois usando GLM.
Os dados que vamos usar estão no objeto ZimmermannTb9.22 do pacote labestData. Consulte a documentação para detalhes. A variável y é a versão transformada da proporção amostral de sementes infectadas.
library(labestData)
if (interactive()) {
help(ZimmermannTb9.22, help_type = "html")
}
# Cria a variável transformada pelo arco seno da raiz quadrada da
# proporção amostral.
tb <- ZimmermannTb9.22
tb$y <- asin(sqrt(tb$nsi/40))
str(tb)## 'data.frame': 55 obs. of 5 variables:
## $ trat : Factor w/ 11 levels "benlate antes",..: 3 1 2 6 4 5 9 7 8 10 ...
## $ fung : Factor w/ 5 levels "benlate","captam",..: 1 1 1 2 2 2 3 3 3 4 ...
## $ aplic: Factor w/ 5 levels "puro","antes",..: 1 2 3 1 2 3 1 2 3 4 ...
## $ nsi : int 5 1 3 7 3 6 0 0 0 6 ...
## $ y : num 0.361 0.159 0.277 0.432 0.277 ...# Contagem dos pontos experimentais.
xtabs(~fung + aplic, data = tb)## aplic
## fung puro antes misturado polimero testemunha
## benlate 5 5 5 0 0
## captam 5 5 5 0 0
## derosal 5 5 5 0 0
## polimero 0 0 0 5 0
## testemunha 0 0 0 0 5# Proporção amostral de sementes infectadas em cada ponto experimental.
with(tb, tapply(nsi/40, list(fung, aplic), FUN = mean))## puro antes misturado polimero testemunha
## benlate 0.025 0.010 0.050 NA NA
## captam 0.185 0.040 0.135 NA NA
## derosal 0.010 0.005 0.000 NA NA
## polimero NA NA NA 0.19 NA
## testemunha NA NA NA NA 0.225# Análise exploratória.
ggplot(data = tb,
mapping = aes(x = aplic, y = y)) +
facet_grid(facets = . ~ fung, scale = "free", space = "free") +
geom_jitter(alpha = 0.5, width = 0.1, height = 0)
Neste experimento o fator fungicida (fung) corresponde ao produto usado para controle de fusarium, com 3 níveis qualitativos: Benlate, Captam e Derosal. O fator aplicação (aplic) indica a forma de aplicação de fungicida quando ao uso associado de polímero, também com 3 níveis: fungicida puro, fungicida antes do polímero e fungicida misturado ao polímero. Os pontos adicionais são a testemunha sem controle de fusarium e a aplicação isolada de polímero.
Para a análise do fatorial com tratamento adicional temos que escolher o conjunto de hipóteses mais adequado. Em ZIMMERMANN (2004), é feita a análise considerando as hipóteses da cláusula 3 que, neste caso, será: i) testemunha vs polímero, ii) (testemunha + polímero) vs pontos fatoriais e iii) entre pontos fatoriais. A hipótese (i) deseja saber se a aplicação de polímero altera a infecção de fusarium. A hipótese (iii) deseja avaliar a existência de efeito do fungicída, da forma de aplicação, ou da interação entre eles. A hipótese (ii) é, no caso de (i) e (iii) não serem rejeitadas, se o controle conseguido com qualquer aplicação dos fungicidas difere das testemunhas. É necessário enfatizar que a interpretação das hipóteses é sequêncial.
Cada ponto experimental foi teve 5 repetições. A proporção média de sementes infectadas apresenta valor 0 para um ponto experimental (derosal:misturado). Essa característica é relevante para a análise com GLM binomial, portanto, será discutido adiante.
O diagrama de dispersão sugere existência de diferença entre os tratamentos para o controle de fusarium. Outro aspecto saliente é que existem muitos zeros observados. No ponto experimental fung = derosal & aplic = misturado foi observado zero em todas as unidades experimentais. Dois alertas decorrem disso: estimativa amostral de proporção de infecção é \(\hat{p} = 0\), ou seja, foram do espaço paramétrico de \(p \in (0, 1)\) da distribuição binomial e variância amostral igual 0. De qualquer forma, essa primeira análise assume distribuição gaussiana para a variável transformada.
As hipóteses que serão testadas já foram estabelecidas. Agora precisa-se criar o conjunto de contrastes que as represente. Isso é feito no bloco a seguir, seguido do ajuste do modelo. A 4º coluna da matriz de contrastes é para a hipótese (ii) e a 5º coluna é para a hipótese (i). Os coeficientes tem que somar sempre zero porque são contrastes. Cada conjunto de contrastes é ortogonal, ou seja, o produto interno das colunas é 0.
# Contrates para o fator fungicida.
ctr <- C(tb$fung, contr = contr.helmert)
attr(ctr, "contrasts")[, 3] <- c(-2, -2, -2, 3, 3)
attr(ctr, "contrasts")[, 4] <- c( 0, 0, 0, -1, 1)
contrasts(tb$fung) <- attr(ctr, "contrasts")
# Contrates para o fator aplicação.
ctr <- C(tb$aplic, contr = contr.helmert)
attr(ctr, "contrasts")[, 3] <- c(-2, -2, -2, 3, 3)
attr(ctr, "contrasts")[, 4] <- c( 0, 0, 0, -1, 1)
contrasts(tb$aplic) <- attr(ctr, "contrasts")
# Mostra que o produto interno é 0.
crossprod(contrasts(tb$fung))## [,1] [,2] [,3] [,4]
## [1,] 2 0 0 0
## [2,] 0 6 0 0
## [3,] 0 0 30 0
## [4,] 0 0 0 2# Criação da matriz de delineamento.
X_full <- model.matrix(~fung * aplic, data = tb)
attr(X_full, "contrasts")## $fung
## [,1] [,2] [,3] [,4]
## benlate -1 -1 -2 0
## captam 1 -1 -2 0
## derosal 0 2 -2 0
## polimero 0 0 3 -1
## testemunha 0 0 3 1
##
## $aplic
## [,1] [,2] [,3] [,4]
## puro -1 -1 -2 0
## antes 1 -1 -2 0
## misturado 0 2 -2 0
## polimero 0 0 3 -1
## testemunha 0 0 3 1# Ajuste do modelo.
m0 <- aov(y ~ 0 + X_full, data = tb)
cbind(coef(m0))## [,1]
## X_full(Intercept) 0.275826699
## X_fullfung1 0.105225294
## X_fullfung2 -0.060828875
## X_fullfung3 0.064274687
## X_fullfung4 0.021269956
## X_fullaplic1 -0.055225036
## X_fullaplic2 0.017711403
## X_fullfung1:aplic1 -0.075120225
## X_fullfung2:aplic1 0.024275859
## X_fullfung1:aplic2 -0.002016153
## X_fullfung2:aplic2 -0.015260595# Diagnóstico não seja discutido aqui para não sair do foco.
# par(mfrow = c(2, 2))
# plot(m0)
# layout(1)
# Para decompor as SQ.
ef <- list("ii) T+P vs Fat" = 4,
"i) T vs P" = 5,
"Fung" = 2:3,
"Aplic" = 6:7,
"Fung:Aplic" = 8:11)
summary(m0, split = list(X_full = ef))## Df Sum Sq Mean Sq F value Pr(>F)
## X_full 11 4.152 0.3775 30.697 < 2e-16 ***
## X_full: ii) T+P vs Fat 1 0.845 0.8450 68.719 1.55e-10 ***
## X_full: i) T vs P 1 0.005 0.0045 0.368 0.54726
## X_full: Fung 2 0.665 0.3326 27.047 2.19e-08 ***
## X_full: Aplic 2 0.120 0.0599 4.868 0.01230 *
## X_full: Fung:Aplic 4 0.190 0.0476 3.871 0.00886 **
## Residuals 44 0.541 0.0123
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1O quadro de análise de variância acima está igual ao disponível em ZIMMERMANN (2004) página 188, exceto por arredondamentos e ordem das linhas.
Pela inspeção do quadro de análise de variância, rejeita-se a hipótese nula de não interação entre fungicida e aplicação ao nível de 5%. Dessa forma, não interpreta-se os efeitos principais. Pelo quadro, não se rejeita a hipótese (i), ou seja, em termos práticos, não houve evidência de que polímero isolado tem efeito sobre a infecção de fusarium. A hipótese (ii) não faz sentido ser intepretada haja visto a existência de interação.
Agora que usamos a aov(), vamos ajustar o mesmo modelo com a lm() para fazermos o estudo da interação com multcomp::glht(). Usaremos uma matriz de delineamento apenas com colunas de parâmetros estimáveis.
X_ok <- X_full[, str_replace(names(coef(m0)), "X_full", "")]
m1 <- lm(y ~ 0 + X_ok, data = tb)
cbind(lm = coef(m1), aov = coef(m0))## lm aov
## X_ok(Intercept) 0.275826699 0.275826699
## X_okfung1 0.105225294 0.105225294
## X_okfung2 -0.060828875 -0.060828875
## X_okfung3 0.064274687 0.064274687
## X_okfung4 0.021269956 0.021269956
## X_okaplic1 -0.055225036 -0.055225036
## X_okaplic2 0.017711403 0.017711403
## X_okfung1:aplic1 -0.075120225 -0.075120225
## X_okfung2:aplic1 0.024275859 0.024275859
## X_okfung1:aplic2 -0.002016153 -0.002016153
## X_okfung2:aplic2 -0.015260595 -0.015260595Por razões didáticas, será feito o estudo da interação nos dois sentidos: comparar aplicações fixando o nível de fungicida e comparar fungicidas fixando a forma de aplicação. No entanto, apesar de sempre ser possível estudar a interação em todos os sentidos, na prática nem todos são igualmente relevantes ou de significado. Tente responder o que faz mais sentido da prática para o controle de fusarium:
- É primeiro feita a escolha do fungicida e depois a forma de aplicação?
- É primeiro feita a escolha da forma de aplicação e depois o fungicida?
- É possível escolher simultâneamente o fungicida e forma de aplicação.
Estou inclinado a pensar que a condição 1 é mais realista que a 2. Todavia, o caso 3 parece ser também apropriado. Para o caso 3 podemos comparar todos os 9 pontos da porção fatorial sem precisar fazer o estudo de um fator fixando-se os demais. Começarei por 1 e 2.
# Pontos experimentais únicos de A * B.
i <- tb %>% select(fung, aplic) %>% duplicated()
X_un <- X_ok[!i, ] # Estimativas.
pts <- tb[!i, c("fung", "aplic")] # Pontos experimentais.
cbind(pts, "|" = "|", unname(X_un))## fung aplic | 1 2 3 4 5 6 7 8 9 10 11
## 1 benlate puro | 1 -1 -1 -2 0 -1 -1 1 1 1 1
## 2 benlate antes | 1 -1 -1 -2 0 1 -1 -1 -1 1 1
## 3 benlate misturado | 1 -1 -1 -2 0 0 2 0 0 -2 -2
## 4 captam puro | 1 1 -1 -2 0 -1 -1 -1 1 -1 1
## 5 captam antes | 1 1 -1 -2 0 1 -1 1 -1 -1 1
## 6 captam misturado | 1 1 -1 -2 0 0 2 0 0 2 -2
## 7 derosal puro | 1 0 2 -2 0 -1 -1 0 -2 0 -2
## 8 derosal antes | 1 0 2 -2 0 1 -1 0 2 0 -2
## 9 derosal misturado | 1 0 2 -2 0 0 2 0 0 0 4
## 10 polimero polimero | 1 0 0 3 -1 0 0 0 0 0 0
## 11 testemunha testemunha | 1 0 0 3 1 0 0 0 0 0 0# Estudo dos níveis de aplicação dentro de fungicida.
L_AinF <- head(by(X_un, pts$fung, FUN = as.matrix), n = -2)
L_AinF <- lapply(L_AinF,
FUN = "rownames<-",
head(levels(tb$aplic), n = -2))
lapply(L_AinF,
FUN = function(s) {
K <- all_pairwise(s, collapse = " vs ")
summary(glht(m1, linfct = K))
})## $benlate
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## puro vs antes == 0 0.008761 0.070134 0.125 0.991
## puro vs misturado == 0 -0.100584 0.070134 -1.434 0.332
## antes vs misturado == 0 -0.109345 0.070134 -1.559 0.274
## (Adjusted p values reported -- single-step method)
##
##
## $captam
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## puro vs antes == 0 0.30924 0.07013 4.409 0.000183 ***
## puro vs misturado == 0 0.06175 0.07013 0.881 0.655305
## antes vs misturado == 0 -0.24749 0.07013 -3.529 0.002811 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)
##
##
## $derosal
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## puro vs antes == 0 0.01335 0.07013 0.190 0.980
## puro vs misturado == 0 0.04510 0.07013 0.643 0.797
## antes vs misturado == 0 0.03176 0.07013 0.453 0.893
## (Adjusted p values reported -- single-step method)# Estudo dos níveis de fungicida dentro de aplicação.
L_FinA <- head(by(X_un, pts$aplic, FUN = as.matrix), n = -2)
L_FinA <- lapply(L_FinA,
FUN = "rownames<-",
head(levels(tb$fung), n = -2))
lapply(L_FinA,
FUN = function(s) {
K <- all_pairwise(s, collapse = " vs ")
summary(glht(m1, linfct = K))
})## $puro
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## benlate vs captam == 0 -0.36472 0.07013 -5.200 <1e-04 ***
## benlate vs derosal == 0 0.02717 0.07013 0.387 0.921
## captam vs derosal == 0 0.39189 0.07013 5.588 <1e-04 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)
##
##
## $antes
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## benlate vs captam == 0 -0.06424 0.07013 -0.916 0.633
## benlate vs derosal == 0 0.03176 0.07013 0.453 0.893
## captam vs derosal == 0 0.09600 0.07013 1.369 0.366
## (Adjusted p values reported -- single-step method)
##
##
## $misturado
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## benlate vs captam == 0 -0.20239 0.07013 -2.886 0.0164 *
## benlate vs derosal == 0 0.17286 0.07013 2.465 0.0457 *
## captam vs derosal == 0 0.37524 0.07013 5.350 <0.001 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)Baseado nas comparações múltiplas feitas, em termos práticos, não existem diferenças entre os fungicidas quando são aplicados antes do polímero (será que o polímero aplicado depois dimunui a concentração do fungicida?). Para o fugicida Derosal e Benlate, não existe diferença entre as formas de aplicação. Para o Captam, existe diferença das outras formas de aplicação comparadas com a aplicação do fungicida antes do polímero.
Com o propósito didático será feita a comparação entre os 9 pontos experimentais. Na prática, essa abordagem pode indicar qual é tratamento mais promissor para o controle de fusarium. Será usando o método FDR (false discovery rate) para a correção dos p-valores para a multicplicidade de hipóteses. O métodos default, single-step, se torna demorado quando o número de hipótese é grande. Isso tem relação com uma integral calculada em alta dimensão.
# Comparação entre todos os pontos experimentais.
i <- !with(pts, fung %in% tail(levels(fung), n = 2))
L <- X_un[i, ]
rownames(L) <- with(pts[i, ], sprintf("%s:%s", fung, aplic))
K <- all_pairwise(L, collapse = " vs ")
# Número de constrastes avaliados.
c(choose(nrow(L), 2), nrow(K))## [1] 36 36summary(glht(m1, linfct = K), test = adjusted(type = "fdr"))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X_ok, data = tb)
##
## Linear Hypotheses:
## Estimate Std. Error
## benlate:puro vs benlate:antes == 0 0.008761 0.070134
## benlate:puro vs benlate:misturado == 0 -0.100584 0.070134
## benlate:puro vs captam:puro == 0 -0.364723 0.070134
## benlate:puro vs captam:antes == 0 -0.055481 0.070134
## benlate:puro vs captam:misturado == 0 -0.302970 0.070134
## benlate:puro vs derosal:puro == 0 0.027171 0.070134
## benlate:puro vs derosal:antes == 0 0.040517 0.070134
## benlate:puro vs derosal:misturado == 0 0.072273 0.070134
## benlate:antes vs benlate:misturado == 0 -0.109345 0.070134
## benlate:antes vs captam:puro == 0 -0.373485 0.070134
## benlate:antes vs captam:antes == 0 -0.064242 0.070134
## benlate:antes vs captam:misturado == 0 -0.311731 0.070134
## benlate:antes vs derosal:puro == 0 0.018409 0.070134
## benlate:antes vs derosal:antes == 0 0.031756 0.070134
## benlate:antes vs derosal:misturado == 0 0.063512 0.070134
## benlate:misturado vs captam:puro == 0 -0.264140 0.070134
## benlate:misturado vs captam:antes == 0 0.045103 0.070134
## benlate:misturado vs captam:misturado == 0 -0.202386 0.070134
## benlate:misturado vs derosal:puro == 0 0.127755 0.070134
## benlate:misturado vs derosal:antes == 0 0.141101 0.070134
## benlate:misturado vs derosal:misturado == 0 0.172857 0.070134
## captam:puro vs captam:antes == 0 0.309242 0.070134
## captam:puro vs captam:misturado == 0 0.061754 0.070134
## captam:puro vs derosal:puro == 0 0.391894 0.070134
## captam:puro vs derosal:antes == 0 0.405241 0.070134
## captam:puro vs derosal:misturado == 0 0.436997 0.070134
## captam:antes vs captam:misturado == 0 -0.247489 0.070134
## captam:antes vs derosal:puro == 0 0.082652 0.070134
## captam:antes vs derosal:antes == 0 0.095998 0.070134
## captam:antes vs derosal:misturado == 0 0.127755 0.070134
## captam:misturado vs derosal:puro == 0 0.330141 0.070134
## captam:misturado vs derosal:antes == 0 0.343487 0.070134
## captam:misturado vs derosal:misturado == 0 0.375243 0.070134
## derosal:puro vs derosal:antes == 0 0.013347 0.070134
## derosal:puro vs derosal:misturado == 0 0.045103 0.070134
## derosal:antes vs derosal:misturado == 0 0.031756 0.070134
## t value Pr(>|t|)
## benlate:puro vs benlate:antes == 0 0.125 0.901153
## benlate:puro vs benlate:misturado == 0 -1.434 0.285465
## benlate:puro vs captam:puro == 0 -5.200 2.98e-05 ***
## benlate:puro vs captam:antes == 0 -0.791 0.577523
## benlate:puro vs captam:misturado == 0 -4.320 0.000287 ***
## benlate:puro vs derosal:puro == 0 0.387 0.763983
## benlate:puro vs derosal:antes == 0 0.578 0.679680
## benlate:puro vs derosal:misturado == 0 1.031 0.482716
## benlate:antes vs benlate:misturado == 0 -1.559 0.238998
## benlate:antes vs captam:puro == 0 -5.325 2.35e-05 ***
## benlate:antes vs captam:antes == 0 -0.916 0.530814
## benlate:antes vs captam:misturado == 0 -4.445 0.000235 ***
## benlate:antes vs derosal:puro == 0 0.262 0.840884
## benlate:antes vs derosal:antes == 0 0.453 0.734538
## benlate:antes vs derosal:misturado == 0 0.906 0.530814
## benlate:misturado vs captam:puro == 0 -3.766 0.001465 **
## benlate:misturado vs captam:antes == 0 0.643 0.649861
## benlate:misturado vs captam:misturado == 0 -2.886 0.015503 *
## benlate:misturado vs derosal:puro == 0 1.822 0.150640
## benlate:misturado vs derosal:antes == 0 2.012 0.113354
## benlate:misturado vs derosal:misturado == 0 2.465 0.042453 *
## captam:puro vs captam:antes == 0 4.409 0.000237 ***
## captam:puro vs captam:misturado == 0 0.881 0.530814
## captam:puro vs derosal:puro == 0 5.588 1.63e-05 ***
## captam:puro vs derosal:antes == 0 5.778 1.29e-05 ***
## captam:puro vs derosal:misturado == 0 6.231 5.56e-06 ***
## captam:antes vs captam:misturado == 0 -3.529 0.002745 **
## captam:antes vs derosal:puro == 0 1.178 0.400803
## captam:antes vs derosal:antes == 0 1.369 0.305163
## captam:antes vs derosal:misturado == 0 1.822 0.150640
## captam:misturado vs derosal:puro == 0 4.707 0.000113 ***
## captam:misturado vs derosal:antes == 0 4.898 6.95e-05 ***
## captam:misturado vs derosal:misturado == 0 5.350 2.35e-05 ***
## derosal:puro vs derosal:antes == 0 0.190 0.874231
## derosal:puro vs derosal:misturado == 0 0.643 0.649861
## derosal:antes vs derosal:misturado == 0 0.453 0.734538
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- fdr method)As médias para cada ponto experimental serão determinadas agora, acompanhadas do respectivo intervalo de confiança para que os resultados sejam apresentados de forma gráfica.
# Obtém os intervalos de confiaça para as médias ajustadas.
ci <- confint(glht(m1, linfct = X_un),
calpha = univariate_calpha())
ci <- ci$confint
colnames(ci) <- tolower(colnames(ci))
# Estimativas pontuais com intervalo de confiança de cobertura
# individual de 95%.
results <- cbind(pts, ci)
results## fung aplic estimate lwr upr
## 1 benlate puro 7.227342e-02 -0.02767258 0.17221943
## 2 benlate antes 6.351209e-02 -0.03643392 0.16345809
## 3 benlate misturado 1.728572e-01 0.07291120 0.27280321
## 4 captam puro 4.369968e-01 0.33705076 0.53694277
## 5 captam antes 1.277545e-01 0.02780852 0.22770053
## 6 captam misturado 3.752432e-01 0.27529718 0.47518919
## 7 derosal puro 4.510268e-02 -0.05484332 0.14504869
## 8 derosal antes 3.175604e-02 -0.06818996 0.13170205
## 9 derosal misturado 3.469447e-17 -0.09994601 0.09994601
## 10 polimero polimero 4.473808e-01 0.34743480 0.54732681
## 11 testemunha testemunha 4.899207e-01 0.38997471 0.58986672A função apmc do pacote wzRfun é uma função de conveniência que faz chamada à glht(), retorna o intervalo de confiança e as letras para indicar a significância dos contrastes. Para usar em modelos em que a matriz de desenho é fornecida ao contrário do convencional uso de fórmula, precisa adicionar cld2 = TRUE.
library(wzRfun)## ----------------------------------------------------------------------
## wzRfun: Walmes Zeviani's collection of functions
##
## wzRfun (0.91, built on 2019-07-11) was loaded.
## Consult online documentation at:
## http://leg.ufpr.br/~walmes/pacotes/wzRfun
## For collaboration, support or bug report, visit:
## https://github.com/walmes/wzRfun/issues
## For general information: packageDescription("wzRfun")
## To access the package documentation: help(package = "wzRfun")
## ----------------------------------------------------------------------# Para comparação entre TODOS os pontos experimentais.
L <- X_un
rownames(L) <- with(pts, sprintf("%s:%s", fung, aplic))
# Estimativas pontuais seguido de IC e letras para os contrastes.
results <- apmc(X = L,
model = m1,
focus = "trat",
test = "fdr",
cld2 = TRUE) %>%
separate(col = "trat", into = c("fung", "aplic")) %>%
mutate(cld = ordered_cld(cld, -fit)) %>%
mutate(txt = sprintf("%0.2f%s", fit, cld))
results## fung aplic fit lwr upr cld
## 1 benlate puro 7.227342e-02 -0.02767258 0.17221943 ab
## 2 benlate antes 6.351209e-02 -0.03643392 0.16345809 ab
## 3 benlate misturado 1.728572e-01 0.07291120 0.27280321 b
## 4 captam puro 4.369968e-01 0.33705076 0.53694277 c
## 5 captam antes 1.277545e-01 0.02780852 0.22770053 ab
## 6 captam misturado 3.752432e-01 0.27529718 0.47518919 c
## 7 derosal puro 4.510268e-02 -0.05484332 0.14504869 ab
## 8 derosal antes 3.175604e-02 -0.06818996 0.13170205 ab
## 9 derosal misturado 3.469447e-17 -0.09994601 0.09994601 a
## 10 polimero polimero 4.473808e-01 0.34743480 0.54732681 c
## 11 testemunha testemunha 4.899207e-01 0.38997471 0.58986672 c
## txt
## 1 0.07ab
## 2 0.06ab
## 3 0.17b
## 4 0.44c
## 5 0.13ab
## 6 0.38c
## 7 0.05ab
## 8 0.03ab
## 9 0.00a
## 10 0.45c
## 11 0.49c# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = aplic, y = fit)) +
facet_grid(facets = . ~ fung, scale = "free", space = "free") +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = txt),
angle = 90,
vjust = 0,
nudge_x = -0.1)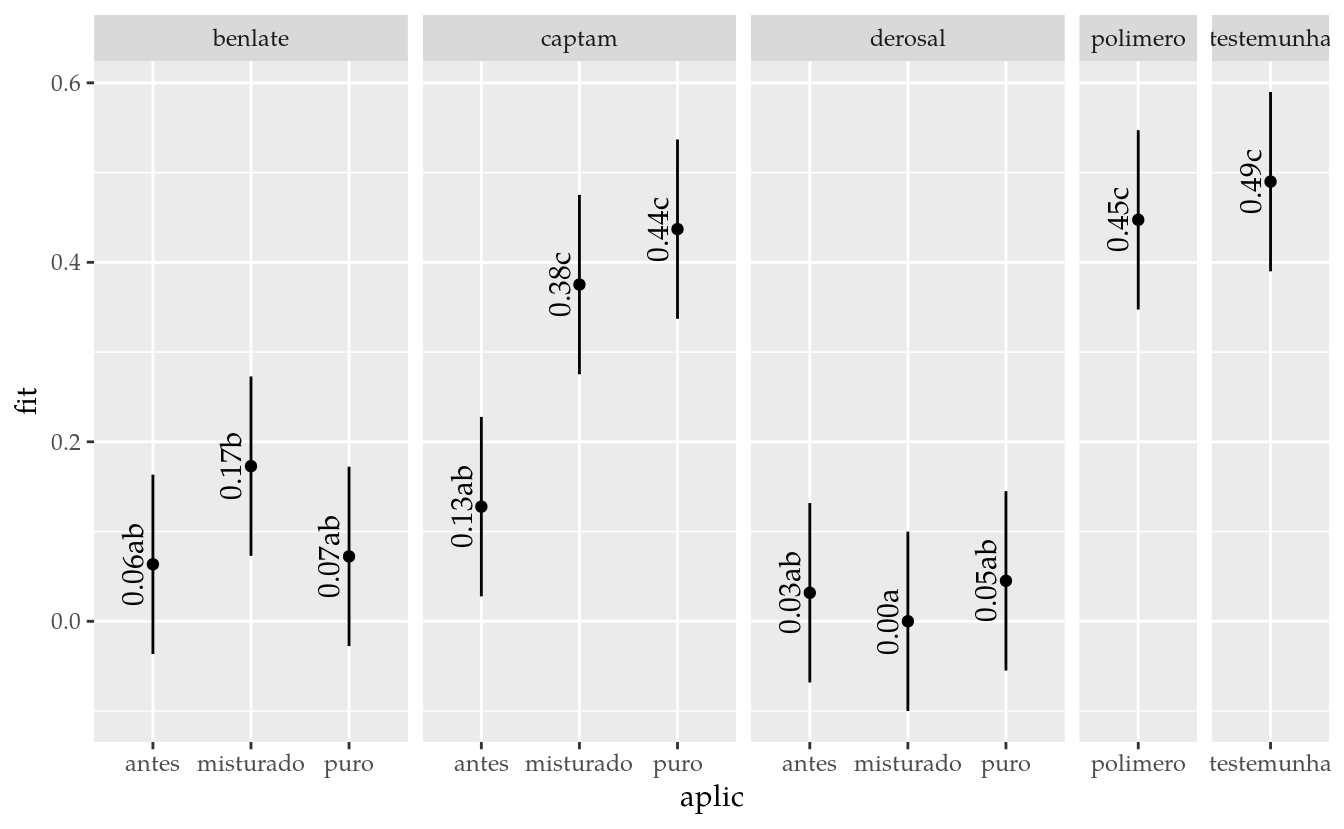
Agora que a análise chegou ao final, pode-se fazer alguns comentários importantes que justificam a adoção do modelo binomial. A mais relevante é que o limite inferior do intervalo de confiança de algumas estimativas de média é negativo. Isso é algo que não tem significado prático.
A função \(f(x) = \arcsin(\sqrt{x})\) tem limites \[ \begin{aligned} \lim_{x \to 0} \arcsin(\sqrt{x}) &= 0 \\ \lim_{x \to 1} \arcsin(\sqrt{x}) &= \pi/2 \end{aligned} \] e a \(f^{-1}(y)\) é \(\sin^{2}(y)\), \(y \in [0, \pi/2]\).
curve(asin(sqrt(x)), from = 0, to = 1, asp = 1, xlim = c(0, pi/2))
curve(sin(x)^2, from = 0, to = pi/2, add = TRUE, col = 2)
abline(a = 0, b = 1, lty = 2)
legend("bottomright", col = c(1, 2), lty = 1, bty = "n",
legend = c(expression(asin(sqrt(x))),
expression(sin^2 * (x))))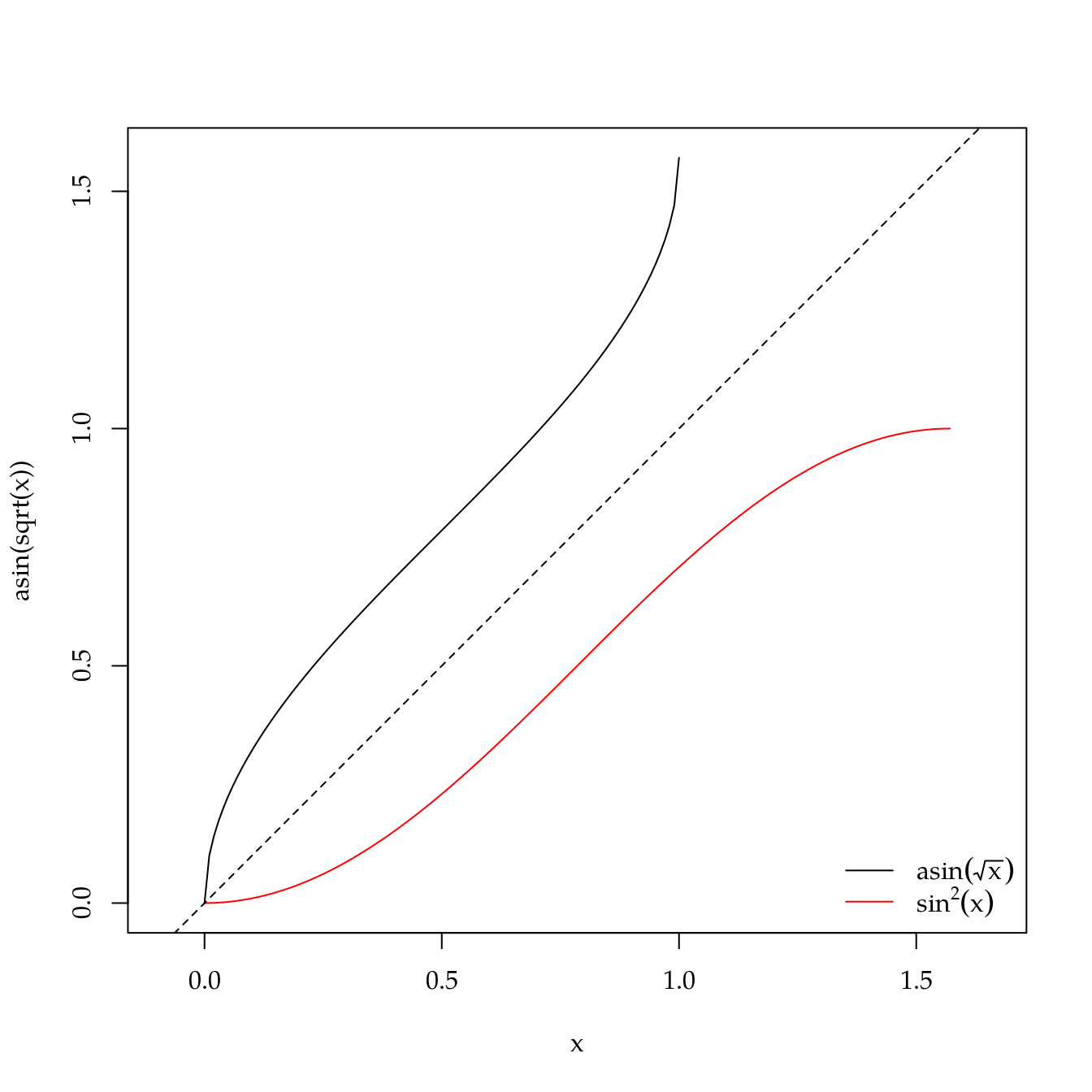
Podemos aplicar a tranformação inversa para interpretar as estimativas na escala original de proporção de sementes infectadas. Mas como os valores limites inferiores são negativos, ao fazer-se \(\sin^2(x)\), eles se tornarão positivos e podem, inclusive deixar a estimativa pontual fora do intervalo de confiança, o que seria bastante estranho.
# Apenas para provar o ponto mencionado.
results %>%
mutate_at(c("fit", "lwr", "upr"), function(x) sin(x)^2) %>%
ggplot(mapping = aes(x = aplic, y = fit)) +
facet_grid(facets = . ~ fung, scale = "free", space = "free") +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0)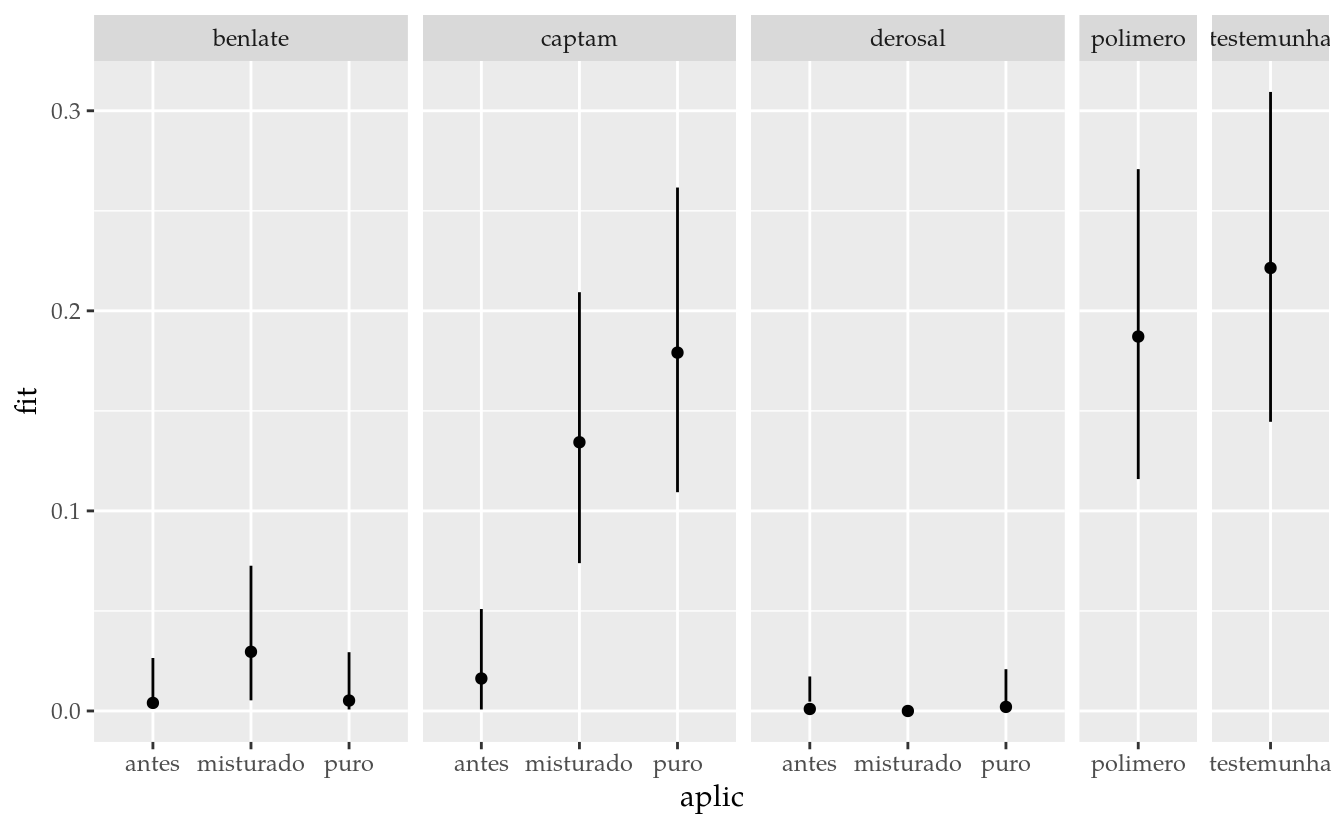
A seguir faremos a análise baseada na distribuição binomial.
10.3 Análise com modelo binomial
Por se tratar de um manual de análise, não serão feitas apresentação de todos dos aspectos teóricos relacionados à classe de modelos lineares generalizados. Recomenda-se que o interessado procure a literatura especializada para isso. Surigo começar pelo material do Prof. Gauss & Profa. Clarice3 e o materiial do Prof. Gilberto de Paula4.
O que tem-se que comentar já foi brevemente mencionado: estimativa de proporção amostral igual a 0. Como os fatores experimentais são todos qualitativos, o modelo para estimar a proporção é baseado em estatísticas amostrais dos pontos experimentais. No entanto, sabemos da distribuição binomial que o espaço paramétrico de \(p\) é o intervalo aberto \((0, 1)\). Ou seja, teremos problema ao ajustar o modelo aos dados por causa de um ponto experimental com estimativa 0. Além do mais, o erro padrão da estimativa assume um valor muito grande pois quando \(p\) tende às bordas do espaço paramétrico, o peso \(w = p(1 - p)\) tende a 0 e, portanto, o recíproco, \(w^{-1}\), tende ao infinito. Isso inflaciona o erro padrão dos efeitos. Chega a ser paradoxal: os dados observados tem variância amostral igual a 0 mas o erro padrão da estimativa é enorme. Confira os resultados no código abaixo.
TODO: o problema está no erro padrão da estimativa.
# Dados simulados para mostrar o problema da proporção amostral 0 em um
# ponto experimental.
y <- rep(0, 10)
m0 <- glm(cbind(y, 40 - y) ~ 1, family = binomial)
summary(m0)##
## Call:
## glm(formula = cbind(y, 40 - y) ~ 1, family = binomial)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -6.048e-06 -6.048e-06 -6.048e-06 -6.048e-06 -6.048e-06
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -28.41 44855.74 -0.001 0.999
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 0.0000e+00 on 9 degrees of freedom
## Residual deviance: 3.6575e-10 on 9 degrees of freedom
## AIC: 2
##
## Number of Fisher Scoring iterations: 24# Quantidades envolvidas no cálculo do erro padrão.
X <- model.matrix(m0)
p <- fitted(m0)
W <- diag(p * (1 - p))
# Erro padrão. NOTE: differença no valor é under/overflow.
sqrt(solve(t(X) %*% W %*% X))## (Intercept)
## (Intercept) 467730sqrt(vcov(m0))## (Intercept)
## (Intercept) 44855.74Dando continuidade, os dados do experimento de fusarium tem um ponto experimental com proporção amostral igual a 0. Já foi discutido que, no modelo de estimativas por ponto experimental como o que será usado, isso é um problema. Dessa forma, a situação será contornada de forma simples. Arbitrariamente iremos somar uma constante positiva pequena ao vetor de resposta. Isso irá induzir um vício, porém não fazê-lo também é comprometedor porque os erros padrões dos efeitos será enorme. Entendo que essa abordagem está sujeita a questionamento e que existem várias abordagens melhores. Mas para o momento é assim que iremos seguir.
Como não temos garantia de que as suposições da binomial serão atendidas, vamos usar a distribuição quasibinomial. Para ser exato, quasibinomial não é uma distribuição de probabilidades. É uma especificação de primeiro e segundo momentos que tem as mesmas equações de média e variância da distribuição binomial. A diferença é que um parâmetro de dispersão é estimado e isso irá acomodar a existência de variância extra binomial. A variância entra binomial é a proveniente das repetições de um mesmo ponto experimental apresentarem proporção (teórica) de sucesso não igual em função do tamanho/idade/fisiologia das sementes.
# Proporção amostral de sementes infectadas em cada ponto experimental.
with(tb, tapply(nsi/40, list(fung, aplic), FUN = mean))## puro antes misturado polimero testemunha
## benlate 0.025 0.010 0.050 NA NA
## captam 0.185 0.040 0.135 NA NA
## derosal 0.010 0.005 0.000 NA NA
## polimero NA NA NA 0.19 NA
## testemunha NA NA NA NA 0.225# Somar 0.2 aos dados observados. Se for binomial, tem-se que somar um
# inteiro. Quasibinomial permite valores decimais.
tb$nsim <- tb$nsi + 1/5
# Ajuste com a variável modificada.
m0 <- glm(cbind(nsim, 40 - nsim) ~ fung * aplic,
data = tb,
family = quasibinomial)
anova(m0, test = "F")## Analysis of Deviance Table
##
## Model: quasibinomial, link: logit
##
## Response: cbind(nsim, 40 - nsim)
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev F Pr(>F)
## NULL 54 256.052
## fung 4 167.058 50 88.994 27.9972 1.359e-11 ***
## aplic 2 22.577 48 66.417 7.5673 0.001498 **
## fung:aplic 4 5.852 44 60.565 0.9807 0.427865
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Usando a matriz.
m1 <- update(m0, . ~ 0 + X_ok)
summary(m1)##
## Call:
## glm(formula = cbind(nsim, 40 - nsim) ~ X_ok - 1, family = quasibinomial,
## data = tb)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -1.7734 -0.7180 -0.3518 0.3204 2.7708
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## X_ok(Intercept) -2.58509 0.14534 -17.787 < 2e-16 ***
## X_okfung1 0.69737 0.18171 3.838 0.000393 ***
## X_okfung2 -0.62895 0.19419 -3.239 0.002287 **
## X_okfung3 0.42400 0.05334 7.949 4.77e-10 ***
## X_okfung4 0.10477 0.14969 0.700 0.487689
## X_okaplic1 -0.45401 0.24952 -1.820 0.075638 .
## X_okaplic2 0.05781 0.16722 0.346 0.731212
## X_okfung1:aplic1 -0.22414 0.24788 -0.904 0.370815
## X_okfung2:aplic1 0.12437 0.20440 0.608 0.545993
## X_okfung1:aplic2 -0.09154 0.11196 -0.818 0.417962
## X_okfung2:aplic2 -0.17948 0.15422 -1.164 0.250792
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for quasibinomial family taken to be 1.491737)
##
## Null deviance: 2031.074 on 55 degrees of freedom
## Residual deviance: 60.565 on 44 degrees of freedom
## AIC: NA
##
## Number of Fisher Scoring iterations: 5# Diagnóstico não será discutivo para não afastar do foco.
# par(mfrow = c(2, 2))
# plot(m0)
# layout(1)
# Modelo declarado com a matriz de delineamento dividida.
m2 <- update(m0, . ~ X_ok[, ef[[1]], drop = FALSE] +
X_ok[, ef[[2]], drop = FALSE] +
X_ok[, ef[[3]], drop = FALSE] +
X_ok[, ef[[4]], drop = FALSE] +
X_ok[, ef[[5]], drop = FALSE])
cbind(m0 = coef(m0)[sub("X_ok", "", names(coef(m1)))],
m1 = coef(m1),
m2 = coef(m2))## m0 m1 m2
## (Intercept) -2.5850913 -2.5850913 -2.5850913
## fung1 0.6973655 0.6973655 0.4240048
## fung2 -0.6289523 -0.6289523 0.1047658
## fung3 0.4240048 0.4240048 0.6973655
## fung4 0.1047658 0.1047658 -0.6289523
## aplic1 -0.4540100 -0.4540100 -0.4540100
## aplic2 0.0578093 0.0578093 0.0578093
## fung1:aplic1 -0.2241365 -0.2241365 -0.2241365
## fung2:aplic1 0.1243729 0.1243729 0.1243729
## fung1:aplic2 -0.0915423 -0.0915423 -0.0915423
## fung2:aplic2 -0.1794795 -0.1794795 -0.1794795# Teste F sequencial para o termos do modelo.
anova(m2, test = "F")## Analysis of Deviance Table
##
## Model: quasibinomial, link: logit
##
## Response: cbind(nsim, 40 - nsim)
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev
## NULL 54 256.052
## X_ok[, ef[[1]], drop = FALSE] 1 82.956 53 173.096
## X_ok[, ef[[2]], drop = FALSE] 1 0.733 52 172.363
## X_ok[, ef[[3]], drop = FALSE] 2 83.369 50 88.994
## X_ok[, ef[[4]], drop = FALSE] 2 22.577 48 66.417
## X_ok[, ef[[5]], drop = FALSE] 4 5.852 44 60.565
## F Pr(>F)
## NULL
## X_ok[, ef[[1]], drop = FALSE] 55.6103 2.457e-09 ***
## X_ok[, ef[[2]], drop = FALSE] 0.4912 0.487097
## X_ok[, ef[[3]], drop = FALSE] 27.9436 1.468e-08 ***
## X_ok[, ef[[4]], drop = FALSE] 7.5673 0.001498 **
## X_ok[, ef[[5]], drop = FALSE] 0.9807 0.427865
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Teste F marginal para o termos do modelo.
drop1(m2, test = "F")## Single term deletions
##
## Model:
## cbind(nsim, 40 - nsim) ~ X_ok[, ef[[1]], drop = FALSE] + X_ok[,
## ef[[2]], drop = FALSE] + X_ok[, ef[[3]], drop = FALSE] +
## X_ok[, ef[[4]], drop = FALSE] + X_ok[, ef[[5]], drop = FALSE]
## Df Deviance F value Pr(>F)
## <none> 60.565
## X_ok[, ef[[1]], drop = FALSE] 1 179.146 86.1478 6.324e-12 ***
## X_ok[, ef[[2]], drop = FALSE] 1 61.298 0.5323 0.4695
## X_ok[, ef[[3]], drop = FALSE] 2 117.488 20.6768 4.667e-07 ***
## X_ok[, ef[[4]], drop = FALSE] 2 65.830 1.9123 0.1598
## X_ok[, ef[[5]], drop = FALSE] 4 66.417 1.0628 0.3862
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Para extrair as estatística do teste produzidas pela cftest().
extract_cftest <- function(x) {
as.data.frame(rbind(unlist(x$test[c("df", "fstat", "pvalue")])))
}
# Para extrair as estatística do teste produzidas pela linearHypothesis().
extract_linearHyphotesis <- function(x) {
x[2, c(2, 1, 3, 4)]
}
# Teste de Wald para hipóteses globais.
w <- lapply(ef, FUN = cftest, model = m1, test = Ftest())
w <- lapply(w, FUN = extract_cftest)
do.call(rbind, w)## df1 df2 fstat pvalue
## ii) T+P vs Fat 1 44 63.1923681 4.769772e-10
## i) T vs P 1 44 0.4898256 4.876892e-01
## Fung 2 44 15.0328478 1.057749e-05
## Aplic 2 44 1.6599004 2.018436e-01
## Fung:Aplic 4 44 0.9411846 4.491539e-01# Matriz de hipóteses lineares (linear hypothesis matrix).
m <- diag(length(coef(m1)))
lhm <- ef
for (i in 1:length(ef)) {
lhm[[i]] <- m[ef[[i]], , drop = FALSE]
}
lhm## $`ii) T+P vs Fat`
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
## [1,] 0 0 0 1 0 0 0 0 0 0 0
##
## $`i) T vs P`
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
## [1,] 0 0 0 0 1 0 0 0 0 0 0
##
## $Fung
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
## [1,] 0 1 0 0 0 0 0 0 0 0 0
## [2,] 0 0 1 0 0 0 0 0 0 0 0
##
## $Aplic
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
## [1,] 0 0 0 0 0 1 0 0 0 0 0
## [2,] 0 0 0 0 0 0 1 0 0 0 0
##
## $`Fung:Aplic`
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
## [1,] 0 0 0 0 0 0 0 1 0 0 0
## [2,] 0 0 0 0 0 0 0 0 1 0 0
## [3,] 0 0 0 0 0 0 0 0 0 1 0
## [4,] 0 0 0 0 0 0 0 0 0 0 1# Teste de Wald para hipóteses globais.
w <- lapply(lhm, FUN = linearHypothesis, model = m1, test = "F")
w <- lapply(w, FUN = extract_linearHyphotesis)
do.call(rbind, w)## Df Res.Df F Pr(>F)
## ii) T+P vs Fat 1 44 63.1924 4.770e-10 ***
## i) T vs P 1 44 0.4898 0.4877
## Fung 2 44 15.0328 1.058e-05 ***
## Aplic 2 44 1.6599 0.2018
## Fung:Aplic 4 44 0.9412 0.4492
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Várias resultados foram obtidos usando diferentes abordagens. Para começar, não existe diferença em resultado entre multcomp::cftest() e car::linearHypothesis(). Ambos aplicam o teste de Wald com estatística F e testam hipóteses marginais, ou seja, testam a hipótese assumindo que todos os demais parâmetros estão presentes no modelo. No código abaixo reproduzimos com operações matriciais a estatística do teste de Wald para a hipótese de interação nula entre os fatores. Vale lembrar que as operações matriciais feitas dessa forma são interessantes para fins didáticos mas não do ponto de vista computacional. Portanto, não implemente funções dessa maneira.
L <- lhm[[5]] # Matriz que especifica uma hipótese linear.
V <- vcov(m1) # Matriz de covariância das estimativas.
b <- coef(m1) # Estimativas.
nrow(L) # Número de hipóteses em L.## [1] 4L %*% b # Estimativa da função linear L.## [,1]
## [1,] -0.2241365
## [2,] 0.1243729
## [3,] -0.0915423
## [4,] -0.1794795L %*% V %*% t(L) # Variâncias.## [,1] [,2] [,3] [,4]
## [1,] 0.0614468181 0.0112313158 -0.0077834133 -0.0008570548
## [2,] 0.0112313158 0.0417779783 -0.0008570548 -0.0031665331
## [3,] -0.0077834133 -0.0008570548 0.0125344930 0.0020030725
## [4,] -0.0008570548 -0.0031665331 0.0020030725 0.0237848298# Estatística F do teste de Wald.
Vinv <- solve(L %*% V %*% t(L))
Lest <- L %*% b
t(Lest) %*% Vinv %*% (Lest)/nrow(L)## [,1]
## [1,] 0.9411846O método anova(), por outro lado, obtém uma estatística F por meio da diferença de deviances e as hipóteses são sequenciais. O método drop1() obtém estatísticas para hipóteses marginais. Para o praticamente, o importante é saber que o nível descritivo (estatística de teste e p-valor) será diferente e que os métodos baseados em diviance devem ser preferidos aos métodos de Wald.
A função multcomp::glht() será usada para obter comparações múltiplas entre todos os pontos experimentais. Isso será feito para poder comparar com o resultado usando o modelo gaussiano na variável transformada da seção anterior. Uma vez que não houve interação, o recomendado é apenas estudar os efeitos principais.
# Para comparação entre TODOS os pontos experimentais.
L <- X_un
rownames(L) <- with(pts, sprintf("%s:%s", fung, aplic))
# Estimativas pontuais seguido de IC e letras para os contrastes.
results <- apmc(X = L,
model = m1,
focus = "trat",
test = "fdr",
cld2 = TRUE) %>%
separate(col = "trat", into = c("fung", "aplic")) %>%
mutate_at(c("fit", "lwr", "upr"), m1$family$linkinv) %>%
mutate(cld = ordered_cld(cld, -fit)) %>%
mutate(txt = sprintf("%0.2f%s", fit, cld))
results## fung aplic fit lwr upr cld txt
## 1 benlate puro 0.030 0.0113360411 0.07699956 a 0.03a
## 2 benlate antes 0.015 0.0037690485 0.05775658 a 0.02a
## 3 benlate misturado 0.055 0.0269532823 0.10896311 a 0.06a
## 4 captam puro 0.190 0.1322178849 0.26531366 b 0.19b
## 5 captam antes 0.045 0.0204006543 0.09634423 a 0.05a
## 6 captam misturado 0.140 0.0908651271 0.20957936 b 0.14b
## 7 derosal puro 0.015 0.0037690502 0.05775656 a 0.02a
## 8 derosal antes 0.010 0.0018396400 0.05245615 a 0.01a
## 9 derosal misturado 0.005 0.0004557433 0.05247659 a 0.01a
## 10 polimero polimero 0.195 0.1364521915 0.27079151 b 0.20b
## 11 testemunha testemunha 0.230 0.1665140637 0.30872531 b 0.23b# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = aplic, y = fit)) +
facet_grid(facets = . ~ fung, scale = "free", space = "free") +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = txt),
angle = 90,
vjust = 0,
nudge_x = -0.1)
Agora serão estudos os efeitos principais. O procedimento é muito similar. No entanto, tem-se que tomar cuidado com um aspecto muitas vezes negligenciado ou não discutido. Para modelos com função de ligação diferente da identidadade (em geral as funções de ligação são não lineares), obter médias ajustadas para os efeitos principais pela marginalização dos efeitos de interação é diferente de obter as médias ajustadas para os efeitos principais com um modelo em que a interação não está presente. A diferença entre essas duas abordagens pode ser entendida com os códigos a seguir.
# Pontos experimentais únicos de A * B.
i <- tb %>% select(fung, aplic) %>% duplicated()
pts <- tb[!i, c("fung", "aplic")] # Pontos experimentais.
# Modelos em que os termos de interação são abandonados.
m3 <- update(m1, . ~ 0 + X_ok[, 1:7, drop = FALSE])
# Matrizes de delineamento por ponto experimental de cada modelo.
X_un1 <- model.matrix(m1)[!i, ] # Contém fung:aplic.
X_un3 <- model.matrix(m3)[!i, ] # Abandonou fung:aplic.
cbind(pts, "|" = "|", unname(X_un1))## fung aplic | 1 2 3 4 5 6 7 8 9 10 11
## 1 benlate puro | 1 -1 -1 -2 0 -1 -1 1 1 1 1
## 2 benlate antes | 1 -1 -1 -2 0 1 -1 -1 -1 1 1
## 3 benlate misturado | 1 -1 -1 -2 0 0 2 0 0 -2 -2
## 4 captam puro | 1 1 -1 -2 0 -1 -1 -1 1 -1 1
## 5 captam antes | 1 1 -1 -2 0 1 -1 1 -1 -1 1
## 6 captam misturado | 1 1 -1 -2 0 0 2 0 0 2 -2
## 7 derosal puro | 1 0 2 -2 0 -1 -1 0 -2 0 -2
## 8 derosal antes | 1 0 2 -2 0 1 -1 0 2 0 -2
## 9 derosal misturado | 1 0 2 -2 0 0 2 0 0 0 4
## 10 polimero polimero | 1 0 0 3 -1 0 0 0 0 0 0
## 11 testemunha testemunha | 1 0 0 3 1 0 0 0 0 0 0cbind(pts, "|" = "|", unname(X_un3))## fung aplic | 1 2 3 4 5 6 7
## 1 benlate puro | 1 -1 -1 -2 0 -1 -1
## 2 benlate antes | 1 -1 -1 -2 0 1 -1
## 3 benlate misturado | 1 -1 -1 -2 0 0 2
## 4 captam puro | 1 1 -1 -2 0 -1 -1
## 5 captam antes | 1 1 -1 -2 0 1 -1
## 6 captam misturado | 1 1 -1 -2 0 0 2
## 7 derosal puro | 1 0 2 -2 0 -1 -1
## 8 derosal antes | 1 0 2 -2 0 1 -1
## 9 derosal misturado | 1 0 2 -2 0 0 2
## 10 polimero polimero | 1 0 0 3 -1 0 0
## 11 testemunha testemunha | 1 0 0 3 1 0 0O termo de interação dupla não está presente no modelo m3, então m3 é um modelo de efeitos aditivos nos fatores fungicida e aplicação. O modelo m1 contém o termo de interação. Dizemos que m3 é um modelo encaixado em m1, obritido pelo abandono de termos do modelo m1. As matrizes de delineamento por ponto experimental diferem justamente em relação às colunas do termo de interação.
compute_lsmeans <- function(X, model, focus) {
apmc(X = X, model = model, focus = focus,
test = "single-step", cld2 = TRUE) %>%
mutate_at(c("fit", "lwr", "upr"),
model$family$linkinv)
}
pd <- position_dodge(0.2)
fit_F <- list()
fit_A <- list()
# Contrastes dois a dois entre níveis de fungicida.
L_F <- do.call(rbind, by(X_un1, pts$fung, colMeans)) %>% head(n = -2)
fit_F$m1 <- compute_lsmeans(X = L_F, model = m1, focus = "fung")
# Contrastes dois a dois entre níveis de fungicida.
L_F <- do.call(rbind, by(X_un3, pts$fung, colMeans)) %>% head(n = -2)
fit_F$m3 <- compute_lsmeans(X = L_F, model = m3, focus = "fung")
fit_F## $m1
## fung fit lwr upr cld
## 1 benlate 0.029269185 0.015939271 0.05314478 b
## 2 captam 0.108439296 0.079078653 0.14696146 a
## 3 derosal 0.009093995 0.003091884 0.02643868 b
##
## $m3
## fung fit lwr upr cld
## 1 benlate 0.029179251 0.016817156 0.05016492 b
## 2 captam 0.112725404 0.084145723 0.14942827 a
## 3 derosal 0.008686109 0.003236612 0.02309832 bfit_F <- bind_rows(fit_F, .id = "modelo")
L_A <- do.call(rbind, by(X_un1, pts$aplic, colMeans)) %>% head(n = -2)
fit_A$m1 <- compute_lsmeans(X = L_A, model = m1, focus = "aplic")
L_A <- do.call(rbind, by(X_un3, pts$aplic, colMeans)) %>% head(n = -2)
fit_A$m3 <- compute_lsmeans(X = L_A, model = m3, focus = "aplic")
fit_A## $m1
## aplic fit lwr upr cld
## 1 puro 0.04578641 0.025963917 0.07950734 a
## 2 antes 0.01898534 0.008778222 0.04057525 a
## 3 misturado 0.03497629 0.015209994 0.07838546 a
##
## $m3
## aplic fit lwr upr cld
## 1 puro 0.05053171 0.032072707 0.07875018 b
## 2 antes 0.01401433 0.006856185 0.02843193 a
## 3 misturado 0.04235757 0.026175542 0.06784654 bfit_A <- bind_rows(fit_A, .id = "modelo")
ggplot(data = fit_F,
mapping = aes(x = fung, y = fit,
color = modelo, group = modelo)) +
geom_point(position = pd) +
geom_errorbar(aes(ymin = lwr, ymax = upr),
position = pd, width = 0.1)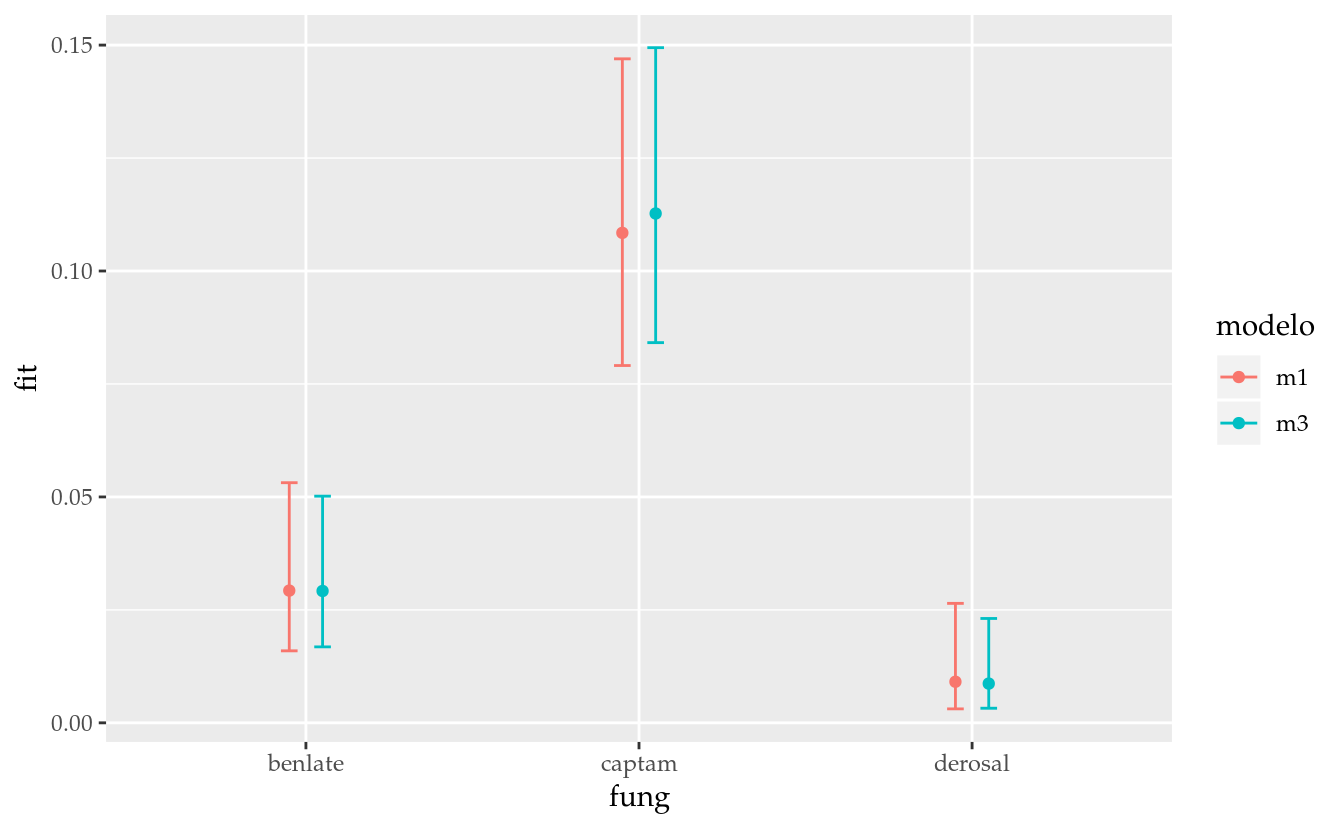
ggplot(data = fit_A,
mapping = aes(x = aplic, y = fit,
color = modelo, group = modelo)) +
geom_point(position = pd) +
geom_errorbar(aes(ymin = lwr, ymax = upr),
position = pd, width = 0.1)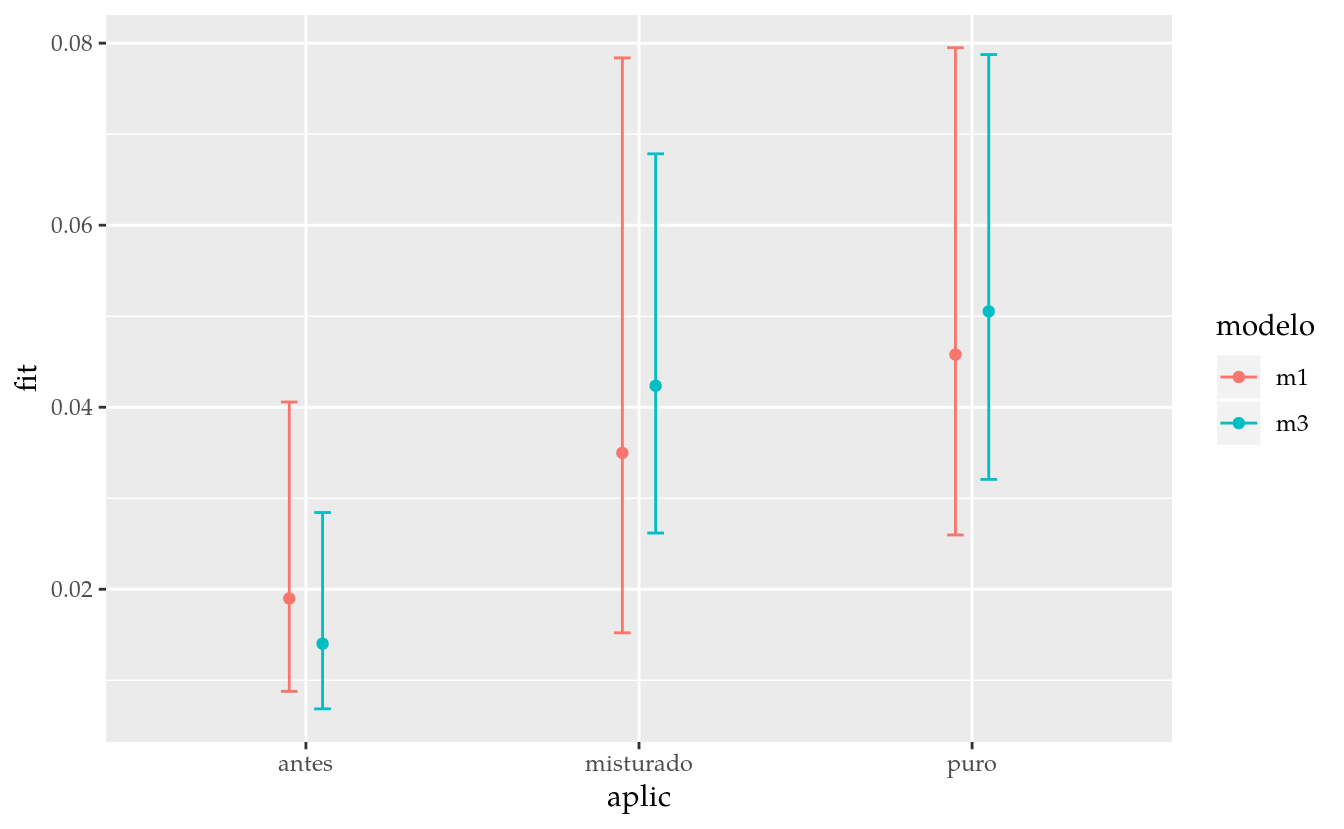
Os resultados parecem bem próximos, afinal, somos um pouco enganados pela escala. Por isso é importante comparar a diferença das estimativas considerando a incerteza associada a elas, que pode ser representada com intervalo de confiança. O gráfico deixa bastante evidente que a escolha do modelo pode resultar em conclusões diferentes. As estimativas pontuais são diferentes mas a diferença em amplitude dos intervalos de confiança é talvez o aspecto mais importante. No entanto, com um modelo que assume distribuição gaussiana de variância constante e dados balanceados, não seria visto diferença entre m1 e m3. Dessa forma, a abordagem mais segura é abandonar os termos que não são relevantes para o modelo. Existem excessões, como termos de blocagem, por exemplo. Eles serão discutidos em outro capítulo.
Se você está curioso ou não ainda convencido do que foi dito, experimente por você mesmo. Defina m1 e m3 como modelos com distribuição Gaussiana e repita os blocos anteriores. As estimativas pontuais são diferentes a partir da 3ª casa decimal, questão de representação numérica.
# Ajuste modelo gaussiano.
m1 <- glm(y ~ 0 + X_ok, data = tb, family = gaussian)
m3 <- update(m1, . ~ 0 + X_ok[, 1:7, drop = FALSE])
# Repita o código após a definição da função `compute_lsmeans()`.10.4 Óleos essenciais
10.5 Considerações finais
Aqui encerra-se o tutorial sobre a análise de experimento fatorial com dois tratamentos adicionais. Na primeira seção o procedimento foi demostrando com dados similados. Na próxima sessão o procedimento foi utilizado para a análise dos dados de fusarium em ZIMMERMANN (2004). Na última seção, a análise considerou modelo linear generalizado com família quasibinomial.
Vários aspectos computacionais e inferenciais foram destacados ao longo das análises. Em caso de dúvida, esclarecimento ou contribuição, contate o autor.
Referências Bibliográficas
ZIMMERMANN, F. J. Estatística aplicada à pesquisa agrícola. 1st ed. Santo Antônio de Goiás, GO: Embrapa Arroz e Feijão, 2004.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani