Capítulo 8 Fatorial duplo com um tratamento adicional
8.1 Motivação
Experimentos fatoriais são aqueles que estudam dois ou mais fatores de interesse simultâneamente. As celas experimentais, pontos experimentais ou tratamentos são as combinações dos níveis dos fatores. Os fatores podem ser combinados de forma (completamente) cruzada, de forma aninhada e de forma irregular.
O experimento fatorial com tratamento adicional é um caso particular de experimento fatorial. Ele tem uma estrutura fatorial, geralmente de cruzamento completo, mas a inclusão de pontos adicionais deixa a análise não trivial. Porém, com as providências corretas, é possível realizar a análise de forma comrpeensível e reproduzível.
Esse tipo de delineamento é bastante utilizado em experimentos agrícolas. O ponto adicional quase sempre corresponde a uma testemunha, ou seja, um tratamento placebo.
Existem experimentos em que dois tratamentos adicionais são usados. Há casos em que eles são chamados de testemunha positiva e negativa. A testemunha seria aquela na qual espera-se o pior resultado pois é um placebo. A testemunha positiva seria um tratamento padrão ouro, para o qual se antecipa bons resultados. Os dados em labestData::ZimmermannTb9.22 são de um experimento fatorial com dois tratamentos adicionais.
8.2 Construção com dados simulados
Serão usados dados simulados para celeridade. Assim pulamos coisas relacionadas a análise exploratória, avaliação dos pressupostos, interpretação, etc. Ou seja, os dados simulados serão apenas a matéria prima necessária para engenho funcionar. Após a construção do procedimento, faremos a aplicação em dados reais.
Para começarmos esse tutorial serão necessários os pacote car e multcomp. O primeiro contém a função glht() para realização de testes de hipóteses lineares gerais, úteis para aplicar procedimentos de comparação múltipla de hipóteses e contrastes planejados. Do segundo será usada a função linearHypothesis() que aplica o teste de Wald. O pacote tidyverse utilizaremos para operações tabulares.
# ATTENTION: Limpa espaço de trabalho.
rm(list = objects())
# Carrega pacotes.
library(car) # linearHypothesis()
library(multcomp) # glht()
library(tidyverse)
# Carrega funções.
source("mpaer_functions.R")
ls()## [1] "all_pairwise" "emmeans_interbloco"
## [3] "extract_cftest" "extract_linearHyphotesis"
## [5] "latin_square" "proj"Uma parte importantíssima do preparo dos dados é fazer com que a testemunha seja último nível dos fatores. Sugere-se usar o mesmo rótulo para ela em todos os fatores. Ou seja, da forma como o experimento será representado é como se a testemunha fosse um nível em cada um dos fatores.
Com a testemunha sendo o último nível, com o uso de contrastes de Helmert teremos a hipótese da média do tratamento adicional igual a média dos pontos fatoriais especificada.
Como variável resposta colocaremos apenas um ruído branco gaussiano. Conforme a tabela de ocorrência cruzada dos pontos experimentais, temos com esses dados simulados, um experimento com estrutura fatorial de 2 \(\times\) 4 mais um tratamento adicional. O nível "0" é o rótulo que indica testemunha como nível dos fatores A e B.
# Criando um conjunto de dados artificial.
da <- rbind(
# Parte fatorial.
crossing(A = 1:2,
B = 1:4,
r = 1:3),
# Parte adicional.
tibble(A = 0, B = 0, r = 1:3))
# Converte fatores experimentais qualitativos para classe
# correspondente. IMPORTANT: para o procedimento que será adotado, é
# preciso que o tratamento adicional seja o último nível.
da <- mutate(da,
A = fct_relevel(factor(A), "0", after = Inf),
B = fct_relevel(factor(B), "0", after = Inf))
# Adiciona uma resposta que é apenas ruído branco.
set.seed(18900217) # Nascimento de Fisher.
da$y <- 10 + round(rnorm(nrow(da)), digits = 2)
str(da)## Classes 'tbl_df', 'tbl' and 'data.frame': 27 obs. of 4 variables:
## $ A: Factor w/ 3 levels "1","2","0": 1 1 1 1 1 1 1 1 1 1 ...
## $ B: Factor w/ 5 levels "1","2","3","4",..: 1 1 1 2 2 2 3 3 3 4 ...
## $ r: int 1 2 3 1 2 3 1 2 3 1 ...
## $ y: num 9.38 9.5 9.21 10.65 9.96 ...# Estrutura de um fatorial 2 x 4 + 1 com 3 repetições.
xtabs(~A + B, data = da)## B
## A 1 2 3 4 0
## 1 3 3 3 3 0
## 2 3 3 3 3 0
## 0 0 0 0 0 3Quando ajustarmos um modelo utilizando a função lm() temos efeitos não estimados uma vez que não existem todos os pontos experimentais do experimento fatorial completo. A model.matrix() cria a matriz de delineamento com todos os termos de interação (abordagem maximal) e eles só serão estimados se houverem observações para todos os pontos experimentais do fatorial completo. Ao examinar a matriz de delieamento será visto que existem colunas linearmente dependentes. Os efeitos associados a essas colunas não serão estimados, obviamente.
É exatamente este aspecto da análise que dificulta: ser um fatorial incompleto. O que será mostrado aqui são formas de contornar esta complicação para que a análise possa ser feita da forma mais reproduzível possível.
# ATTENTION: alguns parâmetros não serão estimados porque são baseados
# em pontos experimentais que não existem.
m0 <- lm(y ~ A * B, data = da)
unique(model.matrix(m0)[, -1])## A2 A0 B2 B3 B4 B0 A2:B2 A0:B2 A2:B3 A0:B3 A2:B4 A0:B4 A2:B0
## 1 0 0 0 0 0 0 0 0 0 0 0 0 0
## 4 0 0 1 0 0 0 0 0 0 0 0 0 0
## 7 0 0 0 1 0 0 0 0 0 0 0 0 0
## 10 0 0 0 0 1 0 0 0 0 0 0 0 0
## 13 1 0 0 0 0 0 0 0 0 0 0 0 0
## 16 1 0 1 0 0 0 1 0 0 0 0 0 0
## 19 1 0 0 1 0 0 0 0 1 0 0 0 0
## 22 1 0 0 0 1 0 0 0 0 0 1 0 0
## 25 0 1 0 0 0 1 0 0 0 0 0 0 0
## A0:B0
## 1 0
## 4 0
## 7 0
## 10 0
## 13 0
## 16 0
## 19 0
## 22 0
## 25 1cbind(coef(m0))## [,1]
## (Intercept) 9.3633333
## A2 0.3266667
## A0 0.6133333
## B2 1.4500000
## B3 -0.1566667
## B4 0.4266667
## B0 NA
## A2:B2 -1.6933333
## A0:B2 NA
## A2:B3 -0.8233333
## A0:B3 NA
## A2:B4 -0.1100000
## A0:B4 NA
## A2:B0 NA
## A0:B0 NANa matriz do modelo, as colunas A0, B0, e por consequência, A0:B0 são todas iguais. Apenas o efeito associado a primeira delas será estimado, no caso, A0 será estimado. Essa coluna é a coluna que indica os pontos experimentais da testemunha. As colunas de parâmetros de segunda ordem envolvendo A0 ou B0 são nulas, só contém zeros. Esses efeitos não serão estimados também.
Os parâmetros com estimativas NA não foram estimados. Em geral existem duas principais razões para isso:
- Não existem pontos experimentais observados para estimar tais parâmetros. Esse é o caso dos parâmetros de interação envolvendo
A0ouB0. - Existem variáveis completamente redundantes sendo declaradas, portanto, presença de colinearidade perfeita, o que faz que efeitos não sejam estimados. É o caso das colunas
A0eB0que são indênticas por construção. Casos de dependências imperfeitas mas fortes ocorrem com frequência em modelos de regressão com variáveis preditoras quantitativas.
O uso dos contrastes de Helmert é um facilitador haja visto que a última hipótese corresponde a diferença de média do ponto adicional contra média dos pontos fatoriais. Vamos chamá-la de hipótese FvsA. Apesar da comodidade, outros contrastes poderiam ser empregados, incluindo os definidos pelo usuário. O importante é que os contrastes tenham o significado apropriado para representar as hipóteses em questão.
É muito comum que a hipótese FvsA seja testada. No entanto existem situações práticas em que ela pode não fazer sentido. Do ponto de vista estatístico, por exemplo, ela só terá significado quando efeito dos fatores A e B forem nulos, caso contrário a média dos pontos fatoriais é uma quantidade sem significado prático pois é uma média de médias de tratamentos que diferem entre si.
A seguir cria-se a matriz do delineamento usando os contrastes de Helmert e ajusta-se o modelo com a função aov(). Internamente, a aov() faz uma chamada da lm(). O que difere são métodos adicionais para a classe aov e a possibilidade de declarar termos de erro na fórmula (Error()), necessários para análise de experimento em parcelas subdivididas, por exemplo.
# Criação da matriz de delineamento.
X_full <- model.matrix(~A * B,
data = da,
contrasts = list(A = contr.helmert,
B = contr.helmert))
# colnames(X_full)
# unique(X_full)
# Codificação usada no contraste de Helmert (i.e. back difference). A
# última hipótese é se há diferença entre tratamento adicional contra a
# média dos níveis anteriores.
attr(X_full, "contrasts")## $A
## [,1] [,2]
## 1 -1 -1
## 2 1 -1
## 0 0 2
##
## $B
## [,1] [,2] [,3] [,4]
## 1 -1 -1 -1 -1
## 2 1 -1 -1 -1
## 3 0 2 -1 -1
## 4 0 0 3 -1
## 0 0 0 0 4# Ajuste do modelo com `aov()` para usar `split =`.
m0 <- aov(y ~ 0 + X_full, data = da)
cbind(coef(m0))## [,1]
## X_full(Intercept) 9.744444444
## X_fullA1 -0.165000000
## X_fullA2 0.116111111
## X_fullB1 0.301666667
## X_fullB2 -0.290000000
## X_fullB3 0.090000000
## X_fullA1:B1 -0.423333333
## X_fullA1:B2 0.003888889
## X_fullA1:B3 0.091111111A função a aov(), diferente da função lm() abandona os termos de efeito não estimável. O objeto retornado por ela tem classe aov e possui métodos adicionais e métodos modificados. O método summary() retorna o quadro de análise de variância e tem um argumento split que permite fazer partições das somas de quadrados. Apenas é necessário informar a posição dos coeficientes para indicar como a partição será feita.
# Linhas distintas da matriz do modelo com as colunas de efeitos
# estimáveis.
unname(unique(model.matrix(m0)[, which(!is.na(coef(m0)))]))## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 1 -1 -1 -1 -1 -1 -1 1 1
## [2,] 1 -1 -1 1 -1 -1 -1 -1 -1
## [3,] 1 -1 -1 0 2 -1 -1 0 0
## [4,] 1 -1 -1 0 0 3 -1 0 0
## [5,] 1 1 -1 -1 -1 -1 -1 -1 1
## [6,] 1 1 -1 1 -1 -1 -1 1 -1
## [7,] 1 1 -1 0 2 -1 -1 0 0
## [8,] 1 1 -1 0 0 3 -1 0 0
## [9,] 1 0 2 0 0 0 4 0 0O 2º coeficiente corresponde ao efeito do fator A. Ele possui 2 níveis já que o seu terceiro nível é na realidade o ponto adicional, também presente como último nível do fator B. Os coefientes de 4 a 6 correspondem ao efeito do fator B que possui quatro níveis, portanto, 3 efeitos estimados (o quinto nível não estimado é o ponto adicional já acomodado pela coluna criada usando o fator A). Os coeficientes de 7 a 9 correspondem ao efeito da interação entre A e B (1 \(\times\) 3 = 3 graus de liberdade). Eles surgem do produto da coluna 2 por cada uma das colunas de 4 a 6. Finalmente, o coeficiente na posição 3 é o da hipótese FvsA que estabelece que a diferença entre o ponto adicional com relação à média dos pontos fatoriais é nula.
Como a variável resposta foi simulada com média zero, não se espera observar rejeição para nenhuma das hipóteses. Caso ocorra, é meramente circunstâncial. Depois serão analisados dados reais para que possamos exercitar a intepretação.
A lista a seguir indica a posição das colunas/efeitos associados com cada termo do modelo. As hipóteses sobre termos de maior ordem estão no final seguindo a lógica: hipótese sob a interação dupla > hipóteses sob os efeitos principais > hipótese FvsA.
# Posição das colunas/efeitos de cada termo do modelo.
ef <- list("FvsA" = 3,
"A" = 2,
"B" = 4:6,
"A:B" = 7:9)
summary(m0, split = list(X_full = ef))## Df Sum Sq Mean Sq F value Pr(>F)
## X_full 9 2531.6 281.29 432.711 <2e-16 ***
## X_full: FvsA 1 0.3 0.32 0.498 0.490
## X_full: A 1 0.7 0.65 1.005 0.329
## X_full: B 3 4.7 1.57 2.411 0.100
## X_full: A:B 3 2.7 0.92 1.409 0.273
## Residuals 18 11.7 0.65
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1A função lm() assume distribuição gaussiana para a variável resposta e a aov() permite fazer o particionamento das somas de quadrados para as hipóteses relacionas aos efeitos experimentais em questão. No entanto, para outras classes de modelo (GLM, sobrevivência, modelos de efeitos aleatórios), abordagem equivalente a lm() e aov() não se aplicam. Então, o que se pode usar são recursos como glht() e linearHypothesis() que serão demontrados a seguir. Com essas funções pode-se tanto obter uma estatística para a hipótese global de efeito de um termo do modelo como aplicar testes para outras hipóteses.
O primeiro passo para usar tais funções é ajustar um modelo de classe lm() com todos os efeitos estimáveis. Para isso passamos a matriz com colunas de efeito estimáveis eliminando as colunas que sabidamente correspondem aos efeitos não estimáveis.
# A `aov()` já abandona parâmetros não estimados do vetor. Mas para
# modelos de outras classes, terá que ser feito manualmente.
X <- X_full[, sub("X_full", "", names(coef(m0)))]
# Modelo linear com matriz onde todos efeitos são estimáveis.
m1 <- lm(y ~ 0 + X, data = da)
cbind(lm = coef(m1), aov = coef(m0))## lm aov
## X(Intercept) 9.744444444 9.744444444
## XA1 -0.165000000 -0.165000000
## XA2 0.116111111 0.116111111
## XB1 0.301666667 0.301666667
## XB2 -0.290000000 -0.290000000
## XB3 0.090000000 0.090000000
## XA1:B1 -0.423333333 -0.423333333
## XA1:B2 0.003888889 0.003888889
## XA1:B3 0.091111111 0.091111111# O mesmo de antes mas passando a matriz de delineamento dividida.
m2 <- lm(y ~ X[, ef[[1]], drop = FALSE] +
X[, ef[[2]], drop = FALSE] +
X[, ef[[3]], drop = FALSE] +
X[, ef[[4]], drop = FALSE],
data = da)
# O mesmo resultado de usar `aov(..., split = ...)`.
anova(m2)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## X[, ef[[1]], drop = FALSE] 1 0.3236 0.32356 0.4977 0.4895
## X[, ef[[2]], drop = FALSE] 1 0.6534 0.65340 1.0051 0.3294
## X[, ef[[3]], drop = FALSE] 3 4.7028 1.56761 2.4115 0.1004
## X[, ef[[4]], drop = FALSE] 3 2.7488 0.91626 1.4095 0.2725
## Residuals 18 11.7012 0.65007# Estatística F com hipóteses marginais.
drop1(m2, test = "F")## Single term deletions
##
## Model:
## y ~ X[, ef[[1]], drop = FALSE] + X[, ef[[2]], drop = FALSE] +
## X[, ef[[3]], drop = FALSE] + X[, ef[[4]], drop = FALSE]
## Df Sum of Sq RSS AIC F value
## <none> 11.701 -4.5759
## X[, ef[[1]], drop = FALSE] 1 0.3236 12.025 -5.8395 0.4977
## X[, ef[[2]], drop = FALSE] 1 0.6534 12.355 -5.1088 1.0051
## X[, ef[[3]], drop = FALSE] 3 4.7028 16.404 -1.4544 2.4115
## X[, ef[[4]], drop = FALSE] 3 2.7488 14.450 -4.8789 1.4095
## Pr(>F)
## <none>
## X[, ef[[1]], drop = FALSE] 0.4895
## X[, ef[[2]], drop = FALSE] 0.3294
## X[, ef[[3]], drop = FALSE] 0.1004
## X[, ef[[4]], drop = FALSE] 0.2725# car::Anova(m2, type = 3) # Idem a drop1().
# Estimativas dos efeitos são identicas.
cbind(aov = coef(m0), lm = coef(m1), lm = coef(m2))## aov lm lm
## X_full(Intercept) 9.744444444 9.744444444 9.744444444
## X_fullA1 -0.165000000 -0.165000000 0.116111111
## X_fullA2 0.116111111 0.116111111 -0.165000000
## X_fullB1 0.301666667 0.301666667 0.301666667
## X_fullB2 -0.290000000 -0.290000000 -0.290000000
## X_fullB3 0.090000000 0.090000000 0.090000000
## X_fullA1:B1 -0.423333333 -0.423333333 -0.423333333
## X_fullA1:B2 0.003888889 0.003888889 0.003888889
## X_fullA1:B3 0.091111111 0.091111111 0.091111111A função multcomp::cftest() testa hipóteses sobre vetores de parâmetros pelo teste de Wald com estatística F ou \(\chi\)-quadrado. Para isso, é só indicar o nome ou posição dos parâmetros sob hipótese. Note que a estatística F é a mesma daquela vista no summary() do modelo m0.
Em modelos de outras classes (glm, survreg, lmerMod, etc) pode haver diferença entre o resultado do F do Wald pro F do retornado pelo método anova(). Por exemplo, um teste de Wald (que é uma aproximação quadrática da verossimilhança) poderá diferir de uma razão de deviances ou razão de verossimilhanças.
# `multcomp::cftest()` é aplicável a uma ampla classe de modelos.
cftest(m1, parm = ef[[1]], test = Ftest()) # A.
cftest(m1, parm = ef[[2]], test = Ftest()) # B.
cftest(m1, parm = ef[[3]], test = Ftest()) # A:B.
cftest(m1, parm = ef[[4]], test = Ftest()) # fat_vs_ad.A chamada da cftest() é bastante simples. Para não ter um resultado tão extenso, vamos usar as funções da família apply para evitar repetição de código e integrar os resultados.
# Serializa a aplicação da `cftest()` e extração das estatísticas.
lapply(ef, FUN = cftest, model = m1, test = Ftest()) %>%
lapply(extract_cftest) %>%
do.call(what = rbind)## df1 df2 fstat pvalue
## FvsA 1 18 0.4977381 0.4895261
## A 1 18 1.0051277 0.3293609
## B 3 18 2.4114621 0.1004375
## A:B 3 18 1.4094794 0.2725467Com a linearHypothesis() podemos obter os mesmos resultados. A diferença é que ela precisa de uma matriz que espeficique as hipóteses. Por isso, pode-se especificar hipóteses mais complexas que não seriam possíveis de avaliar com a cftest(). Um exemplo é quando fazemos análise de covariância, caso que será mostrado em outro capítulo.
# Matriz de hipóteses lineares (linear hypothesis matrix).
m <- diag(length(coef(m1)))
lhm <- ef
for (i in 1:length(ef)) {
lhm[[i]] <- m[ef[[i]], , drop = FALSE]
}
lhm## $FvsA
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 0 0 1 0 0 0 0 0 0
##
## $A
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 0 1 0 0 0 0 0 0 0
##
## $B
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 0 0 0 1 0 0 0 0 0
## [2,] 0 0 0 0 1 0 0 0 0
## [3,] 0 0 0 0 0 1 0 0 0
##
## $`A:B`
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## [1,] 0 0 0 0 0 0 1 0 0
## [2,] 0 0 0 0 0 0 0 1 0
## [3,] 0 0 0 0 0 0 0 0 1# `car::linearHypothesis()` é aplicável a uma ampla classe de modelos.
linearHypothesis(m1, hypothesis.matrix = lhm[[1]], test = "F") # FvsA.## Linear hypothesis test
##
## Hypothesis:
## XA2 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 19 12.025
## 2 18 11.701 1 0.32356 0.4977 0.4895linearHypothesis(m1, hypothesis.matrix = lhm[[2]], test = "F") # A.## Linear hypothesis test
##
## Hypothesis:
## XA1 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 19 12.355
## 2 18 11.701 1 0.6534 1.0051 0.3294linearHypothesis(m1, hypothesis.matrix = lhm[[3]], test = "F") # B.## Linear hypothesis test
##
## Hypothesis:
## XB1 = 0
## XB2 = 0
## XB3 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 21 16.404
## 2 18 11.701 3 4.7028 2.4115 0.1004linearHypothesis(m1, hypothesis.matrix = lhm[[4]], test = "F") # A:B.## Linear hypothesis test
##
## Hypothesis:
## XA1:B1 = 0
## XA1:B2 = 0
## XA1:B3 = 0
##
## Model 1: restricted model
## Model 2: y ~ 0 + X
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 21 14.450
## 2 18 11.701 3 2.7488 1.4095 0.2725Para evitar repetição de código e resultado extenso, vamos usar novamente a família apply. Com isso teremos um resultado compacto com o emprego de poucas linhas de código.
# Serializa a aplicação da `linearHypothesis()`.
lapply(lhm, FUN = linearHypothesis, model = m1, test = "F") %>%
lapply(extract_linearHyphotesis) %>%
do.call(what = rbind)A estatística F foi calculada para testas 4 hipóteses usando o método de Wald (hipóteses marginais) e o quadro de análise de variância (hipóteses sequenciais). O valor das estatísticas foi o mesmo, independente do método. Isso só aconteceu porque:
- Os efeitos são ortogonais entre si, o que algo desejável e conseguido em experimentos planejados. A ortogonalidade do delineamento faz com que não exista diferença de resultado entre dispositivos de testes de hipóteses sequenciais e marginais.
- O modelo considerou distribuição gaussiana. Isso faz com que não exista diferença entre o teste de Wald (baseado na aproximação quadrática da função de log-verossimilhança) e o teste baseado na diferença entre somas de quadrados residuais de modelos encaixados (que tem relação com o teste da razão de verossimilhanças).
Quando as condições 1 ou 2 não estão presentes, em geral, há diferença entre testes sequenciais e marginais e Wald e testes entre modelos encaixados.
Nas instruções acima testamos as hipóteses globais de feito dos fatores experimentais. As hipóteses devem ser interpretadas conforme o grau dos termos: efeito da interação A:B > efeito de A, efeito de B > hipóteses FvsA. Agora será demonstrado como testar as hipóteses de comparações múltiplas entre médias para os níveis de cada fator.
Vamos testar as hipóteses sobre as diferenças entre médias para os níveis do fator A, depois entre médias para os níveis do fator B. Ambos assumem que não houve interação entre os fatores A e B. Quando há interação, recomenda-se fazer o estudo de um fator fixando-se os níveis dos demais. Então serão obtidas as comparações múltiplas de médias dos dois sentidos: para níveis de B fixando o nível de A e para níveis de A fixando o nível de B. Por último, será obtido o teste que envolve a média do ponto adicional contra a média dos pontos fatoriais. Essa hipótese já foi testada, porém não foi obtido a estimativa da diferença entre tais médias.
# Pontos experimentais únicos de A * B.
i <- da %>% select(A, B) %>% duplicated()
pts <- da[!i, c("A", "B")] # Pontos experimentais.
X_un <- X[!i, ] # Estimativas.
cbind(pts, "|" = "|", X_un)## A B | (Intercept) A1 A2 B1 B2 B3 A1:B1 A1:B2 A1:B3
## 1 1 1 | 1 -1 -1 -1 -1 -1 1 1 1
## 4 1 2 | 1 -1 -1 1 -1 -1 -1 1 1
## 7 1 3 | 1 -1 -1 0 2 -1 0 -2 1
## 10 1 4 | 1 -1 -1 0 0 3 0 0 -3
## 13 2 1 | 1 1 -1 -1 -1 -1 -1 -1 -1
## 16 2 2 | 1 1 -1 1 -1 -1 1 -1 -1
## 19 2 3 | 1 1 -1 0 2 -1 0 2 -1
## 22 2 4 | 1 1 -1 0 0 3 0 0 3
## 25 0 0 | 1 0 2 0 0 0 0 0 0# Contrastes dois a dois (pairwise) entre níveis de A.
L_A <- do.call(rbind, by(X_un, pts$A, colMeans))
K_A <- all_pairwise(head(L_A, n = -1))
summary(glht(m1, linfct = K_A))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.3300 0.3292 1.003 0.329
## (Adjusted p values reported -- single-step method)# Contrastes dois a dois (pairwise) entre níveis de B.
L_B <- do.call(rbind, by(X_un, pts$B, colMeans))
K_B <- all_pairwise(head(L_B, n = -1))
summary(glht(m1, linfct = K_B))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.6033 0.4655 -1.296 0.5769
## 1vs3 == 0 0.5683 0.4655 1.221 0.6221
## 1vs4 == 0 -0.3717 0.4655 -0.798 0.8542
## 2vs3 == 0 1.1717 0.4655 2.517 0.0909 .
## 2vs4 == 0 0.2317 0.4655 0.498 0.9586
## 3vs4 == 0 -0.9400 0.4655 -2.019 0.2177
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)# Estudo da interação: entre níveis de B dentro de A.
L_BinA <- by(X_un, pts$A, FUN = as.matrix)
L_BinA <- lapply(L_BinA, "rownames<-", NULL)
lapply(head(L_BinA, n = -1),
FUN = function(s) {
summary(glht(m1, linfct = all_pairwise(s)))
})## $`1`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -1.4500 0.6583 -2.203 0.160
## 1vs3 == 0 0.1567 0.6583 0.238 0.995
## 1vs4 == 0 -0.4267 0.6583 -0.648 0.915
## 2vs3 == 0 1.6067 0.6583 2.441 0.105
## 2vs4 == 0 1.0233 0.6583 1.554 0.428
## 3vs4 == 0 -0.5833 0.6583 -0.886 0.812
## (Adjusted p values reported -- single-step method)
##
##
## $`2`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.2433 0.6583 0.370 0.982
## 1vs3 == 0 0.9800 0.6583 1.489 0.464
## 1vs4 == 0 -0.3167 0.6583 -0.481 0.962
## 2vs3 == 0 0.7367 0.6583 1.119 0.683
## 2vs4 == 0 -0.5600 0.6583 -0.851 0.830
## 3vs4 == 0 -1.2967 0.6583 -1.970 0.236
## (Adjusted p values reported -- single-step method)# Estudo da interação: entre níveis de A dentro de B.
L_AinB <- by(X_un, pts$B, FUN = as.matrix)
L_AinB <- lapply(L_AinB, "rownames<-", NULL)
lapply(head(L_AinB, n = -1),
FUN = function(s) {
summary(glht(m1, linfct = all_pairwise(s)))
})## $`1`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.3267 0.6583 -0.496 0.626
## (Adjusted p values reported -- single-step method)
##
##
## $`2`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 1.3667 0.6583 2.076 0.0525 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)
##
##
## $`3`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 0.4967 0.6583 0.754 0.46
## (Adjusted p values reported -- single-step method)
##
##
## $`4`
##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1vs2 == 0 -0.2167 0.6583 -0.329 0.746
## (Adjusted p values reported -- single-step method)# Contraste do ponto adicional contra a média dos pontos fatoriais.
L_T <- by(X_un, pts$A == "0", colMeans)
summary(glht(m1, linfct = rbind(L_T[[1]] - L_T[[2]])))##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = y ~ 0 + X, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## 1 == 0 -0.3483 0.4937 -0.706 0.49
## (Adjusted p values reported -- single-step method)A título de verificação, vamos calcular as médias amostrais. Como tem-se um experimento balanceado e de efeitos ortogonais, as médias amostrais serão iguais as médias ajustadas. Dessa forma, caso hajam diferenças é sinal de que o código precisa ser revisto. É importante enfatizar que em modelos mais gerais (glm, survreg, lmerMod, etc) ou para experimentos não balanceados ou de efeitos não ortogonais, as médias amostrais não serão iguais as médias ajustadas.
# Diferenças entre médias amostrais apenas para verificação.
# Entre níveis de A.
da %>% group_by(A) %>% summarise(m = -mean(y)) %>%
pull(m) %>% head(n = -1) %>%
outer(., ., "-") %>% as.dist()## 1
## 2 0.33# Entre níveis de B.
da %>% group_by(B) %>% summarise(m = -mean(y)) %>%
pull(m) %>% head(n = -1) %>%
outer(., ., "-") %>% as.dist()## 1 2 3
## 2 -0.6033333
## 3 0.5683333 1.1716667
## 4 -0.3716667 0.2316667 -0.9400000# Entre a testemunha e os pontos fatoriais.
da %>% group_by(A != "0") %>% summarise(m = -mean(y)) %>%
pull(m) %>% diff() %>% setNames(c("FvsA"))## FvsA
## 0.3483333Na próxima seção faremos a análise de um conjunto de dados real para poder exercitar a interpretação e reafirmar o aprendizado.
8.3 Efeito da fonte e modo de aplicação de adubação na produtividade
Os dados que serão analisados são de um experimento fatorial duplo 4 \(\times\) 2 com um controle negativo, comumente representado por 4 \(\times\) 2 \(+\) 1. Os dados são contribuição do meu colaborador e amigo Fabio Ono. Os dados estão no objeto fat2_adic do pacote EACS. A variável resposta é a produtividade da cultura em função de 4 fontes de adubação e dois modos de aplicação (sulco e lanço). A testemunha negativa é o tratamento que não recebeu adubação. A análise desses dados está disponível como vinheta no pacote EACS. Aqui a análise será refeita com mais discussão e algumas modificações.
url <- "https://raw.githubusercontent.com/walmes/EACS/master/data-raw/fat2_adic.csv"
tb <- read_delim(url, delim = ";", locale = locale(decimal_mark = "."))
attr(tb, "spec") <- NULLA primeira dica importante para análise de experimentos fatoriais com tratamentos adicionais é: deixe os tratamentos adicionais serem os últimos níveis em todos os fatores.
Na tabela tb foi usado controle como rótulo do ponto adicional (sem adubação) tanto na coluna do fator fonte quanto na coluna do fator modo. Não é obrigatório que os nomes sejam os mesmos em cada fator mas é recomendável por simplicidade. Não se deve colocar NA (indicador de valor ausente) porque isso leva ao descarte das observações pelas funções de ajuste que precisam de observações completas.
# Coloca o controle como último nível dos fatores.
tb <- tb %>%
mutate(bloc = factor(bloc),
fonte = fct_relevel(fonte, "controle", after = Inf),
modo = fct_relevel(modo, "controle", after = Inf))
summary(tb)## fonte modo bloc prod
## A :8 lanco :16 1:9 Min. :44.06
## B :8 sulco :16 2:9 1st Qu.:56.17
## C :8 controle: 4 3:9 Median :60.54
## D :8 4:9 Mean :59.29
## controle:4 3rd Qu.:63.12
## Max. :68.68# Contagem de repetições por ponto experimental.
xtabs(~fonte + modo, data = tb)## modo
## fonte lanco sulco controle
## A 4 4 0
## B 4 4 0
## C 4 4 0
## D 4 4 0
## controle 0 0 4A tabela de frequência mostra o que já sabemos: temos um experimento fatorial 4 \(\times\) 2 \(+\) 1 com quatro repetições. O experimento foi feito no delineamento de blocos casualizados. O último em cada fator corresponde ao ponto adicional, conforme recomendado.
Estamos lidando com dados reais. Por isso, é imprescindível fazer uma análise gráfica preliminar para diagosticar possíveis problemas, antecipar a existência de efeitos, conhecer a relação entre as variáveis. Esse conhecimento facilita o desenvolvimento do restante da análise dando segurança e clareza e também contribuirá para melhor comunicação dos resultados.
# Análise exploratória.
gg1 <- ggplot(data = tb,
mapping = aes(x = fonte, y = prod)) +
facet_grid(facets = . ~ modo, scale = "free", space = "free") +
geom_jitter(alpha = 0.5, width = 0.1, height = 0) +
stat_summary(mapping = aes(group = 1), geom = "line", fun.y = "mean")
gg2 <- ggplot(data = tb,
mapping = aes(x = modo, y = prod)) +
facet_grid(facets = . ~ fonte, scale = "free", space = "free") +
geom_jitter(alpha = 0.5, width = 0.1, height = 0) +
stat_summary(mapping = aes(group = 1), geom = "line", fun.y = "mean")
gridExtra::grid.arrange(gg1, gg2, ncol = 1)## geom_path: Each group consists of only one observation. Do you
## need to adjust the group aesthetic?
## geom_path: Each group consists of only one observation. Do you
## need to adjust the group aesthetic?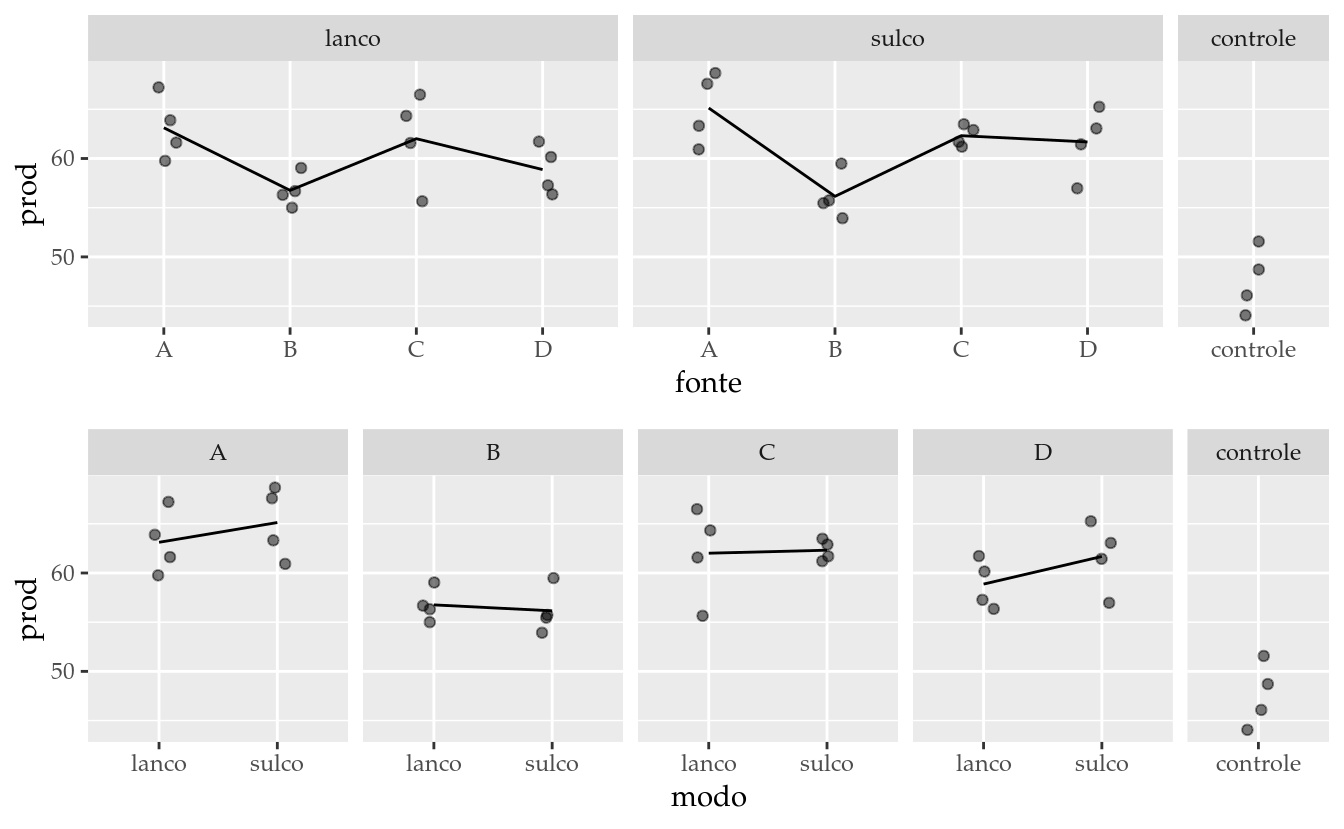
Considerando a linha média e a dispersão dos dados, o gráfico de cima mostra que existe uma leve diferença do desempenho da fonte B em relação as outras fontes de adubação e que o desempenho do controle está abaixo dos pontos fatoriais. O gráfico de baixo indica haver pouca diferença entre as formas de aplicação. Além disso, não fornece evidências que marcam interação entre os fatores.
A especificação do modelo via fórmula é a mesma, seja o experimento fatorial completo ou não. No entanto, devido o fatorial com tratamento adicional na realidade ser um fatorial completo com combinações ausentes, alguns efeitos não serão estimados, justamente por causa dos pontos experimentais ausentes.
# Ajuste do modelo via especificação por fórmula.
m0 <- lm(prod ~ bloc + fonte * modo, data = tb)
cbind(coef(m0))## [,1]
## (Intercept) 62.5927778
## bloc2 1.4148889
## bloc3 0.3732222
## bloc4 0.3277778
## fonteB -6.3635000
## fonteC -1.1107500
## fonteD -4.2482500
## fontecontrole -15.5150000
## modosulco 2.0082500
## modocontrole NA
## fonteB:modosulco -2.6142500
## fonteC:modosulco -1.7017500
## fonteD:modosulco 0.7980000
## fontecontrole:modosulco NA
## fonteB:modocontrole NA
## fonteC:modocontrole NA
## fonteD:modocontrole NA
## fontecontrole:modocontrole NA# Diagnóstico do modelo.
par(mfrow = c(1, 3)); plot(m0, which = c(1, 2, 3)); layout(1)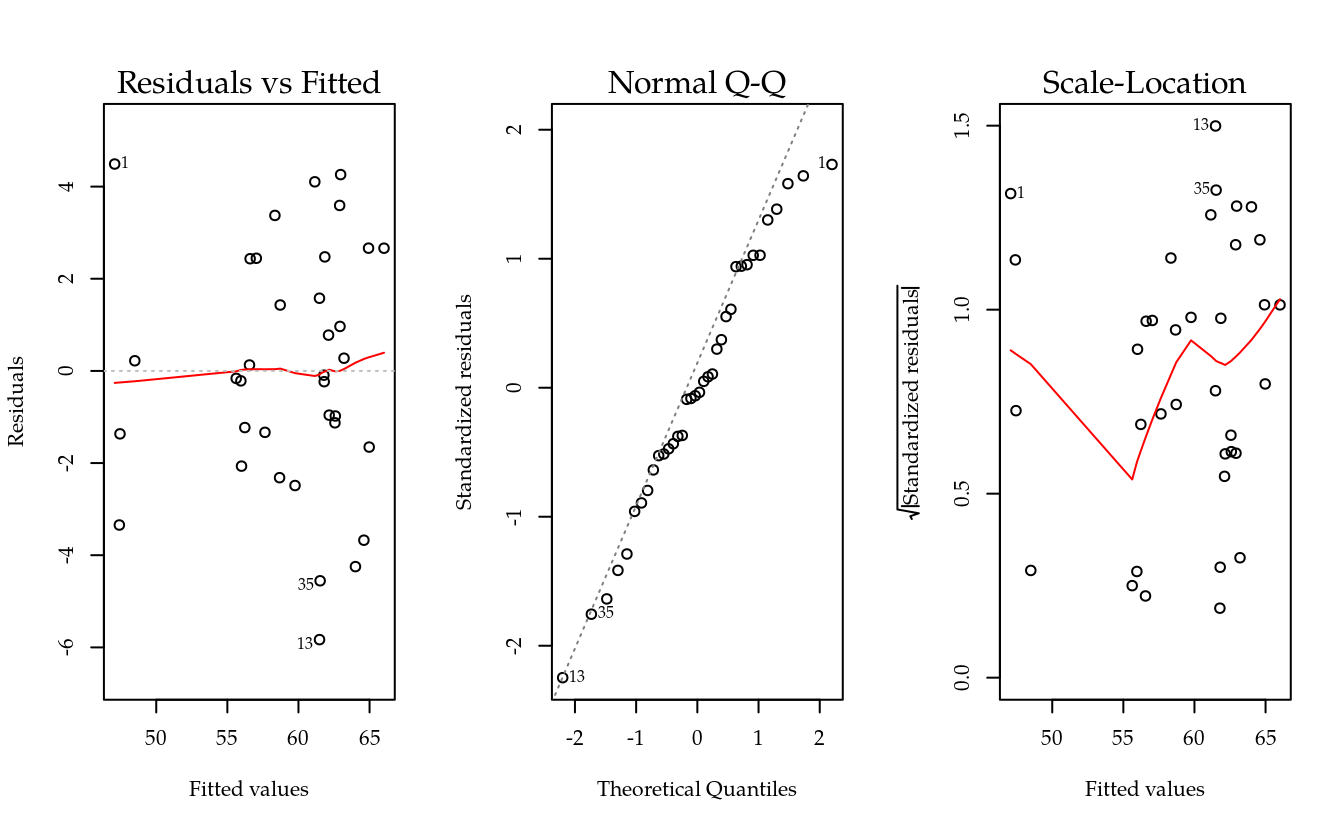
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.16 3.388 0.3355 0.7998
## fonte 4 871.24 217.811 21.5688 1.173e-07 ***
## modo 1 10.19 10.193 1.0093 0.3251
## fonte:modo 3 14.55 4.849 0.4801 0.6991
## Residuals 24 242.36 10.098
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Os gráficos sobre os resíduos não apontam nenhuma evidência contra o bom atendimentos dos pressupostos. Então nenhuma providência será necessária para melhorar o atendimento dos pressupostos.
No quadro de análise de variância mostra a não existência de interação entre fonte e modo de aplicação, a 5% de significância. Também não foi significativo o efeito de modo de aplicação. Restou, portanto, apenas o efeito de fonte de adubo.
O fator fonte tem 4 graus de liberdade (GL) enquanto que modo tem apenas 1 e a interação fonte:modo tem 3. Os 4 GL de fonte decorrem do fator ter 5 níveis (4 níveis próprios e o controle). Porém, o modo que deveria ter 2, porque têm 3 níveis (2 níveis próprios e o controle), fica com apenas 1 porque o efeito de controle já foi acomodado em fonte. Já a interação resulta do produto dos graus de liberdade próprios, ou seja, \((4 - 1) \times (2 - 1) = 3\).
As combinações que não existem possuem valor igual a NA no vetor de estimativas. Isso indica que esses efeitos não foram estimados. De fato, só seriam estimados se estes pontos experimentais existissem.
Para fatiar as somas de quadrados no quadro de análise de variância corretamente podemos definir o modelo com a aov() e usar o parâmetro split do método summary() ou podemos usar a lm() passando o termos por meio de matrizes. Para facilmente conseguir esses resultados, serão utilizados os contrastes de Helmert. Nestes contrastes, a última coluna representa o contraste do ponto adicional contra os pontos fatoriais.
# Cria a matriz do delineamento completa.
X_full <- model.matrix(~bloc + fonte * modo,
data = tb,
contrasts = list(fonte = "contr.helmert",
modo = "contr.helmert"))
# Modelo ajustado passando a matriz completa.
m0 <- aov(prod ~ 0 + X_full, data = tb)
# Efeitos.
ef <- list("bloc" = 2:4,
"FvsA" = 8,
"fonte" = 5:7,
"modo" = 9,
"fonte:modo" = 10:12)
# Desdobramento das somas de quadrados.
summary(m0, split = list("X_full" = ef))## Df Sum Sq Mean Sq F value Pr(>F)
## X_full 12 127476 10623 1051.951 < 2e-16 ***
## X_full: bloc 3 10 3 0.336 0.799767
## X_full: FvsA 1 615 615 60.873 4.9e-08 ***
## X_full: fonte 3 257 86 8.467 0.000517 ***
## X_full: modo 1 10 10 1.009 0.325086
## X_full: fonte:modo 3 15 5 0.480 0.699135
## Residuals 24 242 10
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Matriz de delineamento apenas com colunas de efeitos estimados.
X <- X_full[, sub("X_full", "", names(coef(m0)))]
# Ajuste do modelo passando a matriz particionada.
m1 <- lm(prod ~ 1 +
X[, ef[[1]], drop = FALSE] +
X[, ef[[2]], drop = FALSE] +
X[, ef[[3]], drop = FALSE] +
X[, ef[[4]], drop = FALSE] +
X[, ef[[5]], drop = FALSE], data = tb)
anova(m1)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## X[, ef[[1]], drop = FALSE] 3 10.16 3.39 0.3355 0.7997666
## X[, ef[[2]], drop = FALSE] 1 614.72 614.72 60.8728 4.903e-08 ***
## X[, ef[[3]], drop = FALSE] 3 256.52 85.51 8.4675 0.0005169 ***
## X[, ef[[4]], drop = FALSE] 1 10.19 10.19 1.0093 0.3250863
## X[, ef[[5]], drop = FALSE] 3 14.55 4.85 0.4801 0.6991352
## Residuals 24 242.36 10.10
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Atualiza com a matriz sem particionar.
m0 <- update(m1, . ~0 + X)Pelo quadro de análise de variância, não existe interação entre fonte e modo. Tão pouco se mostrou relevante o efeito de modo de aplicação. Porém, a fonte apresentou efeito. Dito isso, a hipótese FvsA perde significado porque o contraste da testemunha com a média das fontes é sem sentido prático. O único possível sentido seria, por exemplo, comparar a produtividade média de um hectare cultivado totalmente com a testemunha com um hectare no qual uma fonte de adubação foi usada em 1/4 de área de cultivo. Na prática não é isso que fazemos, então embora haja uma forma de interpretar, ela não é praticada.
As médias ajustadas são médias obtidas por meio de funções lineares dos efeitos estimados. Em experimentos balanceados e com efeito ortogonais, as médias ajustadas coincidem as com as médias amostrais. No entanto, em situações mais gerais, as médias amostrais não são os estimadores corretos das médias populacionais. Exceto para situações muito específicas, trabalhar com as médias ajustadas é sempre o adequado.
# Matriz do modelo descartadas as linhas repetidas, caso hajam.
X_un <- unique(X)
# Fatores do modelo, removidas as linhas repetidas também.
pts <- unique(subset(tb, select = names(attr(X_full, "contrasts"))))
# Testa se os tamanhos são iguais.
stopifnot(nrow(pts) == nrow(X_un))
# Cria um objeto agregado considerando os níveis de fonte e modo.
grp <- pts %>% group_by(fonte, modo)
# Matriz com termos para determinar médias ajustadas para os pontos
# experimentais.
pts <- group_keys(grp)
X_ls <-
by(X_un, INDICES = group_indices(grp), FUN = colMeans) %>%
do.call(what = rbind)
# Entenda os pesos, principalmente para os níveis de bloco.
MASS::fractions(t(X_ls))## 1 2 3 4 5 6 7 8 9
## (Intercept) 1 1 1 1 1 1 1 1 1
## bloc2 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
## bloc3 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
## bloc4 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4 1/4
## fonte1 -1 -1 1 1 0 0 0 0 0
## fonte2 -1 -1 -1 -1 2 2 0 0 0
## fonte3 -1 -1 -1 -1 -1 -1 3 3 0
## fonte4 -1 -1 -1 -1 -1 -1 -1 -1 4
## modo1 -1 1 -1 1 -1 1 -1 1 0
## fonte1:modo1 1 -1 -1 1 0 0 0 0 0
## fonte2:modo1 1 -1 1 -1 -2 2 0 0 0
## fonte3:modo1 1 -1 1 -1 1 -1 -3 3 0# Médias amostrais e ajustadas apenas para conferir.
tb %>%
group_by(fonte, modo) %>%
summarise("média amostral" = mean(prod)) %>%
bind_cols("média ajustada" = c(X_ls %*% coef(m0)))## # A tibble: 9 x 4
## # Groups: fonte [5]
## fonte modo `média amostral` `média ajustada`
## <fct> <fct> <dbl> <dbl>
## 1 A lanco 63.1 63.1
## 2 A sulco 65.1 65.1
## 3 B lanco 56.8 56.8
## 4 B sulco 56.2 56.2
## 5 C lanco 62.0 62.0
## 6 C sulco 62.3 62.3
## 7 D lanco 58.9 58.9
## 8 D sulco 61.7 61.7
## 9 controle controle 47.6 47.6Agora com X_ls contendo os coeficientes para obter a média ajustada para cada ponto experimental em pts, pode-se então obter comparações entre as médias marginais para os níveis de fonte. Os coeficientes para a média (ajustada) marginal são obtididos por agregação das linhas da matriz por nível do faltor. Confira no bloco a seguir.
# Funções lineares para média ajustada para níveis de fonte.
L_fonte <- by(X_ls, INDICES = pts$fonte, FUN = colMeans) %>%
do.call(what = rbind)
MASS::fractions(t(L_fonte))## A B C D controle
## (Intercept) 1 1 1 1 1
## bloc2 1/4 1/4 1/4 1/4 1/4
## bloc3 1/4 1/4 1/4 1/4 1/4
## bloc4 1/4 1/4 1/4 1/4 1/4
## fonte1 -1 1 0 0 0
## fonte2 -1 -1 2 0 0
## fonte3 -1 -1 -1 3 0
## fonte4 -1 -1 -1 -1 4
## modo1 0 0 0 0 0
## fonte1:modo1 0 0 0 0 0
## fonte2:modo1 0 0 0 0 0
## fonte3:modo1 0 0 0 0 0results <- wzRfun::apmc(X = L_fonte,
model = m0,
focus = "fonte",
cld2 = TRUE) %>%
mutate(fonte = factor(fonte, levels = levels(tb$fonte)),
cld = wzRfun::ordered_cld(cld, fit))
results## fonte fit lwr upr cld
## 1 A 64.12587 61.80704 66.44471 a
## 2 B 56.45525 54.13641 58.77409 b
## 3 C 62.16425 59.84541 64.48309 a
## 4 D 60.27662 57.95779 62.59546 ab
## 5 controle 47.60675 44.32742 50.88608 c# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = fonte, y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.1f%s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Fonte de adubação",
y = expression("Produtividade" ~ (sacas ~ ha^{-1})))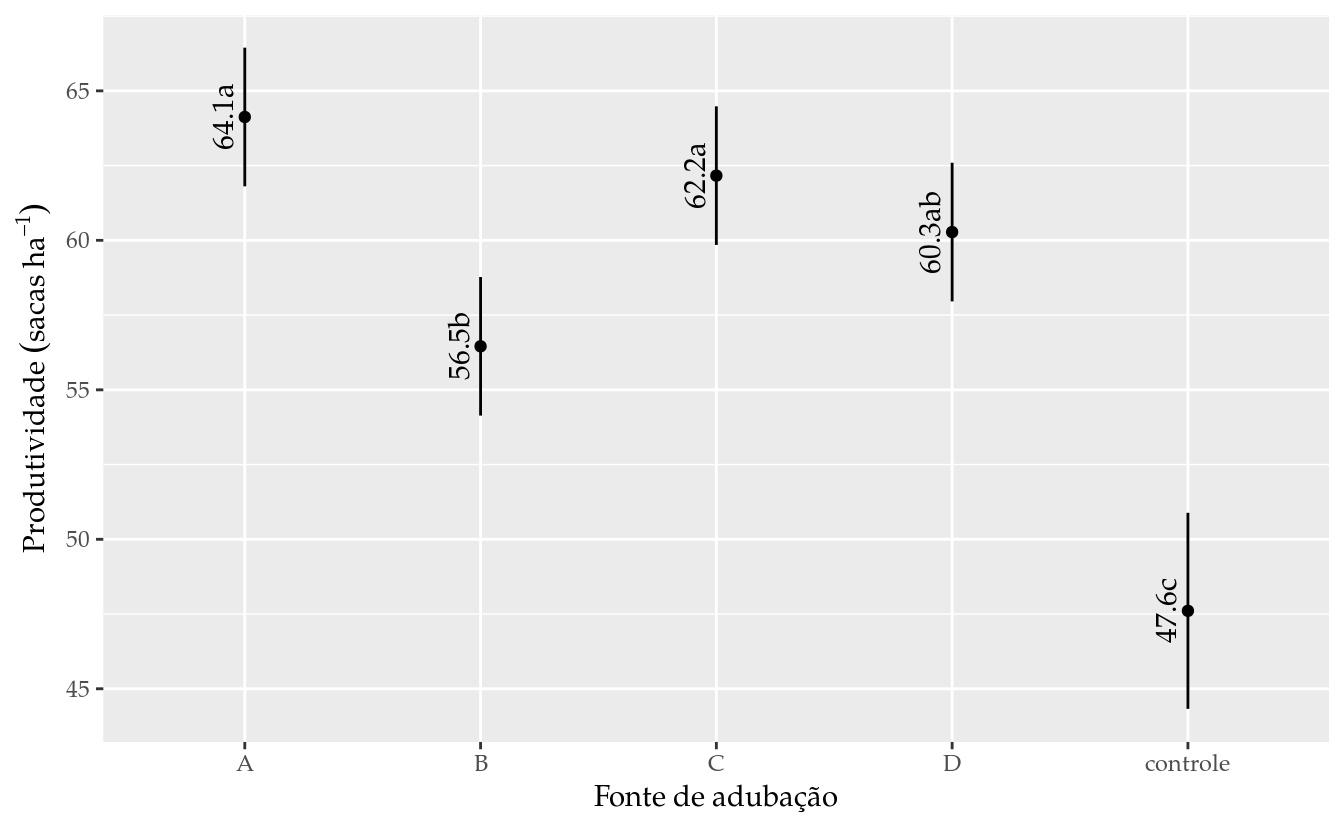
O gráfico de segmentos acima exibe as médias ajustadas acompanhadas o intervalo de confiança individual (95% de cobertura). Os intervalos de confiança para os níveis de fonte tem a mesma amplitude pois incidiram na mesma quantidade de unidades experimentais, 8. O intervalo de confiança do ponto adicional é mais amplo, porque teve 4 repetições. Qualquer fonte de adubação confere uma produtividade diferente da testemunha. Não existe diferença na produtividade média entre as fontes A, C e D.
O pacote emmeans, antigo lsmeans, é o mais recomendado para obter médias ajustadas. O pacote doBy contém a função LSmeans() (e LE_matrix()) que fazem um trabalho equivalente. Nesse tutorial eu preconizei obter as matrizes para que não houvesse mistério ou passe de mágica. No bloco abaixo é usada a função do emmeans().
Mesmo contanto com pacotes especializados para certos procedimentos, para situações irregulares, é preciso ter muito cuidado como o emprego das funções. Sempre que possível, faça testes com exemplos conhecidos, disponíveis em livros ou feitos por outras pessoas, antes de tornar público seus resultados. Você verá no código abaixo que os resultados da própria emmeans() não estão corretos. A função emite uma noticação sobre resultados enganosos (misleading). Isso porque a função foi desenvolvida para trabalhar em casos regulares onde todos os efeitos são estimáveis.
Abaixo declaramos o modelo usando fórmula usando contraste de Helmert (m0) e o contraste Treatment (default). Trata-se, portanto, do mesmo modelo sob diferentes parametrações para o efeito de fonte e modo. Já sabemos que esse modelo retorna estimativas NA mas vamos eliminar tais NA para fazer com a dimensão de coef(.) e vcov(.) sejam compatíveis. Com isso, podemos aplicar a emmeans().
# Modelo com contraste de Helmert para os fatores experimentais.
m0 <- lm(prod ~ bloc + fonte * modo,
data = tb,
contrasts = list(fonte = contr.helmert,
modo = contr.helmert))
# Modelo com contraste de zerar o primeiro nível (treatment = default).
m4 <- lm(formula(m0), data = tb)
# CAUTION: Elimina as entradas `NA` alterando o conteúdo do objeto.
m0$coefficients <- coef(m0)[!is.na(coef(m0))]
m4$coefficients <- coef(m4)[!is.na(coef(m4))]
# Estimativas diferem porque a parametrização é diferente.
cbind(coef(m0), coef(m4))## [,1] [,2]
## (Intercept) 57.59677778 62.5927778
## bloc2 1.41488889 1.4148889
## bloc3 0.37322222 0.3732222
## bloc4 0.32777778 0.3277778
## fonte1 -3.83531250 -6.3635000
## fonte2 0.62456250 -1.1107500
## fonte3 -0.15962500 -4.2482500
## fonte4 -2.62975000 -15.5150000
## modo1 0.56437500 2.0082500
## fonte1:modo1 -0.65356250 -2.6142500
## fonte2:modo1 -0.06577083 -1.7017500
## fonte3:modo1 0.27958333 0.7980000# As dimensões agora são compatíveis.
c(dim(vcov(m0)) == length(coef(m0)),
dim(vcov(m4)) == length(coef(m4)))## [1] TRUE TRUE TRUE TRUEPara salvar espaço, será mostrado os resultados apenas para o fator modo. O código para fonte está disponível no bloco a seguir.
# Médias ajustadas para efeito de fonte.
# NOTE: estimativas pontuais e erros padrões diferem!
(em_m0 <- emmeans::emmeans(m0, specs = "fonte"))
(em_m4 <- emmeans::emmeans(m4, specs = "fonte"))
# Junta todas as médias para comparação.
m <- aggregate(prod ~ fonte, data = tb, FUN = mean)
m$X_ls <- by(X_ls, pts$fonte, colMeans) %>%
do.call(what = rbind) %*% coef(m0)
m$em_m0 <- as.data.frame(em_m0)[, "emmean"]
m$em_m4 <- as.data.frame(em_m4)[, "emmean"]
m
# As matrizes construidas para obter as médias ajustadas.
by(X_ls, INDICES = pts$fonte, FUN = colMeans) %>%
do.call(what = rbind) %>% t() %>% MASS::fractions()
attr(em_m0, "linfct") %>% t() %>% MASS::fractions()
attr(em_m4, "linfct") %>% t() %>% MASS::fractions()# Médias ajustadas para efeito de fonte.
# NOTE: estimativas pontuais e erros padrões diferem!
(em_m0 <- emmeans::emmeans(m0, specs = "modo"))## NOTE: Results may be misleading due to involvement in interactions## modo emmean SE df lower.CL upper.CL
## lanco 57.6 0.786 24 55.9 59.2
## sulco 58.7 0.786 24 57.1 60.3
## controle 58.1 0.550 24 57.0 59.3
##
## Results are averaged over the levels of: bloc, fonte
## Confidence level used: 0.95(em_m4 <- emmeans::emmeans(m4, specs = "modo"))## NOTE: Results may be misleading due to involvement in interactions## modo emmean SE df lower.CL upper.CL
## lanco 57.7 0.711 24 56.2 59.1
## sulco 59.0 0.953 24 57.0 60.9
## controle 57.7 0.711 24 56.2 59.1
##
## Results are averaged over the levels of: bloc, fonte
## Confidence level used: 0.95# Junta todas as médias para comparação.
m <- aggregate(prod ~ modo, data = tb, FUN = mean)
m$X_ls <- by(X_ls, pts$modo, colMeans) %>%
do.call(what = rbind) %*% coef(m0)
m$em_m0 <- as.data.frame(em_m0)[, "emmean"]
m$em_m4 <- as.data.frame(em_m4)[, "emmean"]
m## modo prod X_ls em_m0 em_m4
## 1 lanco 60.19112 60.19112 57.56137 57.67425
## 2 sulco 61.31987 61.31987 58.69012 58.97890
## 3 controle 47.60675 47.60675 58.12575 57.67425# As matrizes construidas para obter as médias ajustadas.
by(X_ls, INDICES = pts$modo, FUN = colMeans) %>%
do.call(what = rbind) %>% t() %>% MASS::fractions()## lanco sulco controle
## (Intercept) 1 1 1
## bloc2 1/4 1/4 1/4
## bloc3 1/4 1/4 1/4
## bloc4 1/4 1/4 1/4
## fonte1 0 0 0
## fonte2 0 0 0
## fonte3 0 0 0
## fonte4 -1 -1 4
## modo1 -1 1 0
## fonte1:modo1 0 0 0
## fonte2:modo1 0 0 0
## fonte3:modo1 0 0 0attr(em_m0, "linfct") %>% t() %>% MASS::fractions()## [,1] [,2] [,3]
## (Intercept) 1 1 1
## bloc2 1/4 1/4 1/4
## bloc3 1/4 1/4 1/4
## bloc4 1/4 1/4 1/4
## fonte1 0 0 0
## fonte2 0 0 0
## fonte3 0 0 0
## fonte4 0 0 0
## modo1 -1 1 0
## fonte1:modo1 0 0 0
## fonte2:modo1 0 0 0
## fonte3:modo1 0 0 0attr(em_m4, "linfct") %>% t() %>% MASS::fractions()## [,1] [,2] [,3]
## (Intercept) 1 1 1
## bloc2 1/4 1/4 1/4
## bloc3 1/4 1/4 1/4
## bloc4 1/4 1/4 1/4
## fonteB 1/5 1/5 1/5
## fonteC 1/5 1/5 1/5
## fonteD 1/5 1/5 1/5
## fontecontrole 1/5 1/5 1/5
## modosulco 0 1 0
## fonteB:modosulco 0 1/5 0
## fonteC:modosulco 0 1/5 0
## fonteD:modosulco 0 1/5 0Esses resultados devem ser entendidos como um alerta: para a análise de casos irregulares tenha muito cuidado ao usar procedimentos que não foram preparados para isso. É relativamente fácil programar funções para casos regualares visto que conhecemos as condições, fazemos todos os tratamentos de exceção, etc. Mas as irregularidades pode acontecer de muitas formas sendo difíveis de cobrir, por isso é difícil de implementar, ou muitas vezes impossível de fazer.
No código abaixo eu mostro mais uma solução possível baseada na criação de uma variável para indicar se a observação pertence ao ponto adicional ou ponto fatorial. Com essa especificação as médias foram calculadas corretamente. A emmeans() notificou que encontrou a estrutura de aninhamento que de fato surgiu quando declaramos o modelo dessa nova maneira. Além do mais, os erros padrões das médias estão corretos também.
# Criando um fator para indicar a testemunha (s = sim).
tb$testemunha <- with(tb, factor(fonte == tail(levels(fonte), n = 1),
labels = c("n", "s")))
# Modelo com o termo para indicar a testemunha.
m5 <- lm(prod ~ bloc + testemunha + fonte * modo,
data = tb,
contrasts = list(fonte = contr.helmert,
modo = contr.helmert))
# m5$coefficients <- coef(m5)[!is.na(coef(m5))]
(em_m5 <- emmeans::emmeans(m5, specs = "fonte"))## NOTE: A nesting structure was detected in the fitted model:
## fonte %in% testemunha, modo %in% testemunha## fonte testemunha emmean SE df lower.CL upper.CL
## A n 64.1 1.12 24 61.8 66.4
## B n 56.5 1.12 24 54.1 58.8
## C n 62.2 1.12 24 59.8 64.5
## D n 60.3 1.12 24 58.0 62.6
## controle s 47.6 1.59 24 44.3 50.9
##
## Results are averaged over the levels of: bloc, modo
## Confidence level used: 0.95(em_m5 <- emmeans::emmeans(m5, specs = "modo"))## NOTE: A nesting structure was detected in the fitted model:
## fonte %in% testemunha, modo %in% testemunha## modo testemunha emmean SE df lower.CL upper.CL
## lanco n 60.2 0.794 24 58.6 61.8
## sulco n 61.3 0.794 24 59.7 63.0
## controle s 47.6 1.589 24 44.3 50.9
##
## Results are averaged over the levels of: bloc, fonte
## Confidence level used: 0.95# Erro-padrão da média com 4, 8 e 16 repetições para conferir.
sqrt(deviance(m5)/df.residual(m5) * (1/c(4, 8, 16)))## [1] 1.5889004 1.1235222 0.7944502Como não houve interação e nem diferença entre modos de aplicação, o modelo pode ser simplificado pelo abandono destes termos de efeito nulo. Com isso, desaparecem as dificuldades causadas pelo fatorial incompleto pois entramos em uma situação com um único fator de 5 níveis no qual o último é a testemunha. Ainda tem o detalhe do número de repetições ser é igual em todos os níveis, mas isso não é um complicador diante dos recursos que temos.
No bloco a seguir, a especificação mcp(. = "Tukey") corresponde a comparações múltiplas por contrastes de Tukey. Atenção: são contrastes de Tukey (comparações para a par) e não o emprego do teste de Tukey baseado na amplitude total studentizada. O Teste de Tukey para essas médias iria envolver aproximações do tipo média geométrica do número de repetições para ter uma diferença mínima significativa e assim testar a hipótese de diferença entre médias. A aproximação não funcionaria bem. Embora o teste de Tukey seja muito popular, a glht() faz comparações múltiplas por um método mais geral e que pode ser aplicado a uma maior classe de modelos.
m6 <- update(m5, prod ~ bloc + fonte)
# Testa se o conjunto de termos abandados tem efeito nulo.
anova(m6, m0)## Analysis of Variance Table
##
## Model 1: prod ~ bloc + fonte
## Model 2: prod ~ bloc + fonte * modo
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 28 267.10
## 2 24 242.36 4 24.739 0.6124 0.6577# Quadro de anova.
anova(m6)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## bloc 3 10.16 3.388 0.3552 0.7857
## fonte 4 871.24 217.811 22.8330 1.796e-08 ***
## Residuals 28 267.10 9.539
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Média ajustadas. Desse objeto é extraído a matriz para obter médias
# ajustadas.
lsm <- emmeans::emmeans(m6, specs = ~fonte)
lsm## fonte emmean SE df lower.CL upper.CL
## A 64.1 1.09 28 61.9 66.4
## B 56.5 1.09 28 54.2 58.7
## C 62.2 1.09 28 59.9 64.4
## D 60.3 1.09 28 58.0 62.5
## controle 47.6 1.54 28 44.4 50.8
##
## Results are averaged over the levels of: bloc
## Confidence level used: 0.95# Tabela com os níveis dos fatores e matriz para obter médias ajustadas.
pred <- attr(lsm, "grid")
X <- attr(lsm, "linfct")
rownames(X) <- pred$fonte
# Cria a matriz com os contrastes entre médias.
K <- all_pairwise(X, collapse = " - ")
# Avaliando cada contraste com correção pelo single-step.
comp <- summary(glht(m6, linfct = K))
comp##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: lm(formula = prod ~ bloc + fonte, data = tb, contrasts = list(fonte = contr.helmert,
## modo = contr.helmert))
##
## Linear Hypotheses:
## Estimate Std. Error t value Pr(>|t|)
## A - B == 0 7.671 1.544 4.967 < 0.001 ***
## A - C == 0 1.962 1.544 1.270 0.70874
## A - D == 0 3.849 1.544 2.493 0.11993
## A - controle == 0 16.519 1.891 8.734 < 0.001 ***
## B - C == 0 -5.709 1.544 -3.697 0.00759 **
## B - D == 0 -3.821 1.544 -2.475 0.12425
## B - controle == 0 8.849 1.891 4.678 < 0.001 ***
## C - D == 0 1.888 1.544 1.222 0.73659
## C - controle == 0 14.558 1.891 7.697 < 0.001 ***
## D - controle == 0 12.670 1.891 6.699 < 0.001 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- single-step method)# Usar `mcp(. = "Tukey")` e passar a matriz `K` é equivalente em modelos
# regulares. Na dúvida, passe a matriz que você construiu assegurando
# estar correta.
comp <- summary(glht(m6,
linfct = mcp(fonte = "Tukey",
covariate_average = TRUE)))
# Compara se as matrizes são as mesmas. Detalhe para o sinal.
all(comp$linfct == -K)## [1] TRUE# Representação com letras (compact letter display).
cld <- cld(comp, decreasing = TRUE)
# Intervalos de confiança para as médias.
ci <- confint(glht(m6, linfct = X), calpha = univariate_calpha())
ci <- as.data.frame(ci$confint)
# Junta todas as colunas em uma tabela só.
pred <- cbind(pred, ci)
pred$cld <- as.character(cld$mcletters$Letters)
pred$fonte <- factor(pred$fonte, levels(tb$fonte))
# NOTE: a função `wzRfun::apmc()` inclui todas essas etapas.# Os resultados pontuais são os mesmos. Os intervalares não.
aux <- bind_rows(list(m6 = pred %>% rename("fit" = "Estimate"),
m0 = results),
.id = "modelo")
aux## modelo fonte .wgt. fit lwr upr cld
## 1 m6 A 8 64.12587 61.88906 66.36269 a
## 2 m6 B 8 56.45525 54.21844 58.69206 b
## 3 m6 C 8 62.16425 59.92744 64.40106 a
## 4 m6 D 8 60.27662 58.03981 62.51344 ab
## 5 m6 controle 4 47.60675 44.44342 50.77008 c
## 6 m0 A NA 64.12587 61.80704 66.44471 a
## 7 m0 B NA 56.45525 54.13641 58.77409 b
## 8 m0 C NA 62.16425 59.84541 64.48309 a
## 9 m0 D NA 60.27662 57.95779 62.59546 ab
## 10 m0 controle NA 47.60675 44.32742 50.88608 c# Gráfico com estimativas, IC e texto.
pd <- position_dodge(0.1)
ggplot(data = aux,
mapping = aes(x = fonte, y = fit, color = modelo)) +
geom_point(position = pd) +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0, position = pd)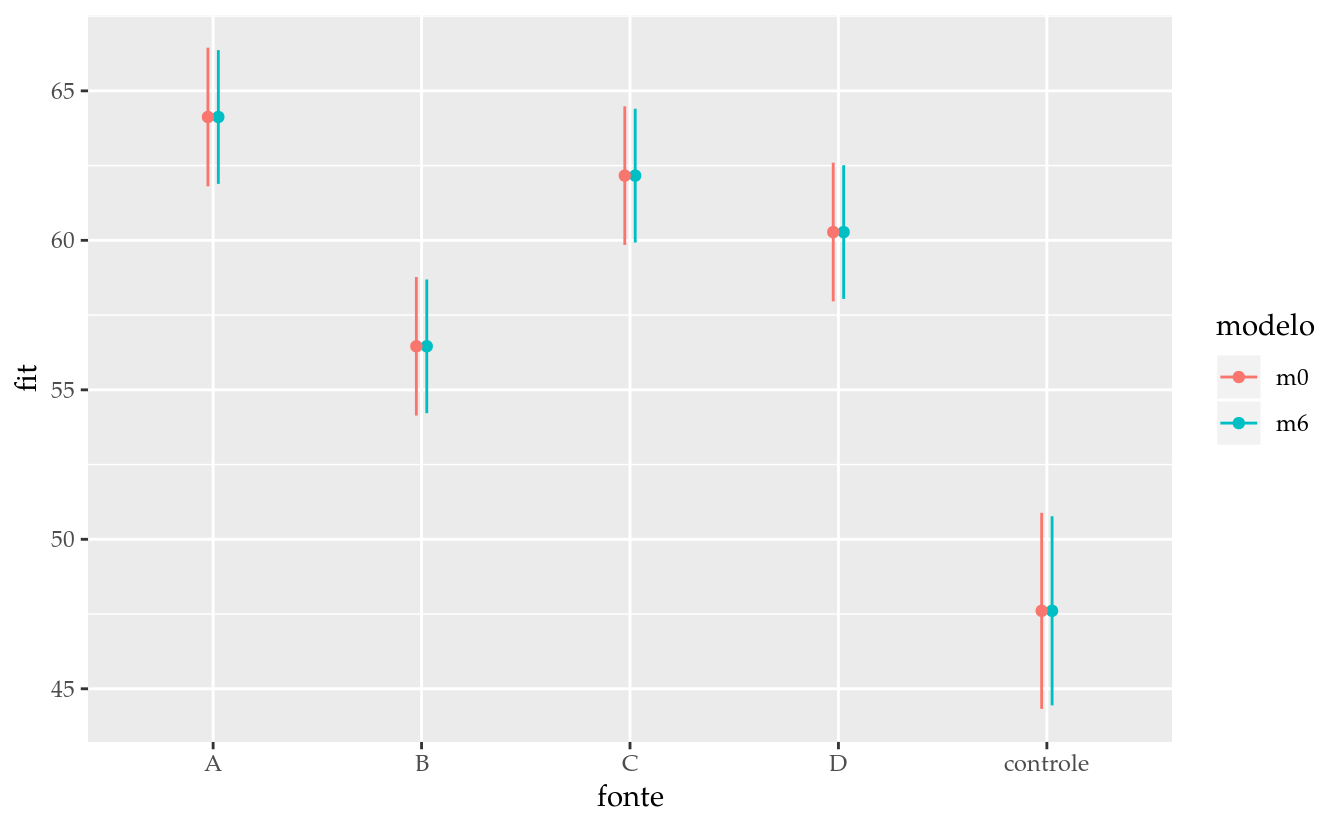
O gráfico de segmentos resume os resultados dos dois modelos. Não existe diferença nas estimativas pontuais, porém, os intervalos de confiança não são os mesmos. A diferença decorreu do abandono dos termos nulos que resultou um uma estimaitiva numérica diferente da variância do erro (\(\sigma^2\)) que é termo componente do intervalo de confiança. A diferença é meramente matemática e não estatística, já que os termos abandonados eram de efeito nulo. Mas é oportuno dizer que, embora ambos os modelos tenham levado às mesmas conclusões, em modelos mais gerais (GLM, sobreviência, de efeitos aleatórios) ou de estrutura irregular, as diferenças podem surgir nas estimativas pontuais além dos erros padrões. Salvo certas excessões como termos de blocagem, o mais seguro é abandonar os termos não significavos do modelo para conduzir as inferências terminais.
8.4 Considerações finais
WALMES FIXME TODO.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani