Capítulo 9 Fatorial duplo com fatores qualitativos
9.1 Motivação
# ATTENTION: Limpa espaço de trabalho.
rm(list = objects())
# Carrega pacotes.
library(car) # linearHypothesis()
library(multcomp) # glht()
library(emmeans) # emmeans()
library(tidyverse)
# Carrega funções.
source("mpaer_functions.R")9.2 Experimento fatorial duplo
TODO: modelo para o experimento, hipóteses, etc.
# https://github.com/pet-estatistica/labestData/blob/devel/data-raw/BanzattoQd5.2.4.txt
# help(BanzattoQd5.2.4, package = "labestData")
da <- labestData::BanzattoQd5.2.4
str(da)## 'data.frame': 24 obs. of 4 variables:
## $ recipie: Factor w/ 3 levels "SPP","SPG","Lam": 1 1 2 2 3 3 1 1 2 2 ...
## $ especie: Factor w/ 2 levels "E. citriodora",..: 1 2 1 2 1 2 1 2 1 2 ...
## $ rept : num 1 1 1 1 1 1 2 2 2 2 ...
## $ alt : num 26.2 24.8 25.7 19.6 22.8 19.8 26 24.6 26.3 21.1 ...da <- da %>%
mutate(especie = str_replace_all(especie, "[^[:alnum:]]", ""),
especie = factor(especie))
xtabs(~recipie + especie, data = da)## especie
## recipie Ecitriodora Egrandis
## SPP 4 4
## SPG 4 4
## Lam 4 4gg1 <- ggplot(data = da,
mapping = aes(x = especie, y = alt, color = recipie)) +
geom_point() +
stat_summary(mapping = aes(group = recipie),
geom = "line",
fun.y = "mean") +
scale_colour_brewer(palette = "Set2") +
theme(legend.position = "top")
gg2 <- ggplot(data = da,
mapping = aes(x = recipie, y = alt, color = especie)) +
geom_point() +
stat_summary(mapping = aes(group = especie),
geom = "line",
fun.y = "mean") +
scale_colour_brewer(palette = "Set1") +
theme(legend.position = "top")
gridExtra::grid.arrange(gg1, gg2, nrow = 1)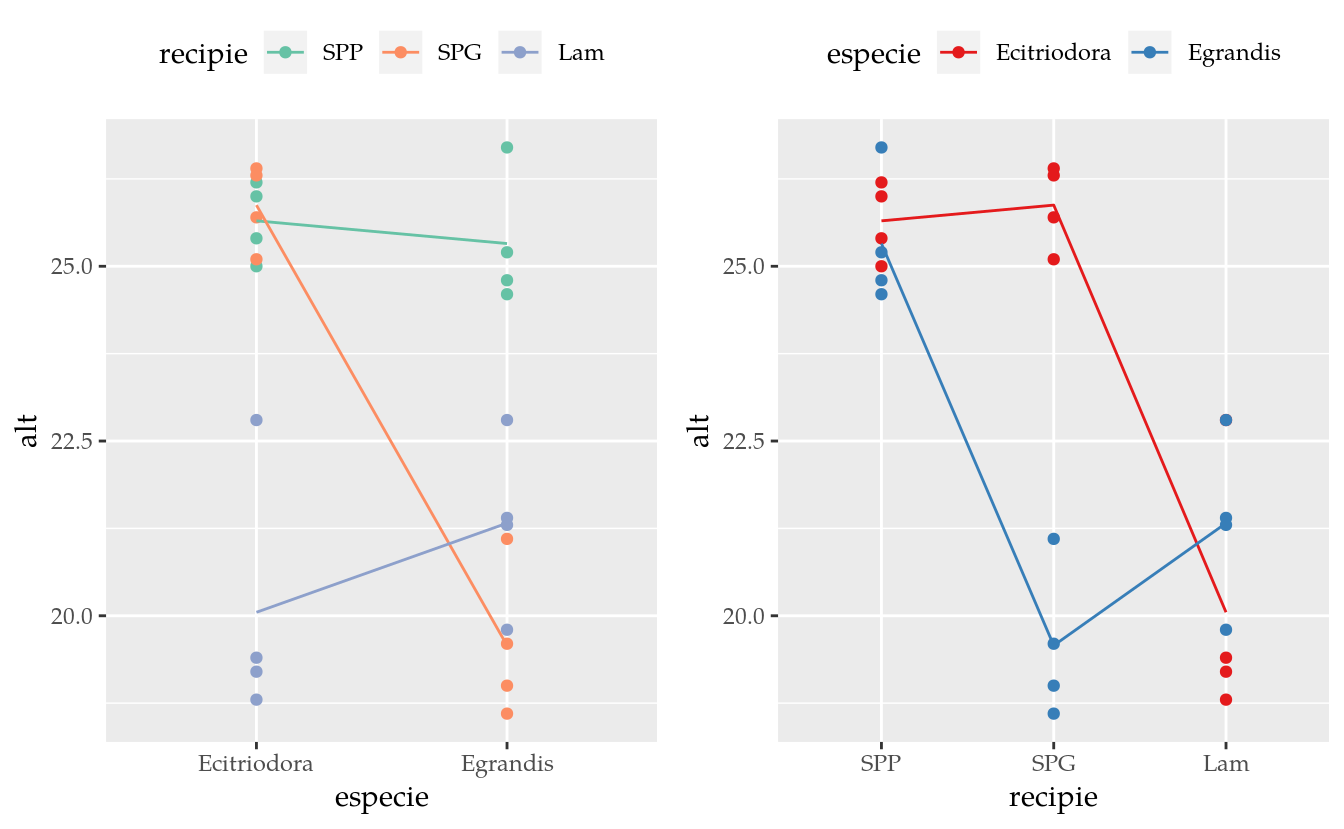
m0 <- lm(alt ~ recipie * especie, data = da)
par(mfrow = c(2, 2))
plot(m0)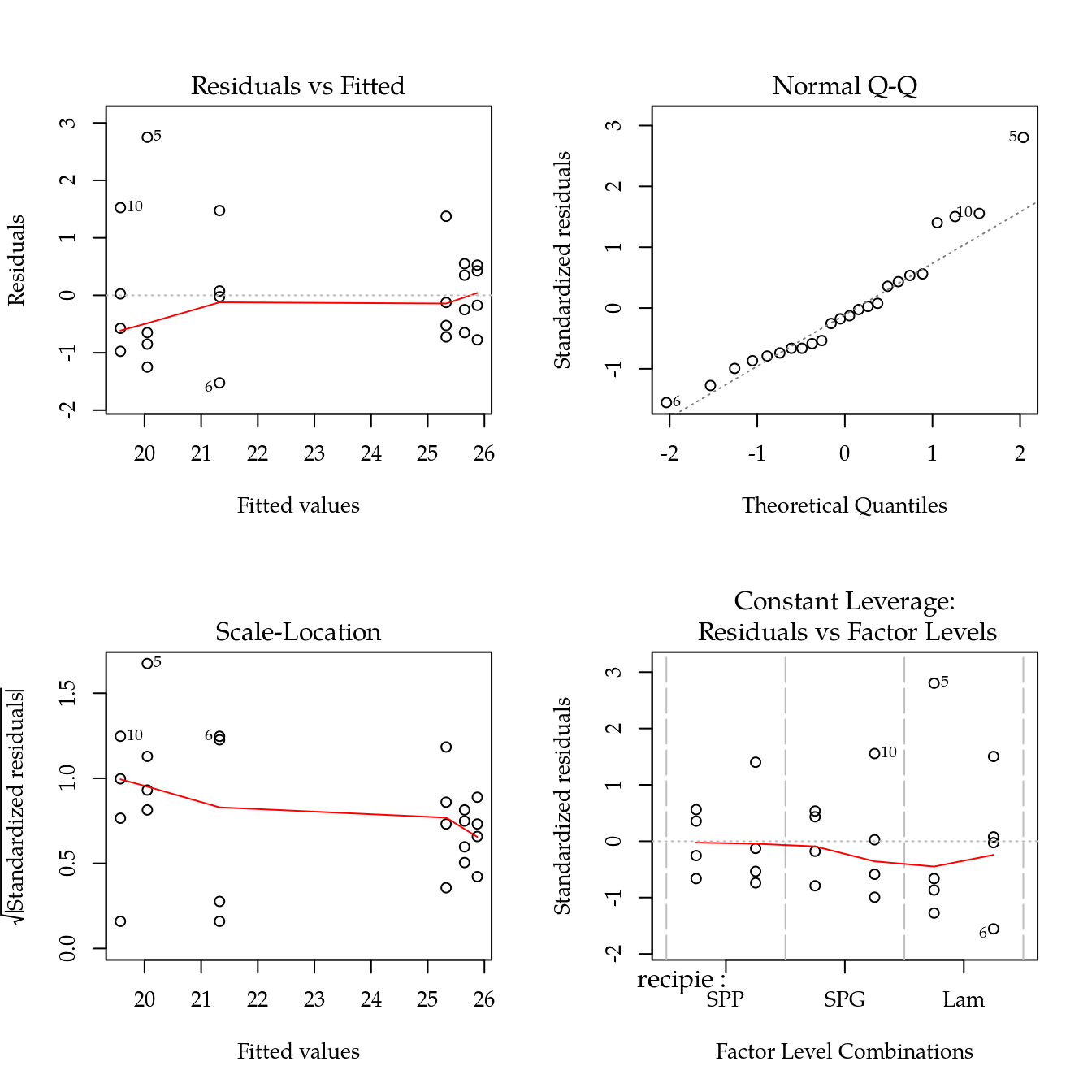
layout(1)
# anova(m0)
Anova(m0)## Anova Table (Type II tests)
##
## Response: alt
## Sum Sq Df F value Pr(>F)
## recipie 92.861 2 36.195 4.924e-07 ***
## especie 19.082 1 14.875 0.001155 **
## recipie:especie 63.761 2 24.853 6.635e-06 ***
## Residuals 23.090 18
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Decomposição de somas de quadrados de efeito de espécie dentro de
# recipiente.
m1 <- aov(alt ~ recipie/especie, data = m0$model)
m1$assign## [1] 0 1 1 2 2 2coef(m1)## (Intercept) recipieSPG
## 25.650 0.225
## recipieLam recipieSPP:especieEgrandis
## -5.600 -0.325
## recipieSPG:especieEgrandis recipieLam:especieEgrandis
## -6.300 1.275esp_in_rec <- list("esp@SSP" = 1,
"esp@SPG" = 2,
"esp@Lam" = 3)
summary(m1, split = list("recipie:especie" = esp_in_rec))## Df Sum Sq Mean Sq F value Pr(>F)
## recipie 2 92.86 46.43 36.195 4.92e-07 ***
## recipie:especie 3 82.84 27.61 21.527 3.51e-06 ***
## recipie:especie: esp@SSP 1 0.21 0.21 0.165 0.690
## recipie:especie: esp@SPG 1 79.38 79.38 61.881 3.11e-07 ***
## recipie:especie: esp@Lam 1 3.25 3.25 2.535 0.129
## Residuals 18 23.09 1.28
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1L <- cbind(0, 0, 0, diag(3))
linearHypothesis(m1, hypothesis.matrix = L[1, ], test = "F")## Linear hypothesis test
##
## Hypothesis:
## recipieSPP:especieEgrandis = 0
##
## Model 1: restricted model
## Model 2: alt ~ recipie/especie
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 19 23.301
## 2 18 23.090 1 0.21125 0.1647 0.6897linearHypothesis(m1, hypothesis.matrix = L[2, ], test = "F")## Linear hypothesis test
##
## Hypothesis:
## recipieSPG:especieEgrandis = 0
##
## Model 1: restricted model
## Model 2: alt ~ recipie/especie
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 19 102.47
## 2 18 23.09 1 79.38 61.881 3.112e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1linearHypothesis(m1, hypothesis.matrix = L[3, ], test = "F")## Linear hypothesis test
##
## Hypothesis:
## recipieLam:especieEgrandis = 0
##
## Model 1: restricted model
## Model 2: alt ~ recipie/especie
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 19 26.341
## 2 18 23.090 1 3.2513 2.5345 0.1288# Decomposição de somas de quadrados de efeito de recipiente dentro de
# espécie.
m1 <- aov(alt ~ especie/recipie, data = m0$model)
m1$assign## [1] 0 1 2 2 2 2coef(m1)## (Intercept) especieEgrandis
## 25.650 -0.325
## especieEcitriodora:recipieSPG especieEgrandis:recipieSPG
## 0.225 -5.750
## especieEcitriodora:recipieLam especieEgrandis:recipieLam
## -5.600 -4.000rec_in_esp <- list("rec@Citri" = c(1, 3),
"rec@Grand" = c(2, 4))
summary(m1, split = list("especie:recipie" = rec_in_esp))## Df Sum Sq Mean Sq F value Pr(>F)
## especie 1 19.08 19.08 14.88 0.00116 **
## especie:recipie 4 156.62 39.16 30.52 8.44e-08 ***
## especie:recipie: rec@Citri 2 87.12 43.56 33.96 7.78e-07 ***
## especie:recipie: rec@Grand 2 69.50 34.75 27.09 3.73e-06 ***
## Residuals 18 23.09 1.28
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1L <- cbind(0, 0, diag(4))
linearHypothesis(m1, hypothesis.matrix = L[c(1, 3), ], test = "F")## Linear hypothesis test
##
## Hypothesis:
## especieEcitriodora:recipieSPG = 0
## especieEcitriodora:recipieLam = 0
##
## Model 1: restricted model
## Model 2: alt ~ especie/recipie
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 20 110.21
## 2 18 23.09 2 87.122 33.958 7.776e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1linearHypothesis(m1, hypothesis.matrix = L[c(2, 4), ], test = "F")## Linear hypothesis test
##
## Hypothesis:
## especieEgrandis:recipieSPG = 0
## especieEgrandis:recipieLam = 0
##
## Model 1: restricted model
## Model 2: alt ~ especie/recipie
##
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 20 92.59
## 2 18 23.09 2 69.5 27.09 3.73e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# De volta ao modelo de efeitos cruzados.
summary(m0)##
## Call:
## lm(formula = alt ~ recipie * especie, data = da)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.5250 -0.6687 -0.1500 0.4500 2.7500
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 25.6500 0.5663 45.294 < 2e-16 ***
## recipieSPG 0.2250 0.8009 0.281 0.782
## recipieLam -5.6000 0.8009 -6.992 1.58e-06 ***
## especieEgrandis -0.3250 0.8009 -0.406 0.690
## recipieSPG:especieEgrandis -5.9750 1.1326 -5.275 5.13e-05 ***
## recipieLam:especieEgrandis 1.6000 1.1326 1.413 0.175
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.133 on 18 degrees of freedom
## Multiple R-squared: 0.8838, Adjusted R-squared: 0.8516
## F-statistic: 27.39 on 5 and 18 DF, p-value: 8.043e-08# Médias marginais ajustadas.
emm <- emmeans(m0, specs = ~especie | recipie)
emm## recipie = SPP:
## especie emmean SE df lower.CL upper.CL
## Ecitriodora 25.6 0.566 18 24.5 26.8
## Egrandis 25.3 0.566 18 24.1 26.5
##
## recipie = SPG:
## especie emmean SE df lower.CL upper.CL
## Ecitriodora 25.9 0.566 18 24.7 27.1
## Egrandis 19.6 0.566 18 18.4 20.8
##
## recipie = Lam:
## especie emmean SE df lower.CL upper.CL
## Ecitriodora 20.1 0.566 18 18.9 21.2
## Egrandis 21.3 0.566 18 20.1 22.5
##
## Confidence level used: 0.95# Contrastes entre médias por estrato.
contrast(emm, method = "tukey")## recipie = SPP:
## contrast estimate SE df t.ratio p.value
## Ecitriodora - Egrandis 0.325 0.801 18 0.406 0.6897
##
## recipie = SPG:
## contrast estimate SE df t.ratio p.value
## Ecitriodora - Egrandis 6.300 0.801 18 7.866 <.0001
##
## recipie = Lam:
## contrast estimate SE df t.ratio p.value
## Ecitriodora - Egrandis -1.275 0.801 18 -1.592 0.1288grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid %>%
unite(col = "x", -.wgt.) %>%
pull(x)
grp <- grid %>%
group_by(recipie)
results <- by(L,
group_indices(grp),
FUN = as.matrix) %>%
map(wzRfun::apmc, model = m0, focus = "especie") %>%
do.call(what = rbind) %>%
separate(col = "especie", into = head(names(grid), n = -1)) %>%
mutate(recipie = factor(recipie, levels = levels(da$recipie)),
especie = factor(especie, levels = levels(da$especie)))
results## especie recipie fit lwr upr cld
## 1 Ecitriodora SPP 25.650 24.46025 26.83975 a
## 2 Egrandis SPP 25.325 24.13525 26.51475 a
## 3 Ecitriodora SPG 25.875 24.68525 27.06475 a
## 4 Egrandis SPG 19.575 18.38525 20.76475 b
## 5 Ecitriodora Lam 20.050 18.86025 21.23975 a
## 6 Egrandis Lam 21.325 20.13525 22.51475 a# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = especie, y = fit)) +
facet_wrap(facets = ~recipie) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
angle = 90,
vjust = 0,
nudge_x = -0.05) +
labs(x = "Espécie", y = "Altura (cm)")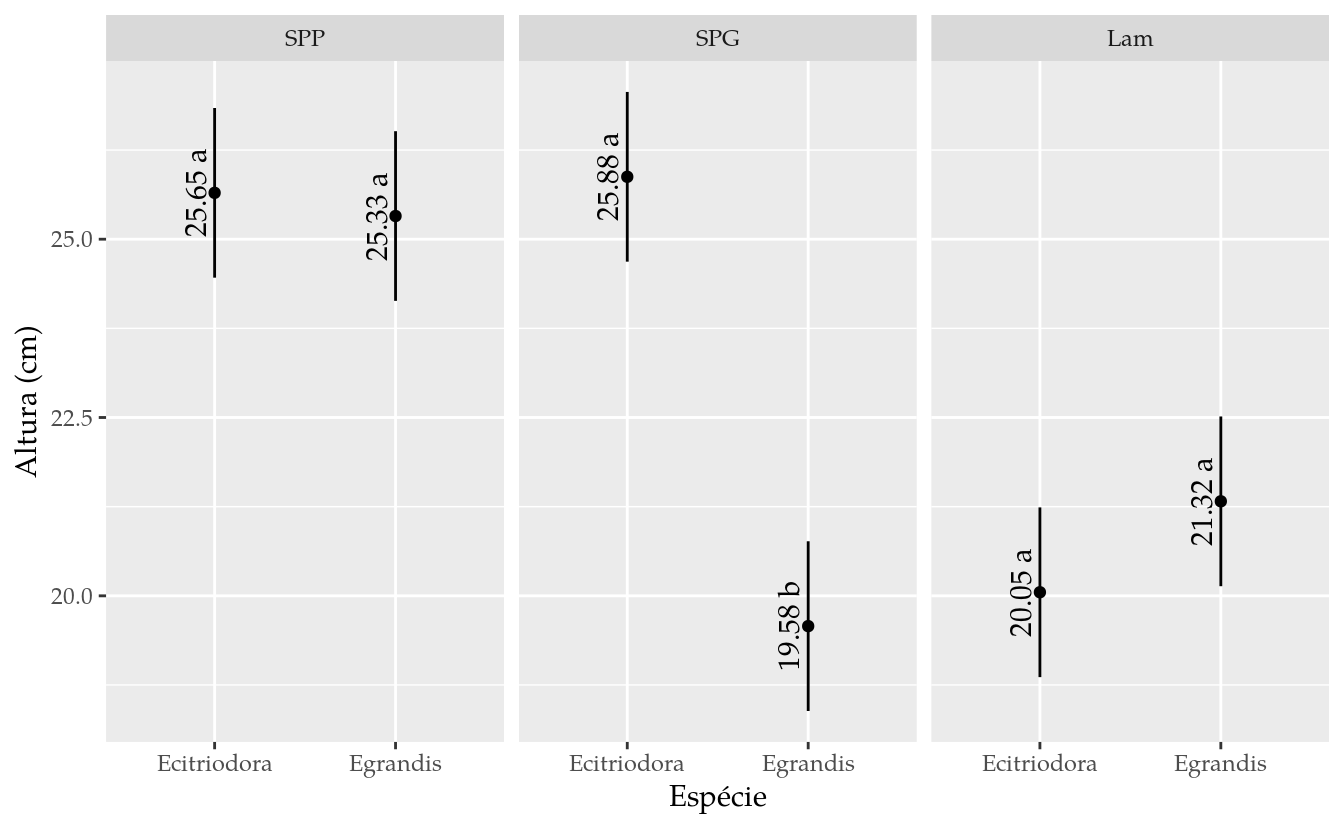
9.3 Transformação da variável resposta
url <- "http://leg.ufpr.br/~walmes/data/volume.txt"
da <- read_tsv(url, comment = "#")## Parsed with column specification:
## cols(
## gen = col_character(),
## dose = col_double(),
## rep = col_double(),
## volu = col_double()
## )attr(da, "spec") <- NULL
da <- da %>%
mutate(gen = factor(gen, levels = unique(gen)),
dose = factor(dose, levels = unique(dose)))
xtabs(~gen + dose, da)## dose
## gen 0 5 25
## ATF06B 3 3 3
## ATF40B 3 3 3
## ATF54B 3 3 3
## BR001B 3 3 3
## BR005B 3 3 3
## BR007B 3 3 3
## BR008B 3 3 3
## P9401 3 3 3
## SC283 3 3 3ggplot(data = da,
mapping = aes(x = dose, y = volu)) +
facet_wrap(facets = ~gen) +
geom_point() +
stat_summary(aes(group = 1), geom = "line", fun.y = "mean")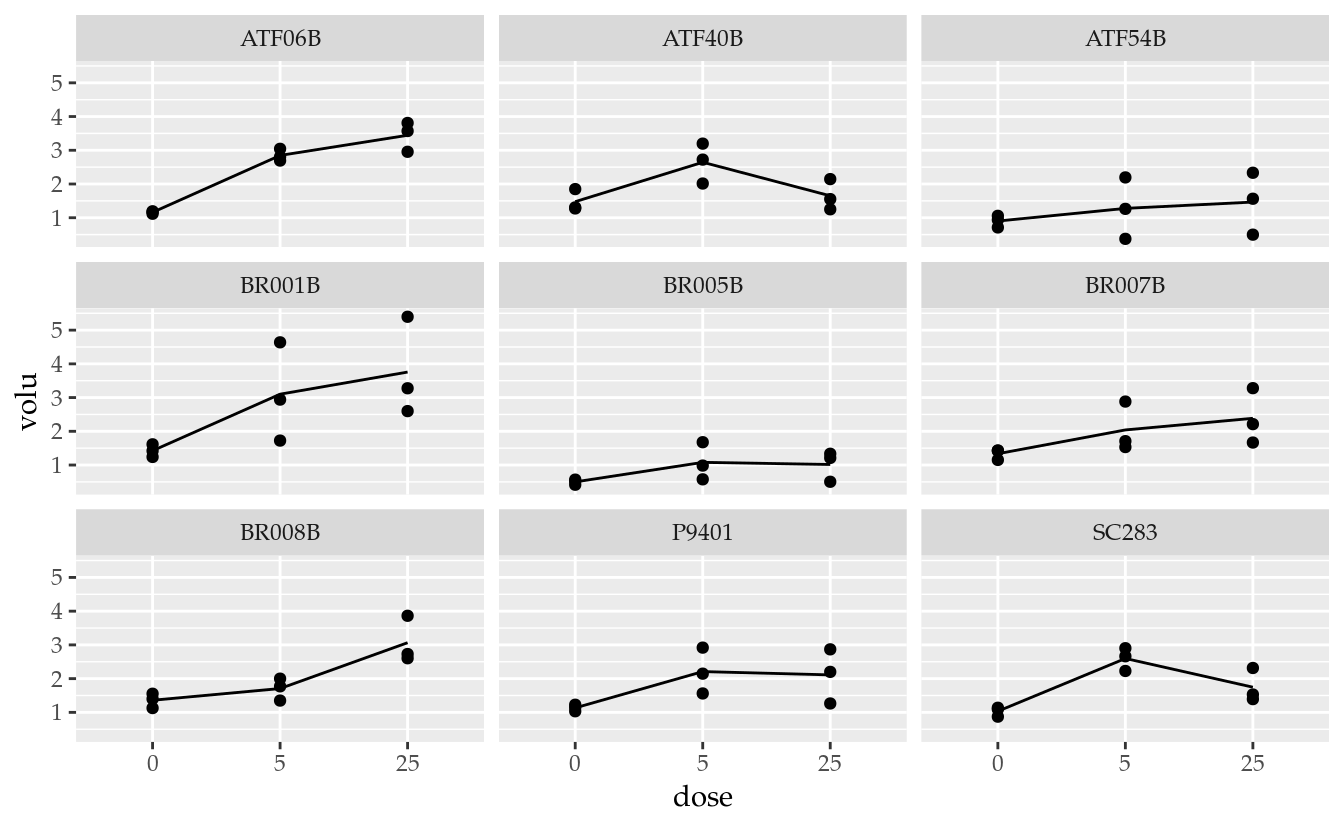
# Modelo para a resposta na escala original.
m0 <- aov(volu ~ gen * dose, data = da)
# Diagnóstico.
par(mfrow = c(2, 2))
plot(m0)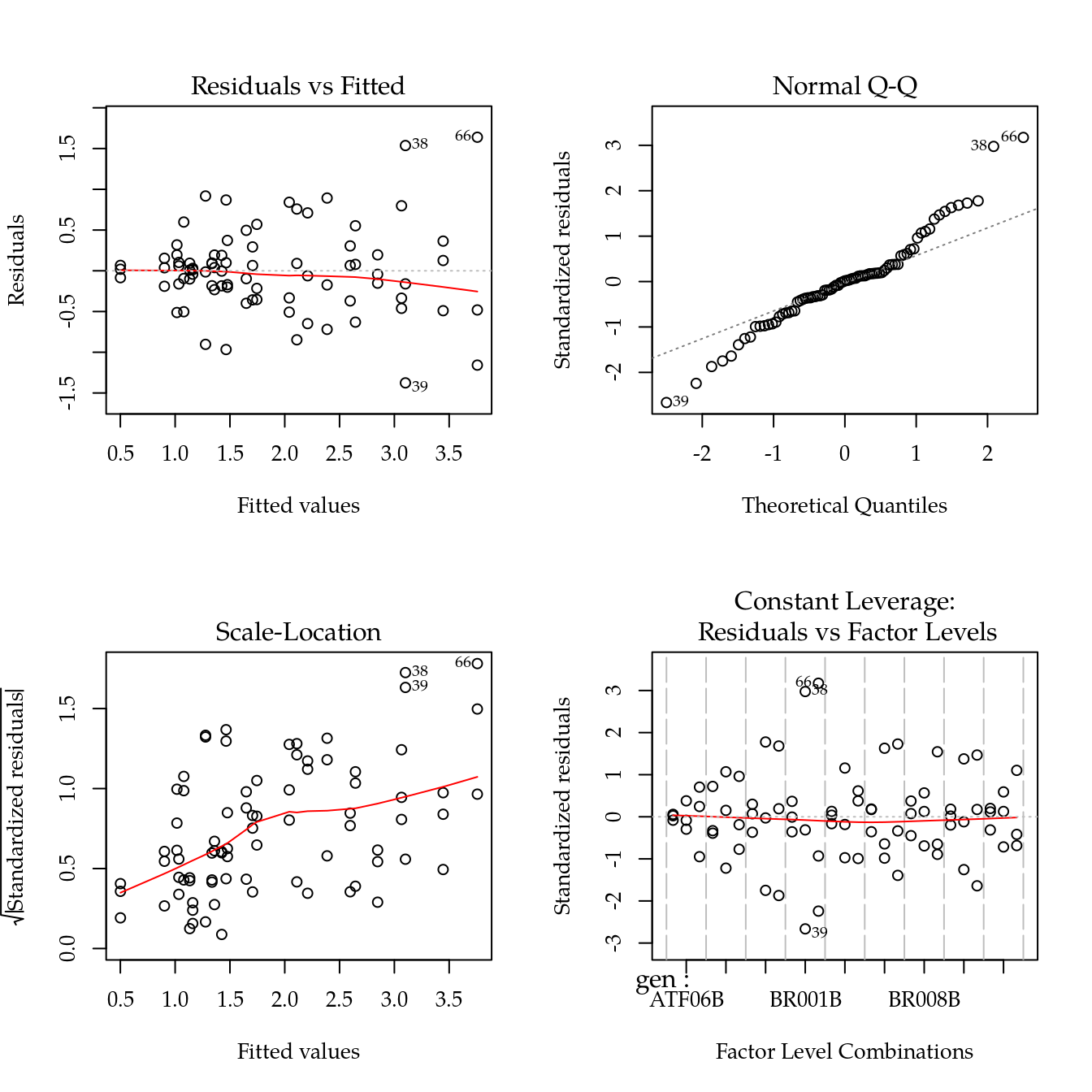
layout(1)
# NOTE: pronuncida relação positiva para média-dispersão!# Busca por transformação da família Box-Cox.
MASS::boxcox(m0)
abline(v = 1/3, col = 2)
# Foi indicada a transformação raíz cúbica. Raíz cúbica de voluma (cm^3)
# está em cm^1. Interessante, certo?# Modelo com a variável transformada.
da$volu_trans <- da$volu^(1/3)
m0 <- aov(volu_trans ~ gen * dose, data = da)
# Diagnóstico.
par(mfrow = c(2, 2))
plot(m0)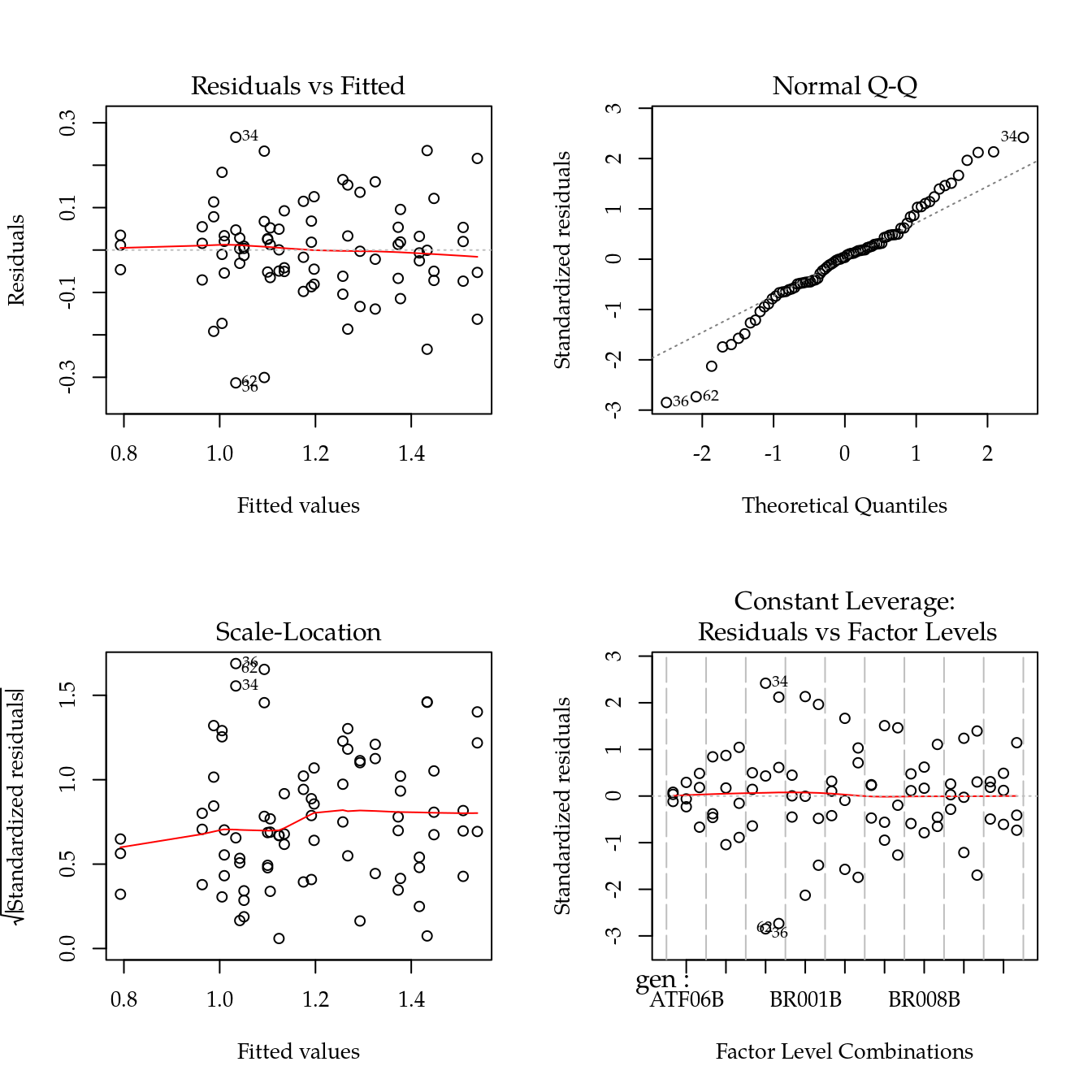
layout(1)
# Quadro de análise de variância.
Anova(m0)## Anova Table (Type II tests)
##
## Response: volu_trans
## Sum Sq Df F value Pr(>F)
## gen 1.34311 8 9.2631 6.310e-08 ***
## dose 1.01867 2 28.1020 4.318e-09 ***
## gen:dose 0.40328 16 1.3906 0.1817
## Residuals 0.97872 54
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Abandona termo de interação que não é relevante.
m0 <- update(m0, . ~ gen + dose)
# Médias marginais ajustadas para níveis de dose.
emm <- emmeans(m0, specs = ~dose)
emm## dose emmean SE df lower.CL upper.CL
## 0 1.04 0.027 70 0.982 1.09
## 5 1.26 0.027 70 1.211 1.32
## 25 1.28 0.027 70 1.228 1.34
##
## Results are averaged over the levels of: gen
## Confidence level used: 0.95# Contrastes de Tukey.
contrast(emm, method = "tukey")## contrast estimate SE df t.ratio p.value
## 0 - 5 -0.2286 0.0382 70 -5.978 <.0001
## 0 - 25 -0.2462 0.0382 70 -6.438 <.0001
## 5 - 25 -0.0176 0.0382 70 -0.460 0.8902
##
## Results are averaged over the levels of: gen
## P value adjustment: tukey method for comparing a family of 3 estimates# Médias marginais ajustadas para níveis de genótipo.
emm <- emmeans(m0, specs = ~gen)
emm## gen emmean SE df lower.CL upper.CL
## ATF06B 1.325 0.0468 70 1.232 1.42
## ATF40B 1.229 0.0468 70 1.136 1.32
## ATF54B 1.030 0.0468 70 0.937 1.12
## BR001B 1.365 0.0468 70 1.272 1.46
## BR005B 0.928 0.0468 70 0.835 1.02
## BR007B 1.227 0.0468 70 1.134 1.32
## BR008B 1.248 0.0468 70 1.155 1.34
## P9401 1.201 0.0468 70 1.107 1.29
## SC283 1.193 0.0468 70 1.100 1.29
##
## Results are averaged over the levels of: dose
## Confidence level used: 0.95# Objetos para obter as comparações múltiplas de genótipo.
grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid[[1]]
# Constrastes de Tukey com correção da multiplicidade pelo método false
# discovery rate.
results <- wzRfun::apmc(L, model = m0, focus = "gen", test = "fdr")
# Aplica a transformação inversa nas estimativas.
results <- results %>%
mutate_at(c("fit", "lwr", "upr"), function(x) x^3) %>%
# mutate_at(c("fit", "lwr", "upr"), "^", 3) %>%
# mutate_at(c("fit", "lwr", "upr"), power(3)$linkfun) %>%
arrange(desc(fit))
results## gen fit lwr upr cld
## 1 BR001B 2.5434283 2.0561841 3.102139 a
## 2 ATF06B 2.3281219 1.8697398 2.855894 ab
## 3 BR008B 1.9444976 1.5397762 2.414567 ab
## 4 ATF40B 1.8567837 1.4647931 2.313124 ab
## 5 BR007B 1.8490295 1.4581736 2.304145 ab
## 6 P9401 1.7314018 1.3579496 2.167722 b
## 7 SC283 1.6986572 1.3301163 2.129667 b
## 8 ATF54B 1.0930788 0.8218622 1.418227 c
## 9 BR005B 0.8002142 0.5821583 1.066877 c# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = reorder(gen, fit), y = fit)) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
size = 3,
angle = 90,
vjust = 0,
nudge_x = -0.1) +
expand_limits(x = c(0, NA)) +
labs(x = "Genótipos", y = "Volume de raízes")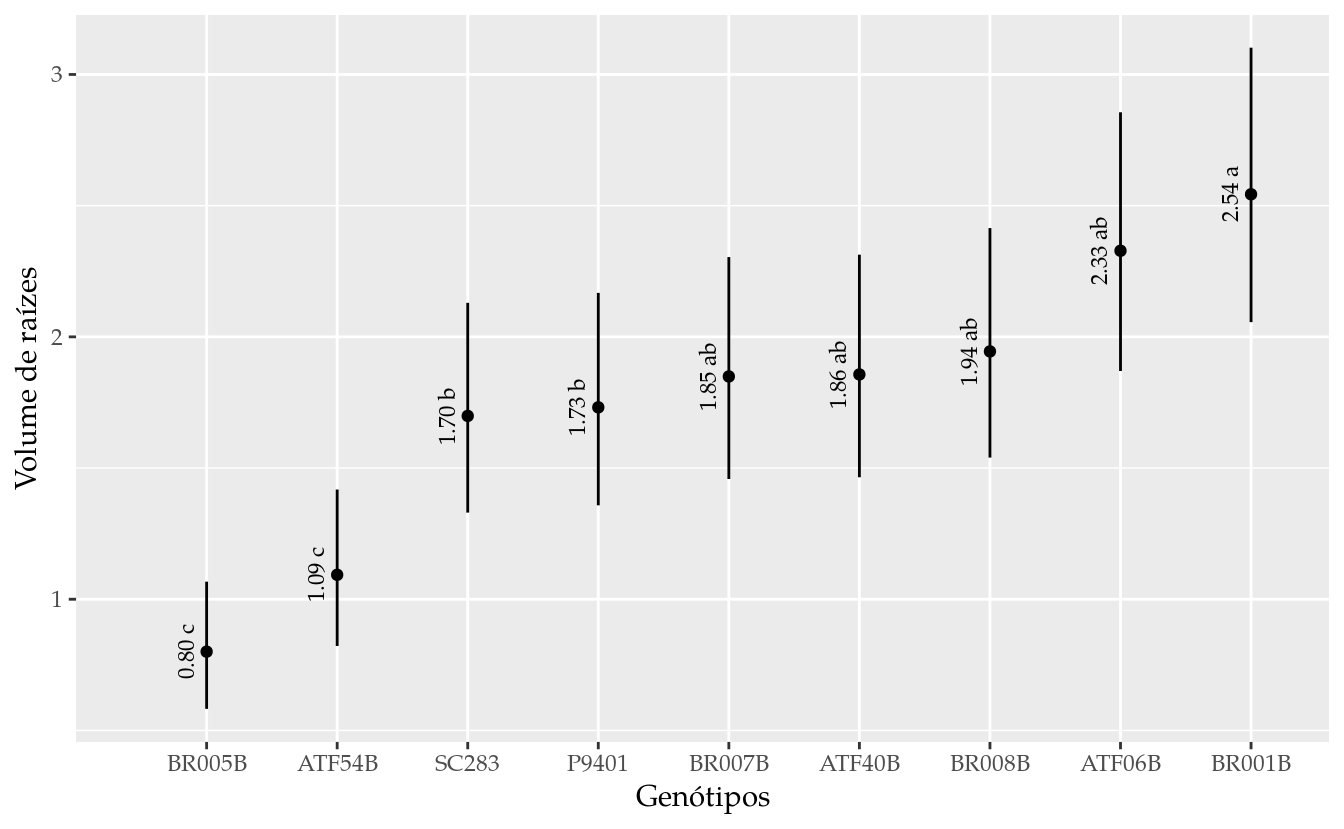
9.4 Resposta de contagem
url <- "http://leg.ufpr.br/~walmes/data/desfolha_algodao.txt"
da <- read_tsv(url, comment = "#")## Parsed with column specification:
## cols(
## fase = col_character(),
## desf = col_double(),
## planta = col_double(),
## bloco = col_double(),
## nestrrep = col_double(),
## ncapu = col_double(),
## alt = col_double(),
## nnos = col_double(),
## pesocap = col_double()
## )attr(da, "spec") <- NULL
# ATTENTION: esse experimento não foi feito em bloco.
da <- da %>%
rename("rept" = "bloco") %>%
mutate(fase = factor(fase, levels = unique(fase)),
desf = factor(desf, levels = unique(desf))) %>%
group_by(fase, desf, rept) %>%
summarise_at("ncapu", "sum") %>%
ungroup()
str(da)## Classes 'tbl_df', 'tbl' and 'data.frame': 125 obs. of 4 variables:
## $ fase : Factor w/ 5 levels "veget","botflor",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ desf : Factor w/ 5 levels "0","25","50",..: 1 1 1 1 1 2 2 2 2 2 ...
## $ rept : num 1 2 3 4 5 1 2 3 4 5 ...
## $ ncapu: num 10 9 8 8 10 11 9 10 10 10 ...xtabs(~fase + desf, da)## desf
## fase 0 25 50 75 100
## veget 5 5 5 5 5
## botflor 5 5 5 5 5
## floresc 5 5 5 5 5
## maça 5 5 5 5 5
## capulho 5 5 5 5 5# Cria um deslocamento sistemático nos pontos para evitar pontos
# sobrepostos.
da <- da %>%
group_by(fase, desf, ncapu) %>%
mutate(desf_shift = {
# as.numeric(as.character(desf)) +
as.integer(desf) +
0.2 * scale(seq(n()), scale = FALSE)
})
# Análise exploratória.
ggplot(data = da,
mapping = aes(x = desf_shift, y = ncapu)) +
facet_wrap(facets = ~fase) +
geom_point() +
stat_summary(aes(x = as.integer(desf), group = 1),
geom = "line",
fun.y = "mean") +
scale_x_continuous(breaks = seq(nlevels(da$desf)),
labels = levels(da$desf))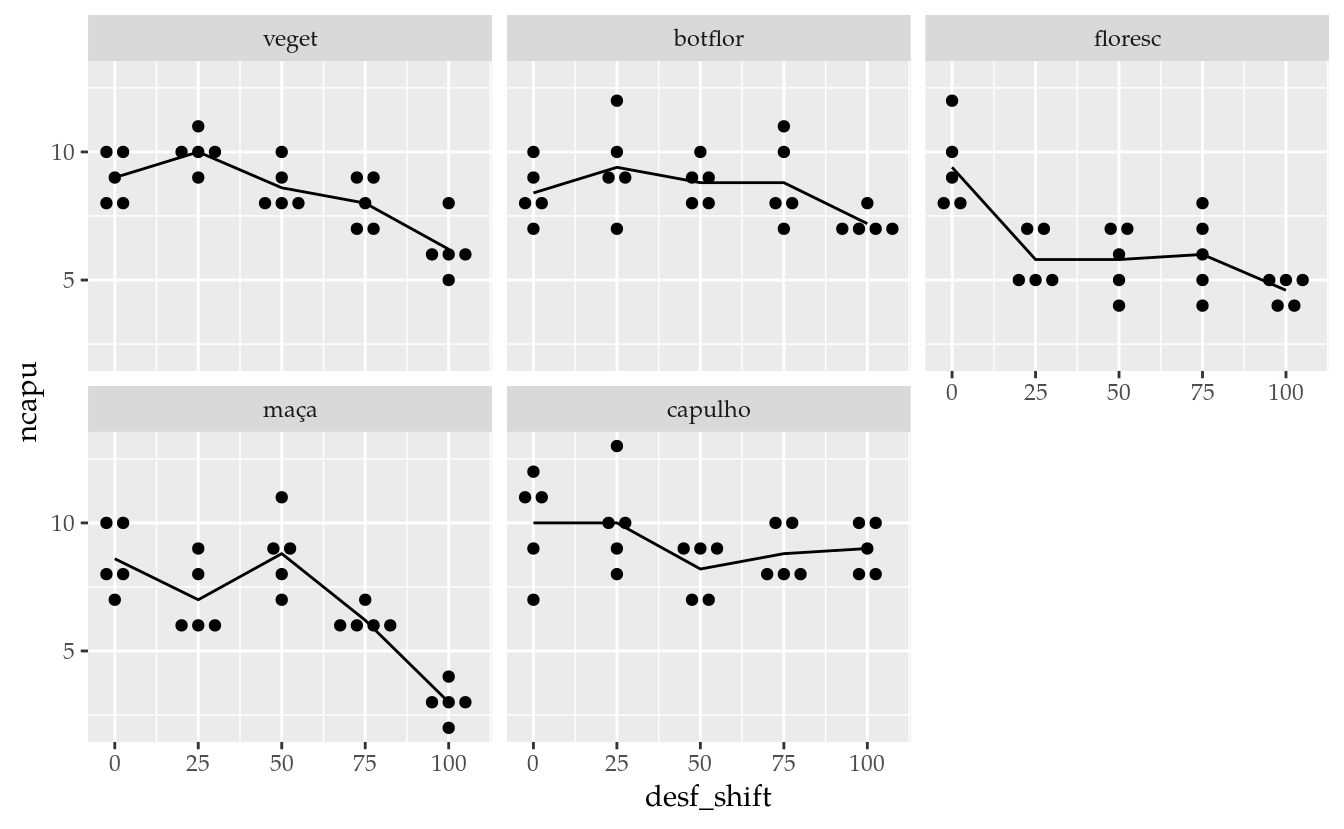
# Modelo linear generalizado com especificação de média e variância.
m0 <- glm(ncapu ~ fase * desf,
data = da,
family = quasipoisson)
# Diagnóstico.
par(mfrow = c(2, 2))
plot(m0)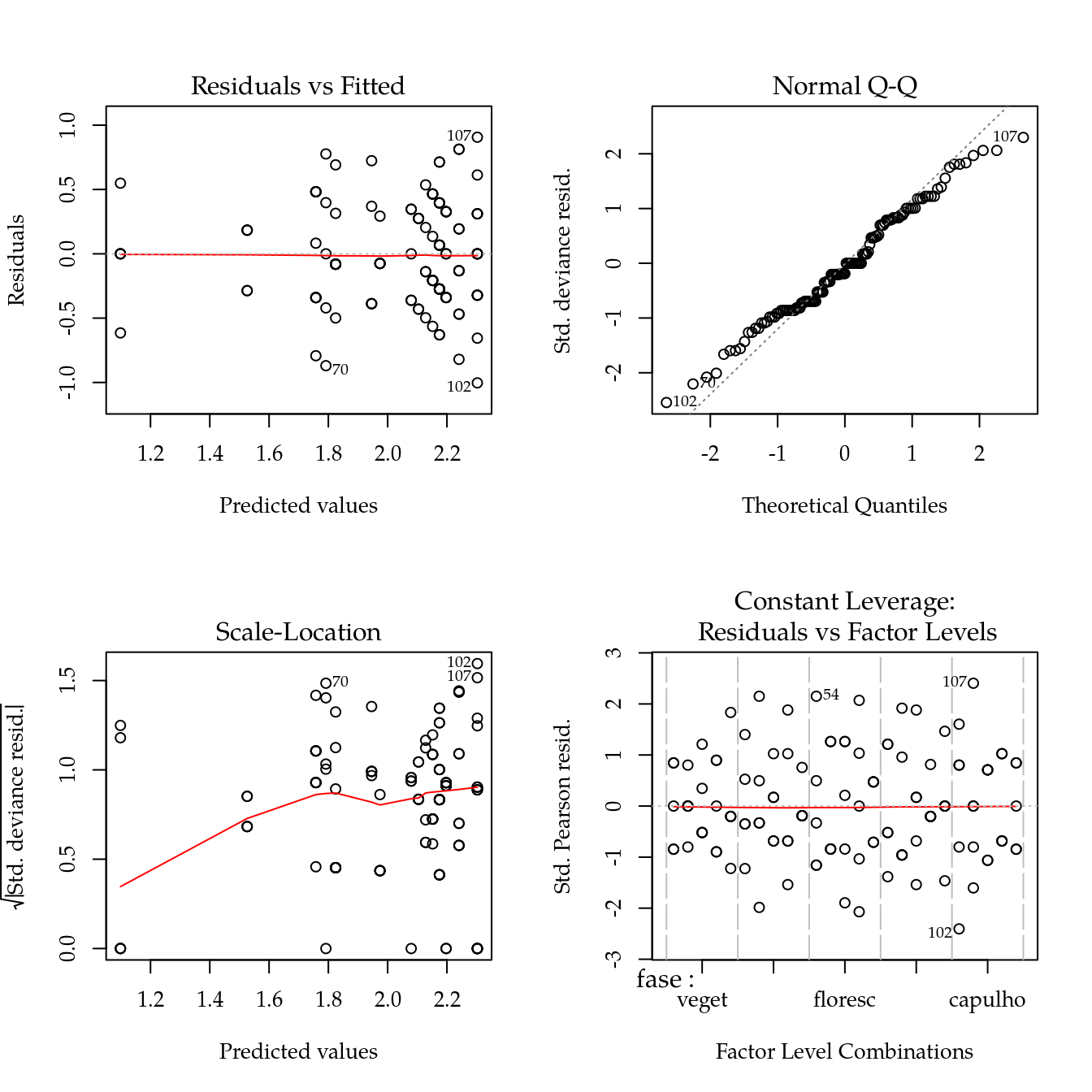
layout(1)
# anova(m0, test = "F")
# drop1(m0, scope = . ~ ., test = "F")
Anova(m0)## Analysis of Deviance Table (Type II tests)
##
## Response: ncapu
## LR Chisq Df Pr(>Chisq)
## fase 102.662 4 < 2.2e-16 ***
## desf 92.150 4 < 2.2e-16 ***
## fase:desf 93.765 16 5.039e-13 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Modelo para testar hipóteses do efeito de desfolha em cada fase.
m1 <- update(m0, . ~ fase/desf)
asgn <- attr(model.matrix(m1), "assign")
cbind(estimativa = round(coef(m1), digits = 4), termo = asgn)## estimativa termo
## (Intercept) 2.1972 0
## fasebotflor -0.0690 1
## fasefloresc 0.0435 1
## fasemaça -0.0455 1
## fasecapulho 0.1054 1
## faseveget:desf25 0.1054 2
## fasebotflor:desf25 0.1125 2
## fasefloresc:desf25 -0.4829 2
## fasemaça:desf25 -0.2059 2
## fasecapulho:desf25 0.0000 2
## faseveget:desf50 -0.0455 2
## fasebotflor:desf50 0.0465 2
## fasefloresc:desf50 -0.4829 2
## fasemaça:desf50 0.0230 2
## fasecapulho:desf50 -0.1985 2
## faseveget:desf75 -0.1178 2
## fasebotflor:desf75 0.0465 2
## fasefloresc:desf75 -0.4490 2
## fasemaça:desf75 -0.3272 2
## fasecapulho:desf75 -0.1278 2
## faseveget:desf100 -0.3727 2
## fasebotflor:desf100 -0.1542 2
## fasefloresc:desf100 -0.7147 2
## fasemaça:desf100 -1.0531 2
## fasecapulho:desf100 -0.1054 2# Para testar hipóteses do efeito de desfolha dentro de cada fase.
desf_in_fase <-
names(coef(m1)[asgn == 2]) %>%
str_replace("^fase", "") %>%
str_split(pattern = ":", simplify = TRUE) %>%
as.data.frame(stringsAsFactors = FALSE) %>%
mutate(i = 1:n()) %>%
split(x = .$i, f = .[["V1"]])
desf_in_fase <- desf_in_fase[levels(da$fase)]
desf_in_fase## $veget
## [1] 1 6 11 16
##
## $botflor
## [1] 2 7 12 17
##
## $floresc
## [1] 3 8 13 18
##
## $maça
## [1] 4 9 14 19
##
## $capulho
## [1] 5 10 15 20# Testes de hipótese por Wald.
cftest(model = m1,
parm = sum(asgn < 2) + desf_in_fase$veget,
test = Ftest())##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## faseveget:desf25 == 0 0.10536
## faseveget:desf50 == 0 -0.04546
## faseveget:desf75 == 0 -0.11778
## faseveget:desf100 == 0 -0.37268
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 6.046 4 100 0.0002125p <- length(asgn) # Número total de parâmetros.
u <- sum(asgn == 2) # Número de parâmetros na interação.
L <- cbind(matrix(0, nrow = u, ncol = p - u), diag(u))
# Testes de hipótese por Wald.
linearHypothesis(m1,
hypothesis.matrix = L[desf_in_fase$veget, ],
test = "F")## Linear hypothesis test
##
## Hypothesis:
## faseveget:desf25 = 0
## faseveget:desf50 = 0
## faseveget:desf75 = 0
## faseveget:desf100 = 0
##
## Model 1: restricted model
## Model 2: ncapu ~ fase + fase:desf
##
## Res.Df Df F Pr(>F)
## 1 104
## 2 100 4 6.0463 0.0002125 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Soma uma constante a todos os vetores componentes da lista.
ef <- lapply(desf_in_fase,
FUN = "+",
sum(asgn < 2))
# Serializa a aplicação da `cftest()` e extração das estatísticas.
lapply(ef,
FUN = cftest,
model = m1,
test = Ftest()) %>%
lapply(extract_cftest) %>%
do.call(what = rbind) %>%
round(digits = 5)## df1 df2 fstat pvalue
## veget 4 100 6.04632 0.00021
## botflor 4 100 2.02103 0.09720
## floresc 4 100 12.97554 0.00000
## maça 4 100 19.69574 0.00000
## capulho 4 100 1.72940 0.14942# Médias marginais ajustadas (na escala do preditor linear).
emm <- emmeans(m0, specs = ~desf | fase)
emm## fase = veget:
## desf emmean SE df asymp.LCL asymp.UCL
## 0 2.20 0.0657 Inf 2.068 2.33
## 25 2.30 0.0624 Inf 2.180 2.42
## 50 2.15 0.0672 Inf 2.020 2.28
## 75 2.08 0.0697 Inf 1.943 2.22
## 100 1.82 0.0792 Inf 1.669 1.98
##
## fase = botflor:
## desf emmean SE df asymp.LCL asymp.UCL
## 0 2.13 0.0680 Inf 1.995 2.26
## 25 2.24 0.0643 Inf 2.115 2.37
## 50 2.17 0.0665 Inf 2.044 2.31
## 75 2.17 0.0665 Inf 2.044 2.31
## 100 1.97 0.0735 Inf 1.830 2.12
##
## fase = floresc:
## desf emmean SE df asymp.LCL asymp.UCL
## 0 2.24 0.0643 Inf 2.115 2.37
## 25 1.76 0.0819 Inf 1.597 1.92
## 50 1.76 0.0819 Inf 1.597 1.92
## 75 1.79 0.0805 Inf 1.634 1.95
## 100 1.53 0.0919 Inf 1.346 1.71
##
## fase = maça:
## desf emmean SE df asymp.LCL asymp.UCL
## 0 2.15 0.0672 Inf 2.020 2.28
## 25 1.95 0.0745 Inf 1.800 2.09
## 50 2.17 0.0665 Inf 2.044 2.31
## 75 1.82 0.0792 Inf 1.669 1.98
## 100 1.10 0.1138 Inf 0.875 1.32
##
## fase = capulho:
## desf emmean SE df asymp.LCL asymp.UCL
## 0 2.30 0.0624 Inf 2.180 2.42
## 25 2.30 0.0624 Inf 2.180 2.42
## 50 2.10 0.0689 Inf 1.969 2.24
## 75 2.17 0.0665 Inf 2.044 2.31
## 100 2.20 0.0657 Inf 2.068 2.33
##
## Results are given on the log (not the response) scale.
## Confidence level used: 0.95# Contrates de Tukey.
# contrast(emm, method = "tukey")
# Extração da matriz para fazer as comparações múltiplas.
grid <- attr(emm, "grid")
L <- attr(emm, "linfct")
rownames(L) <- grid %>%
unite(col = "x", -.wgt.) %>%
pull(x)
# Cria uma estrutura agrupada para usar as chaves.
grp <- grid %>%
group_by(fase)
# Resultados da aplicação dos contrastes de Tukey para níveis de
# desfolha dentro de cada nível de fase. Correção da multiplicidade
# feita com o método single-step. Estimativas levadas para a escala da
# resposta com a aplicação da inversa da função de ligação.
results_tk <- by(L,
group_indices(grp),
FUN = as.matrix) %>%
map(wzRfun::apmc, model = m0, focus = "desf") %>%
do.call(what = rbind) %>%
separate(col = "desf", into = head(names(grid), n = -1)) %>%
mutate(desf = factor(desf, levels = levels(da$desf)),
fase = factor(fase, levels = levels(da$fase))) %>%
mutate_at(c("fit", "lwr", "upr"), m0$family$linkinv)
results_tk## desf fase fit lwr upr cld
## 1 0 veget 9.0 7.912157 10.237411 a
## 2 25 veget 10.0 8.849596 11.299951 a
## 3 50 veget 8.6 7.538145 9.811432 a
## 4 75 veget 8.0 6.978283 9.171310 ab
## 5 100 veget 6.2 5.308653 7.241008 b
## 6 0 botflor 8.4 7.351365 9.598217 ab
## 7 25 botflor 9.4 8.286737 10.662822 a
## 8 50 botflor 8.8 7.725078 10.024495 ab
## 9 75 botflor 8.8 7.725078 10.024495 ab
## 10 100 botflor 7.2 6.234206 8.315413 b
## 11 0 floresc 9.4 8.286737 10.662822 a
## 12 25 floresc 5.8 4.940092 6.809590 b
## 13 50 floresc 5.8 4.940092 6.809590 b
## 14 75 floresc 6.0 5.124243 7.025428 b
## 15 100 floresc 4.6 3.841499 5.508267 b
## 16 0 maça 8.6 7.538145 9.811432 a
## 17 25 maça 7.0 6.048663 8.100963 ab
## 18 50 maça 8.8 7.725078 10.024495 a
## 19 75 maça 6.2 5.308653 7.241008 b
## 20 100 maça 3.0 2.400033 3.749949 c
## 21 0 capulho 10.0 8.849596 11.299951 a
## 22 25 capulho 10.0 8.849596 11.299951 a
## 23 50 capulho 8.2 7.164742 9.384845 a
## 24 75 capulho 8.8 7.725078 10.024495 a
## 25 100 capulho 9.0 7.912157 10.237411 a# Gráfico com estimativas, IC e texto.
ggplot(data = results_tk,
mapping = aes(x = desf, y = fit)) +
facet_wrap(facets = ~fase, nrow = 1) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, cld)),
size = 3,
angle = 90,
vjust = 0,
nudge_x = -0.1) +
expand_limits(x = c(0, NA)) +
labs(x = "Desfolha", y = "Número de capulhos (2 plantas)")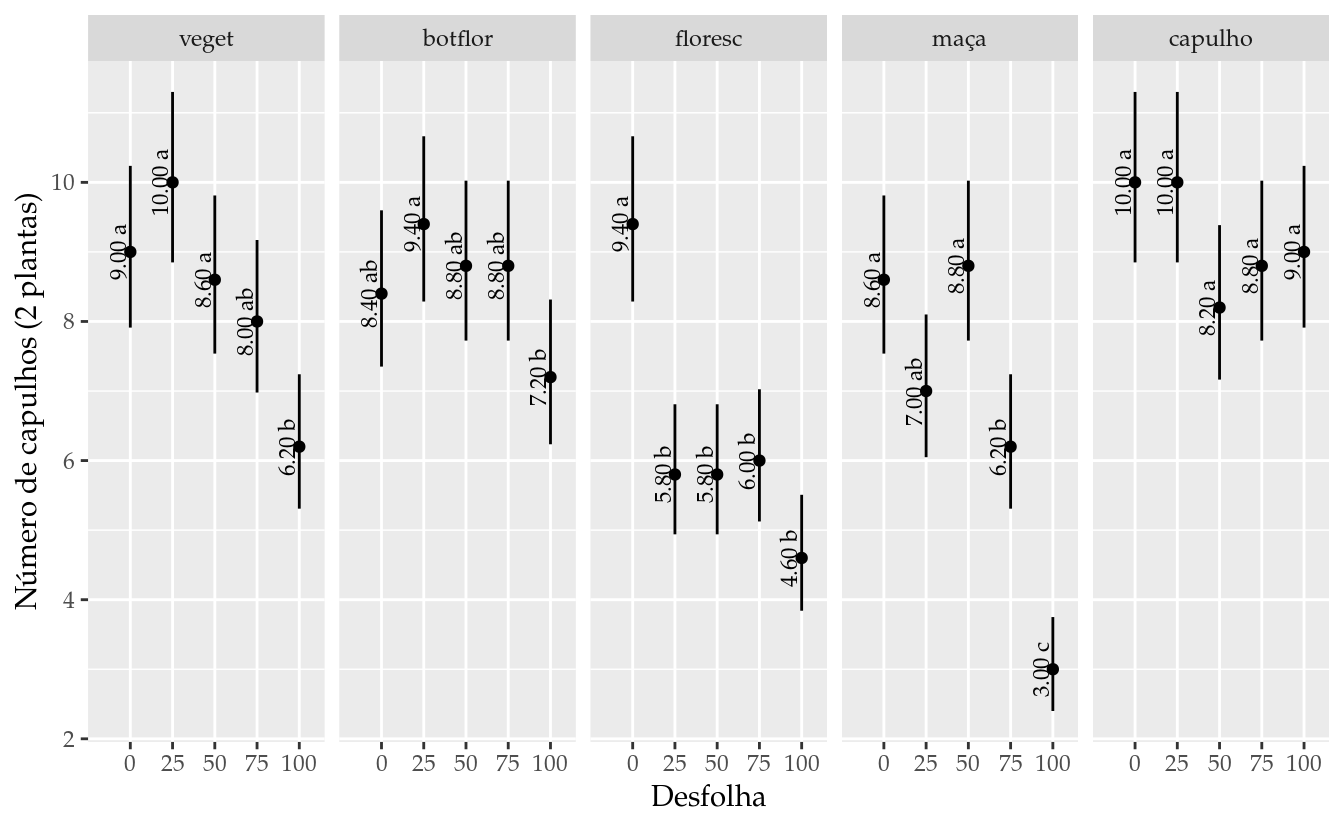
# TODO FIXME: fazer contrastes sempre com relação a desfolha 0.
contrMat(n = 1:4, type = "Dunnet") # Contrastes de Dunnet.##
## Multiple Comparisons of Means: Dunnett Contrasts
##
## 1 2 3 4
## 2 - 1 -1 1 0 0
## 3 - 1 -1 0 1 0
## 4 - 1 -1 0 0 1contrMat(n = 1:4, type = "Tukey") # Contrastes de Tukey.##
## Multiple Comparisons of Means: Tukey Contrasts
##
## 1 2 3 4
## 2 - 1 -1 1 0 0
## 3 - 1 -1 0 1 0
## 4 - 1 -1 0 0 1
## 3 - 2 0 -1 1 0
## 4 - 2 0 -1 0 1
## 4 - 3 0 0 -1 1# Obtém os contrastes com relação ao primeiro nível (desf == 0).
results_dn <- split(x = seq(group_indices(grp)),
f = group_indices(grp)) %>%
lapply(FUN = function(i) {
K <- sweep(x = L[i[-1], ], # Os demais níveis.
STATS = L[i[1], ], # O primeiro nível.
MARGIN = 2,
FUN = "-")
summary(glht(m0, linfct = K),
test = adjusted(type = "none"))
})
results_dn[[1]]##
## Simultaneous Tests for General Linear Hypotheses
##
## Fit: glm(formula = ncapu ~ fase * desf, family = quasipoisson, data = da)
##
## Linear Hypotheses:
## Estimate Std. Error z value Pr(>|z|)
## 25_veget == 0 0.10536 0.09060 1.163 0.244860
## 50_veget == 0 -0.04546 0.09403 -0.483 0.628741
## 75_veget == 0 -0.11778 0.09581 -1.229 0.218963
## 100_veget == 0 -0.37268 0.10291 -3.621 0.000293 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## (Adjusted p values reported -- none method)results_dn <-
map_df(results_dn,
function(u) {
bk <- c(-Inf, 0.1, 1, 5, Inf)/100
lb <- c("***", "**", "*", "")
u$test[c(3, 6)] %>%
as.data.frame() %>%
rownames_to_column() %>%
separate(col = 1,
into = head(names(grid), n = -1)) %>%
mutate(star = cut(pvalues, breaks = bk, labels = lb),
star = as.character(star))
})
str(results_dn)## 'data.frame': 20 obs. of 5 variables:
## $ desf : chr "25" "50" "75" "100" ...
## $ fase : chr "veget" "veget" "veget" "veget" ...
## $ coefficients: num 0.1054 -0.0455 -0.1178 -0.3727 0.1125 ...
## $ pvalues : num 0.24486 0.628741 0.218963 0.000293 0.229593 ...
## $ star : chr "" "" "" "***" ...results <- full_join(results_tk,
select(results_dn, -c(coefficients, pvalues))) %>%
mutate(desf = factor(desf, levels = levels(da$desf)),
fase = factor(fase, levels = levels(da$fase)),
star = replace_na(star, ""))## Joining, by = c("desf", "fase")str(results)## 'data.frame': 25 obs. of 7 variables:
## $ desf: Factor w/ 5 levels "0","25","50",..: 1 2 3 4 5 1 2 3 4 5 ...
## $ fase: Factor w/ 5 levels "veget","botflor",..: 1 1 1 1 1 2 2 2 2 2 ...
## $ fit : num 9 10 8.6 8 6.2 8.4 9.4 8.8 8.8 7.2 ...
## $ lwr : num 7.91 8.85 7.54 6.98 5.31 ...
## $ upr : num 10.24 11.3 9.81 9.17 7.24 ...
## $ cld : chr "a" "a" "a" "ab" ...
## $ star: chr "" "" "" "" ...# Gráfico com estimativas, IC e texto.
ggplot(data = results,
mapping = aes(x = desf, y = fit)) +
facet_wrap(facets = ~fase, nrow = 1) +
geom_point() +
geom_errorbar(mapping = aes(ymin = lwr, ymax = upr),
width = 0) +
geom_text(mapping = aes(label = sprintf("%0.2f %s", fit, star)),
size = 3,
angle = 90,
vjust = 0,
nudge_x = -0.1) +
expand_limits(x = c(0, NA)) +
labs(x = "Desfolha", y = "Número de capulhos (2 plantas)")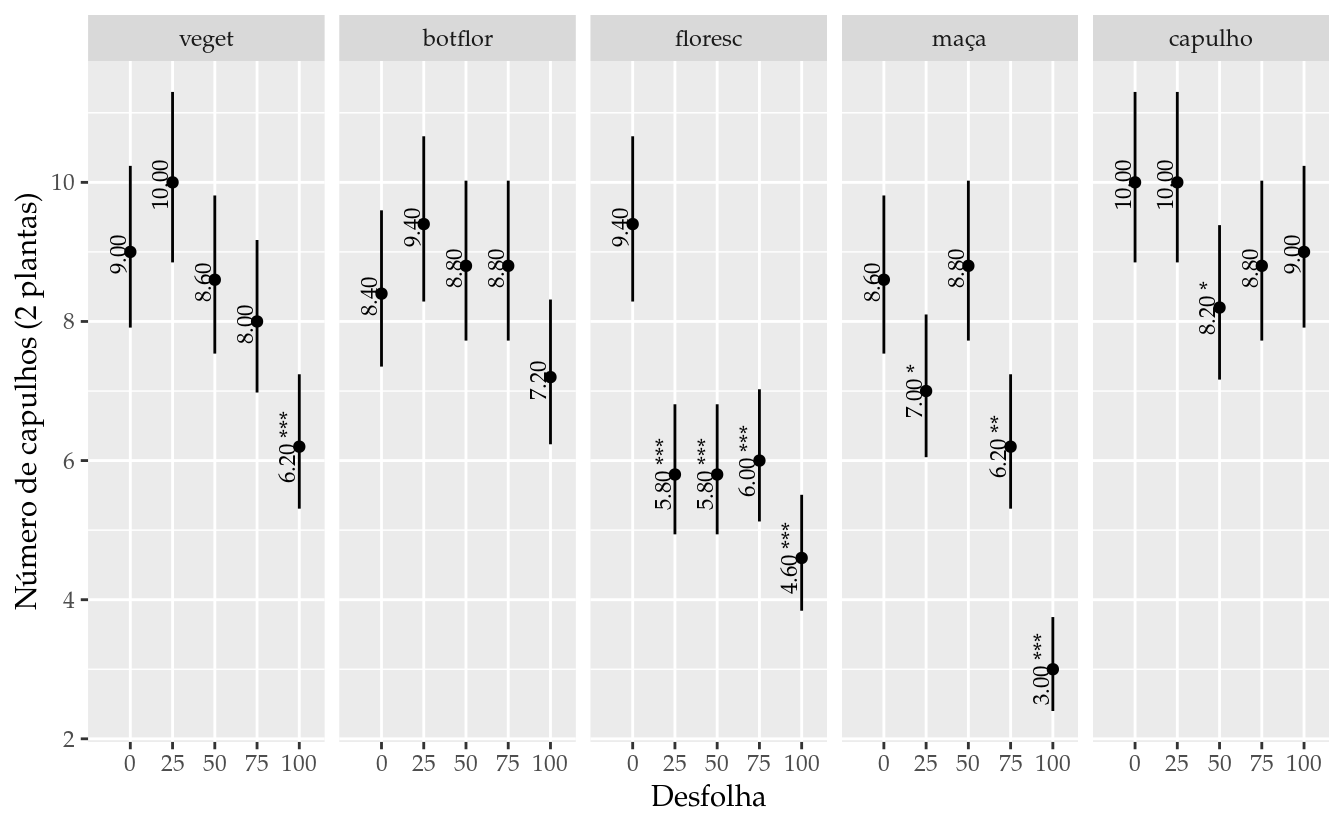
9.5 Resposta de proporção
url <- "http://leg.ufpr.br/~walmes/data/caiaue.txt"
da <- read_tsv(url, comment = "#")## Parsed with column specification:
## cols(
## periodo = col_double(),
## umidade = col_character(),
## trat = col_double(),
## rep = col_double(),
## germ = col_double(),
## pcont = col_double(),
## ivg = col_double(),
## sg = col_double(),
## sng = col_double()
## )attr(da, "spec") <- NULL
# Tabela de frequência dos pontos experimentais.
xtabs(~periodo + umidade, data = da)## umidade
## periodo 18-19 19-20 20-21 21-22 22-23
## 55 3 3 3 3 3
## 75 3 3 3 3 3
## 100 3 3 3 3 3# Converte para fator.
da <- da %>%
mutate(periodo = factor(periodo),
umidade = factor(umidade))
# Análise exploratória.
ggplot(data = da,
mapping = aes(x = umidade, y = sg/(sg + sng))) +
facet_wrap(facets = ~periodo) +
geom_jitter(width = 0.1, height = 0, pch = 1) +
stat_summary(aes(group = 1), geom = "line", fun.y = "mean")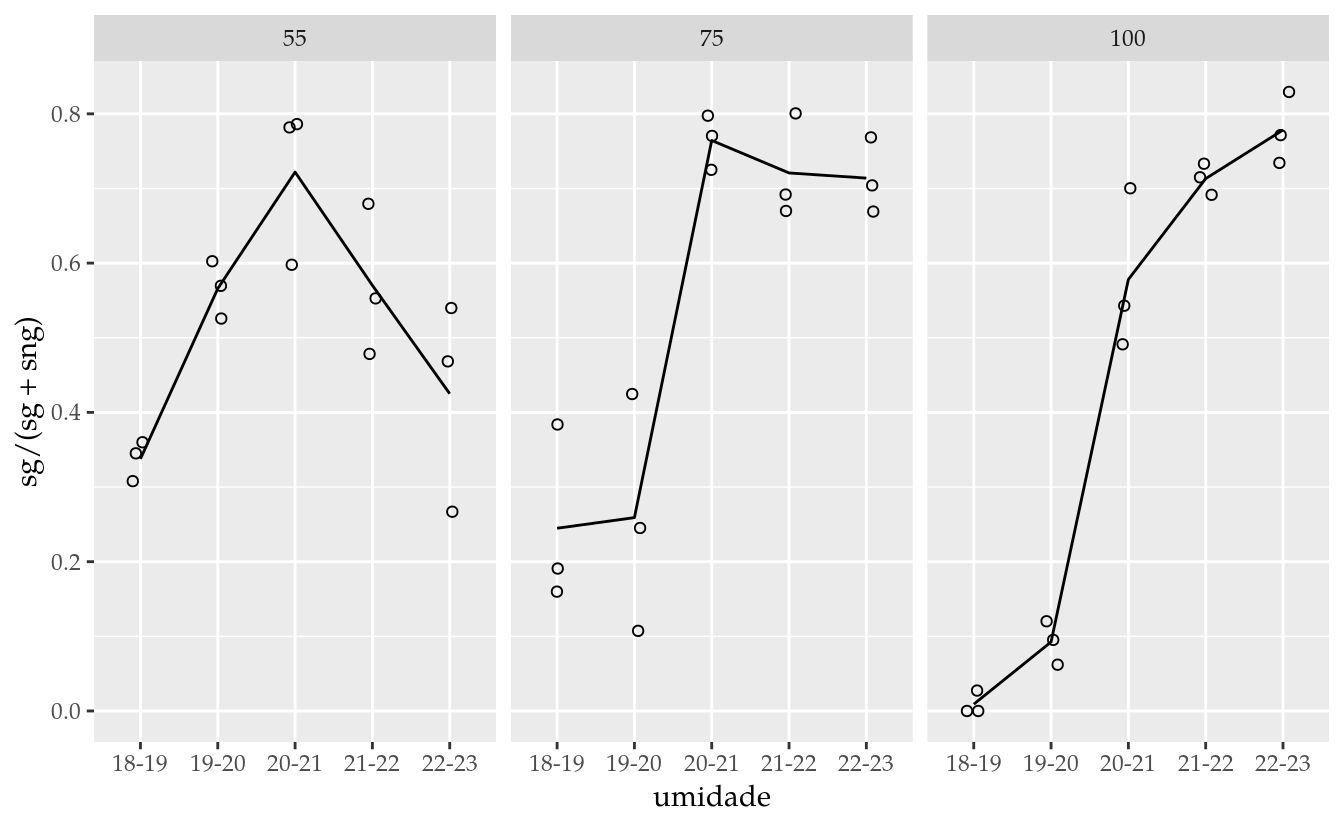
9.6 Nematóide
url <- "http://leg.ufpr.br/~walmes/data/aveia_nematoide.txt"
da <- read_tsv(url, comment = "#")## Parsed with column specification:
## cols(
## cultivar = col_character(),
## nivel = col_double(),
## mfr = col_double(),
## mfpa = col_double(),
## mspa = col_double(),
## fr = col_double(),
## nema = col_double()
## )attr(da, "spec") <- NULL
da <- da %>%
mutate(cultivar = factor(cultivar, levels = unique(cultivar)),
nivel = factor(nivel, levels = unique(nivel)))
xtabs(~cultivar + nivel, da)## nivel
## cultivar 0 0.0625 0.125 0.25 0.5 1 2 4 8 16
## Afrodite 4 4 4 4 4 4 4 4 4 4
## Torena 4 4 4 4 4 4 4 4 4 4
## Slava 4 4 4 4 4 4 4 4 4 4ggplot(data = da,
mapping = aes(x = nivel, y = mspa)) +
facet_wrap(facets = ~cultivar) +
geom_point() +
stat_summary(aes(group = 1), geom = "line", fun.y = "mean")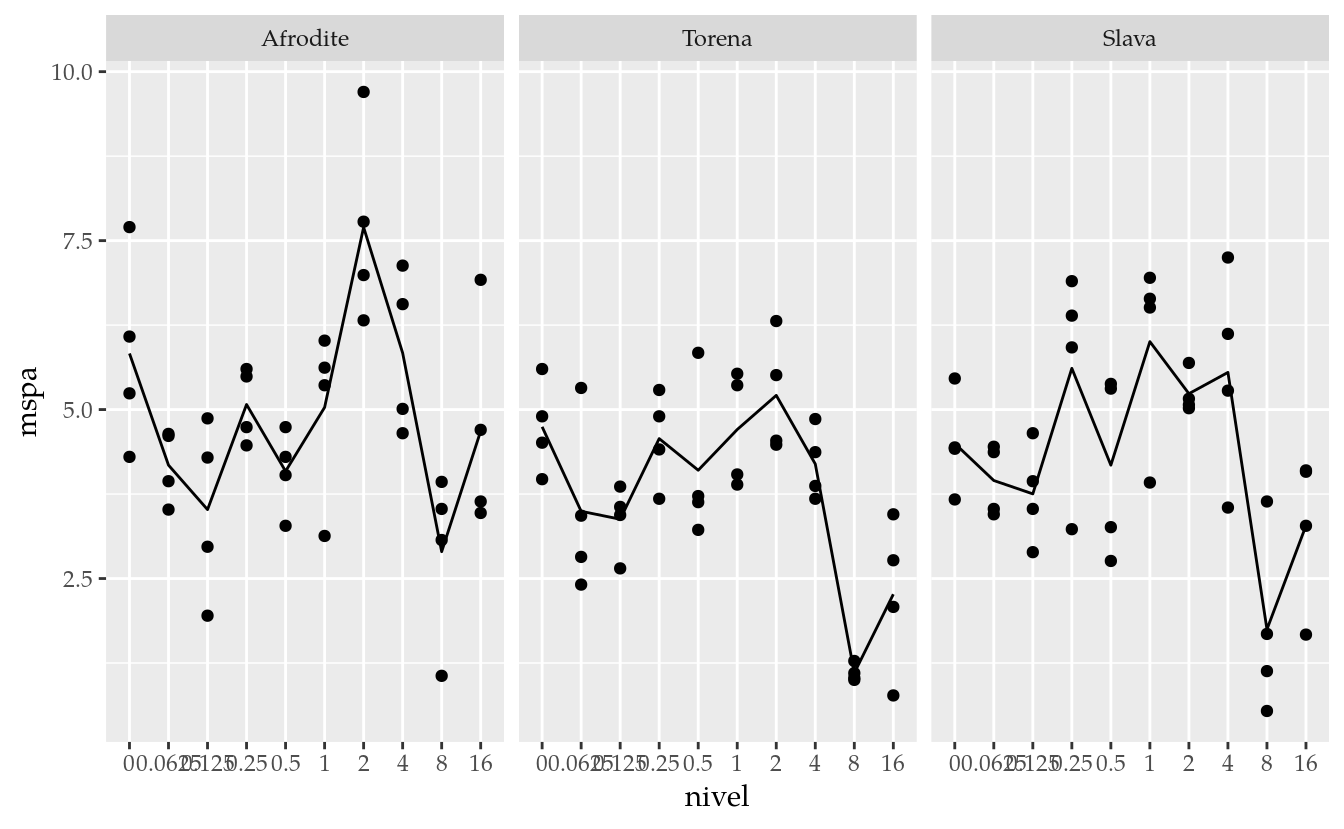
# DANGER ATTENTION: a mesma titulação é usada nas 3 cultivares em todas
# as repetições para uma mesma concentração de nematóides.9.7 StorckTb60
9.8 Soja
9.9 Com repetições dentro de parcela
file:///home/walmes/Dropbox/CursoR/GeneticaEsalq/script12.html
9.10 Considerações finais
WALMES FIXME TODO.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani