Capítulo 2 Delineamento Inteiramente Casualizado
2.1 Fundamentos
Experimentos no delineamento inteiramente casualizado são os mais simples.
Esse delineamento deve ser usado quando as i) unidades experimentais não apresentam heterogeneidade em nenhum aspecto com potencial influência nas variáveis a serem medidas e ii) o ambiente ou condições de contorno nas quais estarão as unidades experimentais não apresentará fatores que as influenciem de forma distinta. Caso alguma destas condições não for atendida, deve-se considerar outro delineamento. Vejamos alguns exemplos.
Um suposto experimento de campo para avaliação de produção de cultivares, por exemplo, só deve ser instalado no DIC se apresentar uniformidade entre unidades experimentais, ou seja, a fertilidade das parcelas é, para fins práticos, a mesma; a irrigação das parcelas é com a mesma intensidade; não existe influência de estradas, sobreamento por árvores ou vizinhança de outro experimento que possa provocar deriva de aplicações ou inóculo.
Um experimento de ganho de peso de aves em função do tipo de dieta só deve ser instalado em DIC se as aves tem aproximadamente o mesmo peso e idade FIXME.
A imagem abaixo ilustra o mapa de fertilidade do solo em uma área hipotética.
IMPROVE Suponha que um fator de 5 níveis será estudado. Se a casualização dos níveis às unidades experimentais for ao acaso, existe uma considerável chance de algum nível ficar nas zonas de maior ou menor fertilidade. Com isso, o resultado desse nível poderá ser maior ou menor por uma questão local e não por devido mérito.
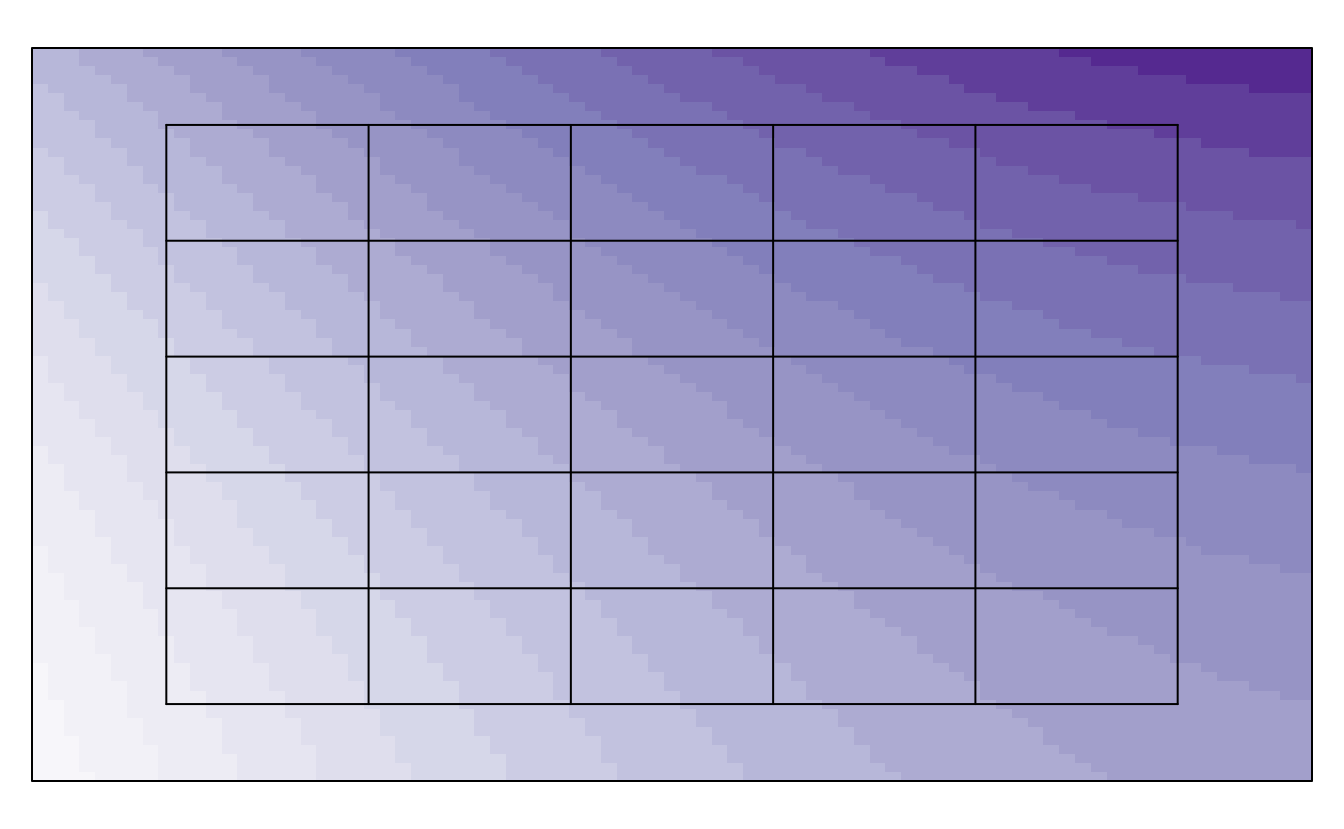
Figura 2.1: Mapa de fertilidade de uma área experimental hipotética. Os quadrados são as 25 parcelas do experimento. Tons mais escuros indicam maior fertilidade do solo. Dessa forma, existe um gradiente de fertilidade do canto inferior esquerdo para o canto superior direito.
2.2 Especificação e ajuste do modelo
\[ \begin{aligned} y_{ij}|i &\sim \text{Normal}(\mu_{ij}, \sigma^2)\\ \mu_i &= \mu + \tau_i \end{aligned} \]
em que \(y_{ij}\) corresponde ao valor observado do nível \(i\) e repetição \(j\), \(\mu_{ij}\) é a sua correspondente média (paramétrica), \(\sigma^2\) é a variância entre repetições, \(\mu\) é a média de todos os níveis sob a ausência de efeito dos níveis e \(\tau_{i}\) é o efeito do nível \(i\). IMPROVE
Se o fator em estudo for de níveis métricos, seu efeito pode ser representado por uma função, como por exemplo, um polinômio. Indicações isso por \[ \mu_i = s(x_i) = \beta_0 + \beta_1 x + \cdots + \beta_k x^k. \]
Para ilustrar como fazer a análise de um experimento no delineamento inteiramente casualizado, consideraremos o experimento descrito em ZIMMERMANN (2004) (página 50), onde estudou-se o efeito de quatro níveis de adubação nitrogenada em arroz irrigado
- Aplicação de 80 kg ha-1 no plantio;
- Aplicação de 40 kg ha-1 no plantio e 40 kg ha-1 quarenta dias após a emergência (DAE);
- Aplicação de 13,2 kg ha-1 no plantio e 66,8 kg ha-1 aos quarenta DAE;
- Aplicação de 13,2 kg ha-1 no plantio, 33,4 aos quarenta DAE e 33,4 aos sessenta DAE.
Todos os níveis correspondem à aplicação total de 80 kg ha-1 de adubação nitrogenada. Muda como o total é distribuido em frações e quando estas são aplicadas.
| Adubação | Plantio | 40 DAE | 60 DAE |
|---|---|---|---|
| 1 | 80.0 (100.0) | 0.0 (0.0) | 0.0 (0.0) |
| 2 | 40.0 (50.0) | 40.0 (50.0) | 0.0 (0.0) |
| 3 | 13.2 (16.5) | 66.8 (83.5) | 0.0 (0.0) |
| 4 | 13.2 (16.5) | 33.4 (41.8) | 33.4 (41.8) |
Estes dados estão no objeto ZimmermannTb3.5 do pacote labestData. Para termos mais agilidade na escrita do código, vamos copiar o conteúdo do objeto ZimmermannTb3.5 para um objeto de nome curto: zim. Os rótulos dos níveis de adubação serão reescritos para que fiquem mais recordáveis os apropriados para os gráficos. Decidi usar um rótulo que exibe a fração da adubação aplicada em cada época, já que o total aplicado é o mesmo.
library(labestData)## ----------------------------------------------------------------------
## labestData: Biblioteca de Dados para Aprendizado de Estatística
##
## Para colaboração, suporte ou relato de bugs, visite:
## http://gitlab.c3sl.ufpr.br/pet-estatistica/labestData,
## https://github.com/pet-estatistica/labestData
##
## labestData version 0.1-1.462 (feito em 2016-08-29) foi carregado.
## ----------------------------------------------------------------------str(ZimmermannTb3.5)## 'data.frame': 32 obs. of 3 variables:
## $ adub: Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 1 1 2 2 ...
## $ rept: int 1 2 3 4 5 6 7 8 1 2 ...
## $ prod: num 6276 6035 6086 5594 6321 ...# Nomes curtos dão mais agilidade.
zim <- ZimmermannTb3.5
# Atribui novos rótulos para os níveis.
levels(zim$adub) <- c("100/0/0", "50/50/0", "16/84/0", "16/42/42")
# Produção média.
aggregate(prod ~ adub, data = zim, FUN = mean)## adub prod
## 1 100/0/0 6125.00
## 2 50/50/0 6651.50
## 3 16/84/0 6247.00
## 4 16/42/42 6869.75Medidas descritivas da produção de grãos para cada nível de adubação são exibidas com a função tables::tabular(). Ela permite obtenção de tabelas resumo com flexibilidade. Vale a pena estudar os seus recursos.
Pela tabela, verificamos que existe diferença numérica nas médias. Caso as médias fossem numericamente iguais, estaríamos diante de um nexus vorti1 e um portal para outra dimensão iria se abrir. Brincadeiras a parte, quero enfatizar que as médias serão, com alta probabilidade, numericamente diferentes quando estivermos medindo uma variável continua, como a produção de grãos. Variáveis de contagem, por outro lado, tem uma chance maior de apresentar médias iguais, já que o suporte é o dos números inteiros.
library(tables)
# Tabela com medidas descritivas: média, desvio-padrão e número de obs.
tabular(adub + 1 ~ prod * (mean + sd + length), data = zim)| prod | |||
|---|---|---|---|
| adub | mean | sd | length |
| 100/0/0 | 6125 | 355.1 | 8 |
| 50/50/0 | 6652 | 404.3 | 8 |
| 16/84/0 | 6247 | 310.9 | 8 |
| 16/42/42 | 6870 | 373.8 | 8 |
| All | 6473 | 460.5 | 32 |
Apesar da tabela com medidas descritivas, considero o gráfico mais interessante para fornecer impressão sobre os dados.
library(lattice)
xyplot(prod ~ reorder(adub, prod),
data = zim,
type = c("p", "a"),
ylab = expression("Produção de grãos de arroz" ~ kg ~ ha^{-1}),
xlab = expression("Percentual aplicado de 80" ~
kg ~ ha^{-1} ~ "(Plantio/40 DAE/60 DAE)"))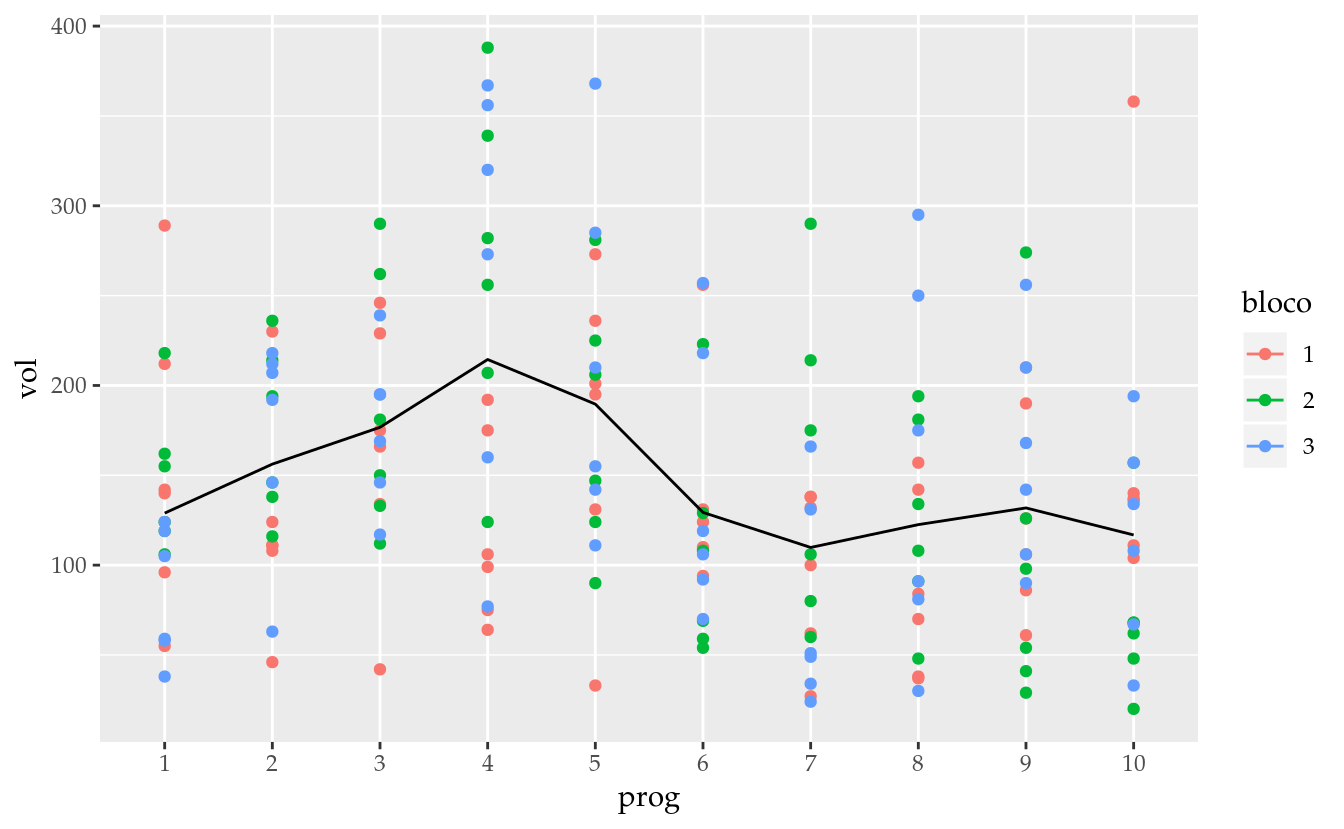
Figura 2.2: Diagrama de dispesão da produção de grãos de arroz (kg ha\(^{-1}\)) para cada nível de adubação nitrogenada. Os níveis de adubação estão ordenadaos pela média amostral das repetições. A linha contínua une as médias amostrais de produção.
A análise dos resultados do experimento começa pela especificação do modelo. Como produção é uma variável contínua, podemos considerar a distribuição normal. O fato dela ser uma variável biométrica, influenciada por inúmeros genes e fatores ambientais com contribuição infinitesimal, é o que induz a resposta ter distribuição normal. Obviamente que alguém poderá mencionar variáveis biométricas em igual condição que não apresentam normalidade, mas existem outras formas de pensar sobre isso.
Temos a premissa de trabalho que as diferentes forma de adubação irão provocar mudanças na média da produção de grãos de arroz. Não temos porque acreditar que a variabilidade dos resultados será influenciado, por isso, assumimos que a variãncia é constante. O modelo que resume isso é \[ \begin{aligned} y_{ij} &\sim \text{Normal}(\mu_i, \sigma^2)\\ \mu_i &= \mu + \tau_i, \end{aligned} \] em que \(y_{ij}\) é o valor observado na \(j\)-ésima repetição do nível \(i\) de adubação que tem média \(\mu_i\), \(\sigma^2\) é a variância das observações, de distribuição normal, ao redor da média. Na expressão debaixo, \(\mu_i\) é decomposto em dois conjuntos de parâmetros: \(\mu\) que é não indexado, representa uma quantidade invariável ao nível de adubação e \(\tau_i\) representa o efeito do nível \(i\).
Um problema surge com essa decomposição: do lado dereito tem-se, desnecessariamente, um parâmetro a mais que do lado direito. Para eliminá-lo, utilizados uma restrição paramétrica que nada mais é que uma função linear dos parâmetros com valor conhecido.
- Contraste soma zero (
contr.sum): considera-se que \(\sum_{i=1}^{I} \tau_i = 0\), isso implica em \(\tau_I = -\sum_{i=1}^{I-1} \tau_i\). - Contraste tratamento (
contr.treatment, default): considera-se que \(\tau_1 = 0\) e com isso \(\mu_1 = \mu\), e também que \(\tau_i = \mu - \mu_i, i \geq 2\). Os \(\tau\)’s são diferenças entre cada média com relação a do primeiro nível. - Contraste SAS (
contr.SAS): considera-se que \(\tau_I = 0\). Esse contraste é a imagem espelhada do anterior pois zera o último nível ao invés do primeiro. - Contraste de Helmert (
contr.helmert): contrasta cada nível contra os níveis que o precedem: \((\mu_i - \sum_{u = 1}^{i - 1} \mu_u)/i, i \geq 2\).
Os contrastes conferem particular significado para as estimativas dos parâmetros. Variáveis de níveis categóricas não ordenadas.
Os que zeram um nível são interessantes quando existe uma referência, baseline. O soma zero é usado quando … O Helmert, para mim, tem uso em experimentos fatoriais com tratamentos adicionais. De um ponto da análise em diante, é irrelevante o tipo de contraste. Ele tem importância quando-se que quer desdobrar SQ dentro da anova.
# Fator de 4 níveis.
x <- gl(4, 1)
# Nome dos contrastes.
ctr <- paste0("contr.", c("treatment", "SAS", "sum", "helmert"))
# Obtendo a matriz de contrantes para cada tipo.
L <- lapply(ctr,
FUN = function(type) {
cbind(1, contrasts(C(x, type)))
})
names(L) <- ctr
L
# Obtendo qual função da média cada parâmetro corresponde.
lapply(L, function(x) MASS::fractions(solve(x)))# Ajuste do modelo.
m0 <- lm(prod ~ adub, data = zim)
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: prod
## Df Sum Sq Mean Sq F value Pr(>F)
## adub 3 2891619 963873 7.3299 0.000895 ***
## Residuals 28 3681943 131498
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas dos efeitos.
summary(m0)##
## Call:
## lm(formula = prod ~ adub, data = zim)
##
## Residuals:
## Min 1Q Median 3Q Max
## -531.00 -303.31 -16.75 263.06 621.00
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 6125.0 128.2 47.774 < 2e-16 ***
## adub50/50/0 526.5 181.3 2.904 0.007116 **
## adub16/84/0 122.0 181.3 0.673 0.506547
## adub16/42/42 744.8 181.3 4.108 0.000315 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 362.6 on 28 degrees of freedom
## Multiple R-squared: 0.4399, Adjusted R-squared: 0.3799
## F-statistic: 7.33 on 3 and 28 DF, p-value: 0.000895Qualquer que seja o contraste usado, é possível estimar as médias \(\hat{\mu}_i\). Essas médias são chamadas médias de mínimos quadrados pois são calculadas como funções das estimativas de mínimos quadrados para diferenciar das médias amostrais.
ctr <- paste0("contr.", c("treatment", "SAS", "sum", "helmert"))
sapply(ctr,
FUN = function(type) {
m1 <- update(m0,
contrasts = list(adub = type))
K <- cbind("(Intercept)" = 1,
contrasts(C(zim$adub, type)))
K %*% coef(m1)
})## contr.treatment contr.SAS contr.sum contr.helmert
## [1,] 6125.00 6125.00 6125.00 6125.00
## [2,] 6651.50 6651.50 6651.50 6651.50
## [3,] 6247.00 6247.00 6247.00 6247.00
## [4,] 6869.75 6869.75 6869.75 6869.752.3 Análise dos pressupostos
par(mfrow = c(2, 2))
plot(m0); layout(1)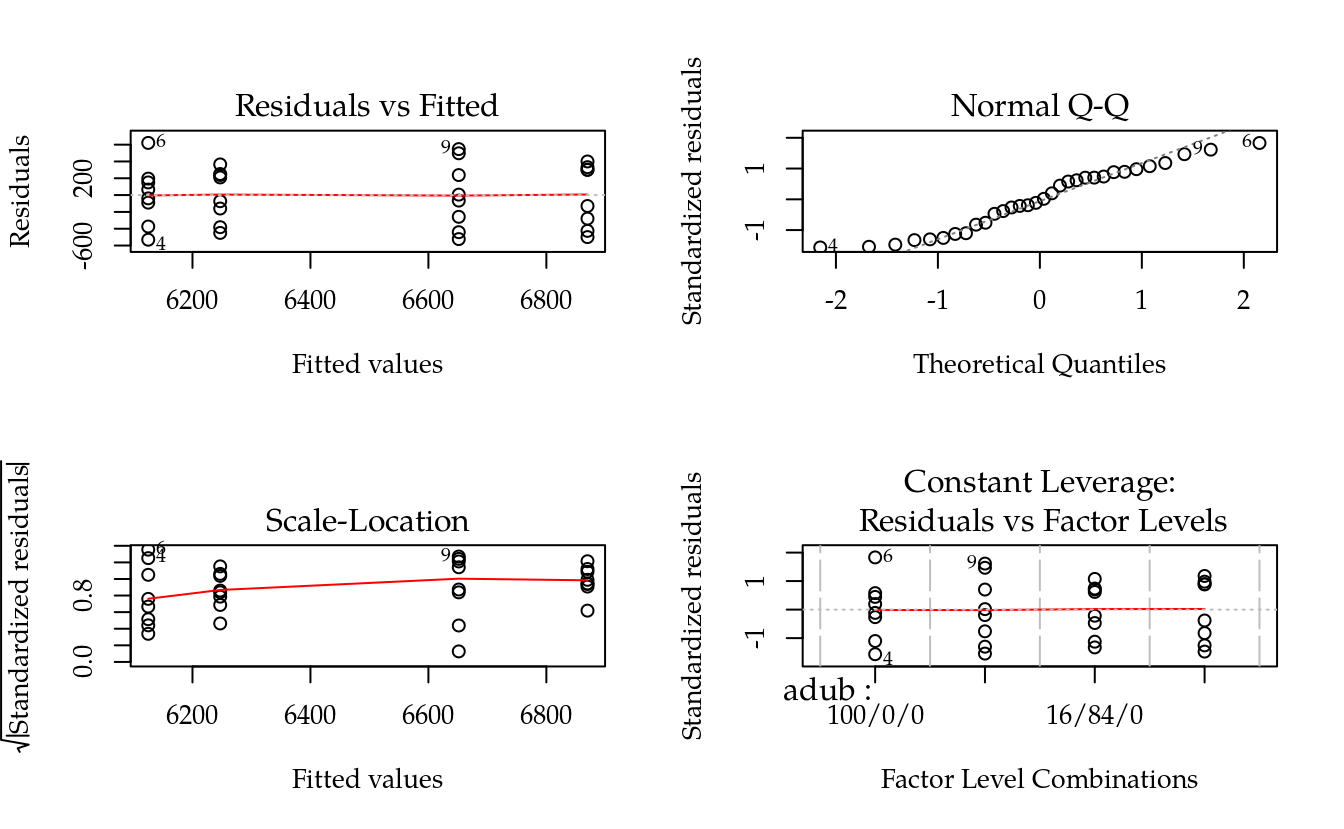
Interpretar cada gráfico.
Definir brevemente cada resíduo.
2.4 Contrastes
Como sair com os contrastes ortogonais dentro do quadro de anova e fazer contrastes fora.
2.5 Compações múltiplas
Conduzir testes baseados em DMS, SK e LS means.
2.6 Uso de regressão
Ajustar regressão.
2.7 Geração do delineamento
Fazer a casualização de um novo experimento.
2.8 Simulação do modelo
Simular do modelo
2.9 Termos de efeito fixo vs aleatório
Referências Bibliográficas
ZIMMERMANN, F. J. Estatística aplicada à pesquisa agrícola. 1st ed. Santo Antônio de Goiás, GO: Embrapa Arroz e Feijão, 2004.
Manual de Planejamento e Análise de Experimentos com R
Walmes Marques Zeviani